
Texto escrito por Marco Antônio Marques Pretti e Ana Arnt
A evolução da pandemia de COVID-19 trouxe à tona diferenças marcantes na taxa de mortalidade entre os países (WHO, 2020). É de se esperar que países mais populosos tenham um maior número absoluto de casos e de desfechos letais. Contudo, quando comparamos estes dados analisando-os pelo total de habitantes de cada país (o que chamamos de “normalizar” os dados), os países mais populosos não possuem, necessariamente, uma taxa maior de mortalidade associada à COVID-19.
Existem vários fatores socioeconômicos e políticos conhecidos relacionados a estas diferenças de mortalidade. Todavia, há fatores genéticos que também poderiam estar envolvidos a isto. E nosso grupo de pesquisa buscou compreender exatamente estes aspectos!
Sobre fatores genéticos, populações e COVID-19
Nosso grupo de pesquisa, o Laboratório de Bioinformática e Biologia Computacional (LBBC), utiliza diversas ferramentas de bioinformática para o estudo do câncer e também das moléculas de HLA. Mas, o que vem a ser essas moléculas de HLA?
As moléculas de HLA são proteínas localizadas na superfície da célula (Figura 1). Elas são responsáveis por apresentar ao sistema imune duas coisas:
- porções de proteínas da própria célula,
- Porções de proteínas de patógenos que invadem nosso corpo, como é o caso do vírus causador da COVID-19, o SARS-CoV-2.
Essas partes (ou porções) de proteínas – de nossas células ou dos vírus – são chamadas de peptídeos. Dessa forma, o sistema imune consegue realizar uma varredura no que está sendo expresso pela célula e identificar células infectadas por patógenos e que precisam ser eliminadas.
Mas há mais uma informação muito importante sobre estas moléculas!
Elas são muito diversas entre as populações. Isto é, existem milhares de “tipos” de HLA no mundo. No entanto, quando analisamos as HLAs nas pessoas de um mesmo país ou região, estas moléculas tendem a ser bem parecidas. As HLAs são proteínas e, portanto, definidas geneticamente. Cada indivíduo possui até 6 moléculas diferentes de HLA, definidas por alelos (versões dos genes) diferentes, três de origem materna e três de origem paterna (Figura 1). Estas diferenças entre as HLAs é o que possibilita às HLAs se ligarem a diferentes partes dos vírus.
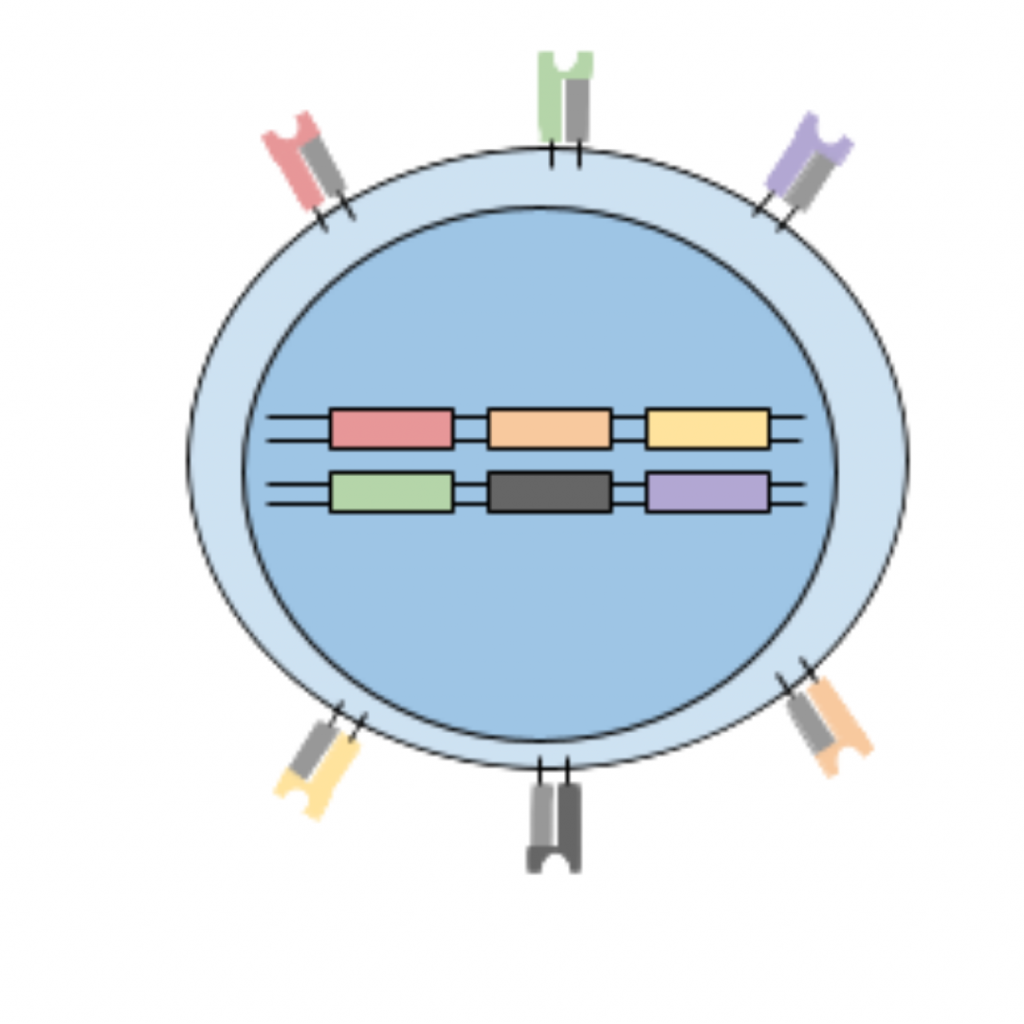
Nosso trabalho utilizou ferramentas de predição (previsão) para identificar porções do vírus da COVID-19 com a capacidade de se ligar a mais de 100 alelos de HLA majoritários em 37 países (PRETTI et al., 2020). Assim, ao considerarmos a frequência das diferentes moléculas de HLA-I entre as populações analisadas, foi possível perceber semelhanças entre países com melhor e pior desfecho frente à COVID-19. O que encontramos foi:
Alguns dos alelos de HLA estão associados a um efeito protetor enquanto outros não.
Isso nos deu indícios que a participação das moléculas de HLA é importante na COVID-19!
Para analisar e prever que partes das proteínas dos vírus – os peptídeos, lembra? – que se ligam às moléculas de HLA usamos ferramentas de bioinformática. Isso é importante no desenvolvimento de algumas vacinas e também no estudo da biologia da doença. Imagine que se fosse possível identificar um conjunto de 5 peptídeos virais apresentados por HLAs de todos os povos teríamos em mãos uma vacina universal! Infelizmente não é tão simples encontrar 5 peptídeos apresentados por diferentes HLAs ao mesmo tempo… Por que isso acontece?
Sobre a Cobertura Antigênica
Considere as letras do “LBBC” (a sigla do nosso laboratório). Agora vamos fazer um jogo de suposições. Imagine que 15% dos brasileiros têm no seu sobrenome a letra “L” (Ladeira ou Silva, por exemplo). Em uma segunda etapa, vamos analisar quantos brasileiros têm sobrenome com a letra “L”, mais a quantidade de brasileiros que possuem a letra “B” em seus sobrenomes e essa proporção subirá para 25%. Por fim, adicionando ainda a letra “C”, e chegamos na proporção total de 30%. Logo, podemos dizer que as letras “L”, “B” ou “C” cobrem 30% dos sobrenomes brasileiros (e os sobrenomes podem ter 1, 2 ou 3 dessas letras em diferentes combinações).
Pois bem, é razoável imaginar que nas populações chinesa, estadunidense ou francesa, possa existir estas 3 letras nos sobrenomes das pessoas. Todavia, a proporção de pessoas com essas letras em seus sobrenomes não será a mesma que na população brasileira, pois os sobrenomes tendem a ser característicos de um povo.
Se pensarmos nestas letras como os peptídeos que falávamos antes fica mais fácil de compreender como é muito difícil encontrar 5 letras (ou 5 peptídeos) que contenham 100% dos sobrenomes de todo o mundo. Cada população tende a apresentar uma gama diferente de peptídeos, pois seus HLAs são diferentes entre si. Contudo, uma mesma população apresentará peptídeos (letras) parecidas entre si. Isso se chama cobertura antigênica, o conjunto de peptídeos que um indivíduo ou população é capaz de apresentar pelo HLA.
O que nossa pesquisa fez?
O que fizemos, em seguida, foi calcular a cobertura antigênica de cada país, a fim de investigar se existe alguma associação entre essa cobertura antigênica e a mortalidade associada à COVID-19. Pera! Que relação seria essa? Associar cobertura antigênica e mortalidade? Isso mesmo. Uma de nossas hipóteses foi de que populações que mostram mais peptídeos virais ao sistema imune (através da HLA) pudessem estar mais protegidos. Ou seja, se um país tem, em média, uma maior cobertura antigênica a mortalidade poderia ser menor. Contudo, não observamos nenhuma associação da cobertura antigênica do vírus como um todo e dados de mortalidade..
Apesar disso, não nos desanimamos! Continuamos investigando os dados gerados e conseguimos observar outras associações ainda mais interessantes. Entretanto, antes disso, é importante dizer que o vírus da COVID-19 não é formado de uma só “peça” ou de uma única proteína. Ele possui algumas proteínas. Algumas destas são responsáveis por manter a estrutura do vírus, outras por facilitar a invasão de células, dentre outras. Duas delas são particularmente importantes: 1) a proteína Nucleocapsídeo, responsável por revestir e proteger o genoma do vírus; e 2) a tão famosa proteína Spike que forma espículas na superfície do vírus.
Mas, e depois disso?
Resolvemos então analisar a cobertura antigênica dos países não mais com o vírus inteiro, mas com cada proteína viral. Assim, observamos que a cobertura antigênica para a proteína viral Nucleocapsídeo possui uma correlação positiva com o número de mortes. Em outras palavras, quanto mais peptídeos virais derivados dessa proteína são apresentados ao sistema imune, maiores as chances de morte (em se tratando de população, nunca individualmente). Por outro lado, a cobertura da proteína viral Spike possui uma correlação negativa com o número de mortes. O que significa… que quanto maior a cobertura antigênica para essa proteína, menores as chances de morte!
Ufa! Se você chegou até aqui, parabéns! Aprendemos bastante coisa, vamos resumir? O estudo sugere que uma maior cobertura para peptídeos derivados da proteína Spike, ao invés da proteína Nucleocapsídeo, pode ter efeitos benéficos. Ou seja, ter uma maior cobertura antigênica para Spike se vincula a um menor número de mortes associadas à COVID-19.
Não é possível mudar nossas moléculas de HLA (nem queremos isso!), pois elas são uma herança genética. Por outro lado, poderíamos, um dia, determinar grupos que possuem HLAs de risco e que deveriam vacinar-se primeiro.
Por fim
De modo geral, o trabalho associou coberturas antigênicas do SARS-CoV-2 com dados de mortalidade de cada país. Nós observamos correlações entre número de mortes relacionadas à COVID-19 e coberturas antigênicas para proteínas do vírus até então não descritas no nível populacional. Além disso, selecionamos diversos peptídeos derivados de porções virais associadas a uma resposta predita como protetora para a COVID-19 e potencialmente apresentados por HLAs com maior frequência na população mundial.
Em suma, onde esta pesquisa pode nos levar? A compreensão destas relações entre peptídeos e coberturas antigênicas nos possibilitam perceber melhor detalhes moleculares da doença, tanto sobre gravidade da doença e possibilidades de proteção. Estes resultados também podem nos trazer condições para o desenvolvimento de vacinas de peptídeos seguras, mais eficientes e que abrangem uma maior parcela da população mundial!
Referências
PRETTI, MAM et al (2020) Class I HLA Allele Predicted Restricted Antigenic Coverages for Spike and Nucleocapsid Proteins Are Associated With Deaths Related to COVID-19. Frontiers in immunology, v11, p565730.
THE NEW YORK TIMES (2021) Bad News Wrapped in Protein: Inside the Coronavirus Genome – The New York Times.
WHO, COVID-19 situation reports.
O Autor
Marco é graduado em Farmácia pela Universidade Federal do Rio de Janeiro, mestre em oncologia pelo Instituto Nacional de Câncer e aluno de doutorado pelo mesmo Instituto. Além da oncologia, possui interesse em imunologia, bioinformática e ciências da vida. Durante o confinamento teve que substituir a prática de voleibol por atividades físicas indoor.
Ana Arnt, Bióloga, Mestre e Doutora em Educação. Professora do Departamento de Genética, Evolução, Microbiologia e Imunologia, do Instituto de Biologia (DGEMI/IB) da UNICAMP e do Programa de Pós-Graduação em Ensino de Ciências e Matemática (PECIM). Pesquisa e da aula sobre História, Filosofia e Educação em Ciências, e é uma voraz interessada em cultura, poesia, fotografia, música, ficção científica e… ciência! 😉

Leave a Reply