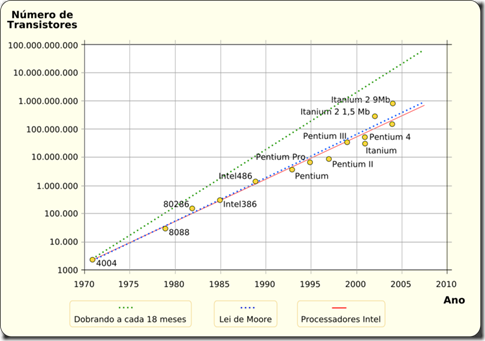
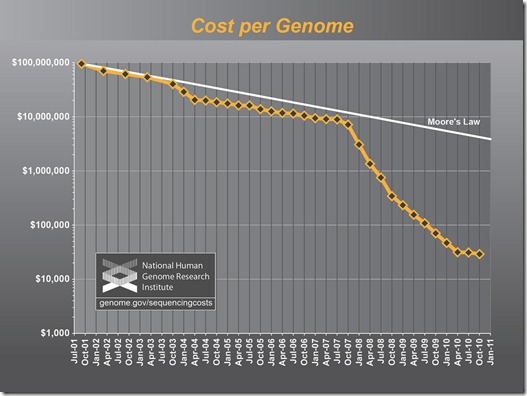
Comecei a falar de bioinformática (bioinfo) no post passado, e continuarei falando dela aqui porque não só é uma ótima dica de carreira científica como é uma necessidade científica e social estimular essa carreira pela importância que ela tem e terá no futuro para ampliar nosso conhecimento.
Porque há tantos projetos?
Quando começamos na carreira científica geralmente é assumindo um projeto, de iniciação cinetífica, mestrado ou doutorado direto, e muitas pessoas podem parar por aí por não encontrarem um orientador com projetos que o interessem no momento. Em bioinfo isso não acontece pois os projetos surgem aos borbotões e nas mais diversas áreas, afinal a quantidade de dados que surge é imensa e sempre dá pra tirar algo novo de um banco de dados que já existe, onde a informação está lá, esperando para ser minerada, e novos dados estão sempre surgindo em velocidade cada vez maior, com os novos sequenciadores, por exemplo. Assim, projetos nunca faltarão, e as bolsas junto com eles.
Porque há tão pouca gente?
Esse é o maior gargalo da área: achar bioinformatas. Isso se explica por dois fatos principais: 1- biólogos não gostam de exatas e nem de programação; 2- computeiros até gostam de biologia, mas ganham muito mais em outras áreas como recem-fromados.
Assim, quem cobre esta área são na maioria físicos e matemáticos. Mas há sim computeiros e biólogos, claro, e estes se destacam bem se aprendem o outro lado da moeda bio/info.
O que preciso saber para ser bioinformata?
Hoje em dia, pelo desespero das instituições por bioinformatas, você não precisa de nada, só gostar de pelo menos duas dessas áreas e não chegar a odiar nenhuma delas: biologia, programação, estatística e matemática. Você terá um ano ou dois para aprender o que lhe faltar nessa equação tocando um projeto de mestrado. Mas as áreas de interesse são estas que eu mencionei. Claro que quanto mais preparado você estiver mais chances terá, por isso segue aqui a dica de onde encontrar cursos de especialização de interesse com nosso parceiro Educaedu Brasil.
Uma área nova e promissora
A bioinfo é nova no Brasil, estamos na segunda geração de bioinformatas se considerarmos que o projeto genoma da Xylela foi o que introduziu o Brasil na biologia molecular contemporânea criando a primeira geração de bioinformatas (direta ou indiretamente), tais como Emmanuel Dias-Neto, Helena Brentani, Sandro Souza, Anamaria Camargo e Paulo Oliveira. Esses são só os que eu conheço, mas olhar o lattes deles já pode dar uma idéia das possibilidades da carreira.
E sobram vagas de alto nível para pesquisadores nesta área. O Hospital AC Camargo e o INCOR, ambos hospitais fortes em pesquisa que perderam recentemente seus bioinformatas, têm dificuldade de encontrar alguém para esta posição.
Então meu filho, sente aí e aprenda a programar.