Muita coisa depende do DNA. Ele guarda informação para fazer proteínas, e isso ele faz nossa vida inteira. E além de ter que durar muito ele ainda se estressa demais, porque cada vez que a célula vai se duplicar, o DNA tem que separar as suas fitas e fazer uma cópia de cada. Quando vai mandar a mensagem pra fazer proteínas, ela se abre para copiar o pedaço que interessa. Esse abre e fecha vai danificando o pobre do DNA. Além disso ainda tem radicais livres e radiações como a UV que detonam ainda mais a pobre da molécula.
Se não tivesse como arrumar, o DNA se desfaria rapidinho. E quando isso acontece os resultados podem ser dois: câncer ou envelhecimento precoce.
Mas calma, temos os ganhadores do Nobel de química de 2015 para nos ajudar! Eles descobriram mecanismos de reparo que as células têm para corrigir os erros.
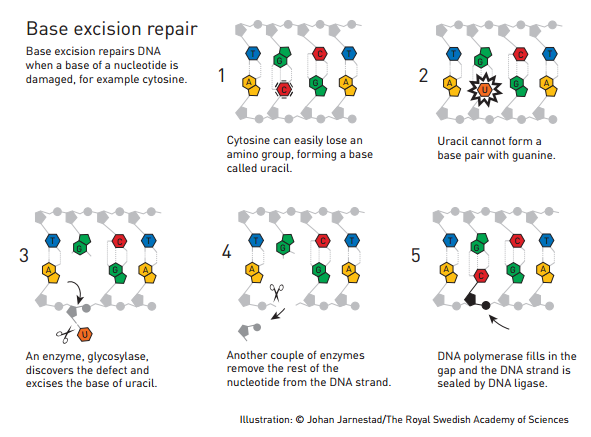
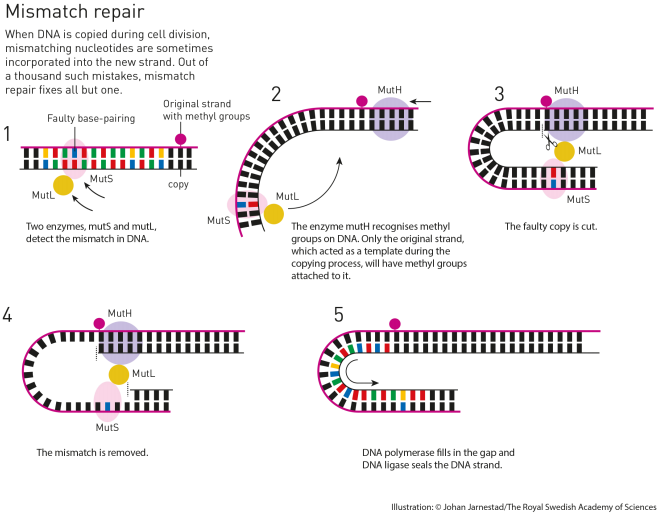
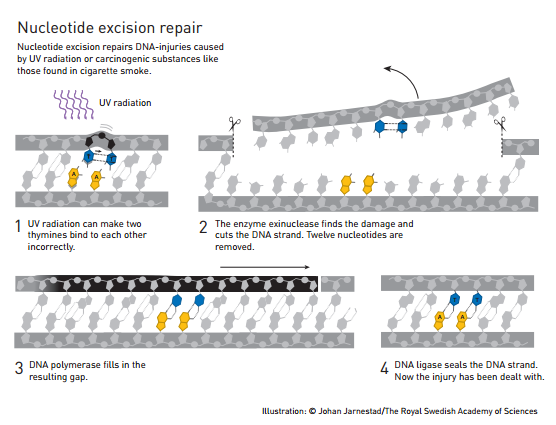
E cada um descobriu um tipo de reparo, já que pra cada tipo de dano tem um tipo de reparo. É como um carro que se levou uma batida você leva no funileiro, se for motor, no mecânico, e se for elétrico, só resolve uma autoelétrica. No caso do DNA os danos são reparo por excisão de bases (base excision repair), reparo por mau pareamento (mismatch repair), e reparo por excisão de nucleotídeos (nucleotide excision repair).
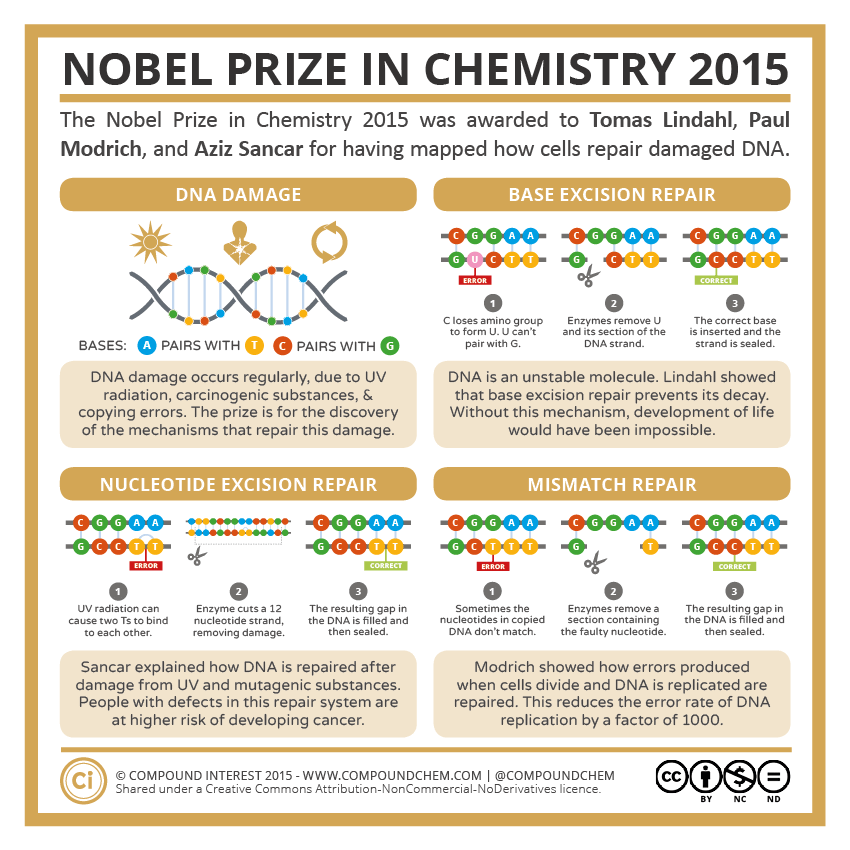
O infográfico que eu fiz alí acima mostra quem descobriu qual tipo de reparo. Claro que tem muito mais gente pesquisando sobre isso. Aliás, qualquer coisa sobre câncer pode apostar que tem muita gente pesquisando, porque é um assunto importante, complexo e muito interessante.
A própria organização do prêmio Nobel fez esquemas para mostrar como funciona cada tipo de reparo, o que eu achei bem legal da parte deles.
E aqui um infográfico de um site muito bacana, o Compound Interest, que só faz infográficos de química. Muito bons e nada chatos, mas em inglês.
Publicado originalmente em inglês no blog do Mind the Graph
Saiba mais:
http://www.compoundchem.com/nobel2015/
Ferramente de infografia que usei: Mind the Graph