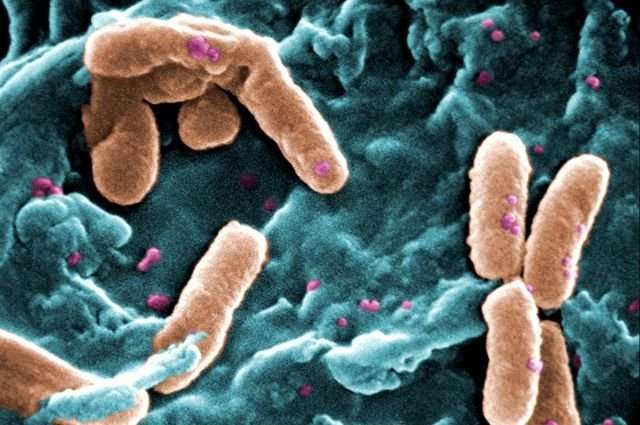
Causadora de pneumonia e infecções em implantes ósseos, a P. aeruginosa (acima) é uma das bactérias mais difíceis de combater com antibióticos. Duas pesquisas quase concomitantes nos explicam dois mecanismos de defesa desse micro-organismo.
Causadora de pneumonias problemáticas e colonizadora de implantes que substituem ossos, a bactéria conhecida como Pseudomonas aeruginosa é o terror dos ambientes hospitalares. Num relatório do Centro de Prevenção e Controle de Doenças dos EUA (a Vigilância Sanitária de lá), esse micro-organismo foi listado entre as “bactérias de pesadelo”. Segundo o documento, testes de laboratório descobriram que a P. aeruginosa teve “uma resistência incomum em mais de 200 casos em 2017”. Dado o potencial de causar epidemias graves de pneumonia, a Pseudomonas vem sendo estudada com afinco em diversos laboratórios do mundo.
Na Universidade de Notre-Dame (em Indiana, EUA), pesquisadores coordenados por Shahriar Mobashery investigaram como uma enzima ajuda a bactéria a reagir quando a dose de antibiótico não é o bastante para matá-la. Ao mesmo tempo e de forma independente, uma equipe internacional liderada por Gerard Wong, da Universidade da Califórnia em Los Angeles (UCLA) descobriu na P. aeruginosa uma espécie de “memória” que lhe permite passar informações sensoriais de uma geração para outra. Desse grupo também participaram cientistas das Universidades da Pensilvânia (EUA), Oxford (Reino Unido) e Tianjin (China).
O foco de Mobashery — que pesquisa resistência a antibiótico há 30 anos — foi uma enzima da P. aeruginosa chamada transglicosilase lítica Slt. O nome é complicado mas a função da enzima é simples: é ela que tenta arrumar a membrana celular quando a bactéria é atacada. “É uma estratégia de sobrevivência”, explica Mobashery em comunicado ao Phys.org. “A membrana celular é a estrutura que embala a bactéria inteira e sua saúde é crítica para a sobrevivência da bactéria. Se você tem uma droga que danifica a membrana, a bactéria não consegue reagir e morre.”
Medicamentos assim já existem: são os antibióticos beta-lactâmicos, como a penicilina. Usada (muitas vezes indiscriminadamente) desde os anos 1940, a penicilina e sua turma foi exposta tantas vezes à P. aeruginosa que ela desenvolveu um mecanismo de resistência.
Alfaiatezinho valente
Tudo é uma questão de manter as coisas costuradas. Acontece que a membrana da P. aeruginosa é formada por longas unidades adjacentes que são interligadas de modo parecido com uma colcha de retalhos. Na presença de antibióticos, os fios que mantém essa colcha íntegra não se formam e a membrana começa a se desfazer. A Slt age como um costureiro de emergência, cortando os retalhos longos em peças menores e costurando-os novamente.

Para estudar esse mecanismo detalhadamente, Mobashery et. al. sintetizaram pedaços da membrana celular da P. aeruginosa e aplicaram Slt para ver como a enzima a degrada e a reconstrói. A análise estrutural da enzima e da membrana foi feita através de cristalografia de raios-X por colaboradores no Consejo Superior de Investigaciones Científicas da Espanha. Publicado na Proceedings of the National Academy of Sciences (PNAS) em abril, o estudo abre mais uma porta para combater a resistência aos antibióticos da P. aeruginosa.
Cola aqui, cara
Só que essa não é a única linha de defesa da Pseudomonas. Além de reforçar suas costuras, ela costuma grudar nas superfícies, formando biofilmes que geram infecções difíceis de combater. Embora normalmente ataquem as vias respiratórias, os biofilmes dessa bactéria também podem se formar em próteses que substituem ossos. Quando isso acontece, a infecção pode ser tão grave que obriga a realização de uma nova cirurgia para trocar a prótese.
“O primeiro passo para a formação de um biofilme é que a bactéria deve sondar a superfície e desenvolver um meio de se fixar”, esclarece Calvin Lee, um dos co-autores da pesquisa da UCLA. “Pela primeira vez, fomos capazes de monitorar o comportamento de linhagens inteiras de células individuais e descobrimos que os descendentes podiam lembrar dos sinais sensoriais de superfície de seus ancestrais.”
Embora o conceito de fixação que gera os biofilmes seja conhecido desde dos anos 1930, só agora seu funcionamento está sendo investigado e explicado. Para compreender o fenômeno de sensoreamento bacteriano, Wong et. al. usaram um método de rastreamento multigeracional. Para analisar os dados colhidos assim, foi necessário usar uma técnica de processamento que é mais comum no estudo da altura de uma nota musical.
Essa abordagem incomum foi possível graças à descoberta de dois padrões rítmicos interligados: a expressão cíclica da AMP, uma molécula sinalizadora dentro da P. aeruginosa, e o nível de atividade dos pili de tipo IV, apêndices semelhantes a pelos no exterior das bactérias. Poucas horas depois do surgimento da AMP, os pelinhos da Pseudomonas começam a parar, o que facilita seu acúmulo e fixação sobre uma superfície.
O professor Wong explica: “As bactérias sentem e lembram-se desse padrão rítmico, que é decisivo para a supressão da mobilidade. Uma vez estacionárias, elas acabam por se fixar na superfície de modo irreversível e formam um biofilme.” Os resultados dessa pesquisa foram apresentados em paper que também foi publicado em abril na PNAS.
Agora que sabemos como tornam-se resistentes a antibióticos e formam biofilmes, podemos buscar maneiras mais eficazes de combater a P. aeruginosa antes que ela vire uma fonte de pesadelos como infecções mais graves e mais frequentes. No entanto, novas pesquisas ainda são necessárias para detalhar como bloquear a costura de sua membrana ou a colagem de uma colônia. Explicar essa espécie de memória sensorial num organismo que nem tem sistema nervoso pode nos levar às origens evolutivas da maneira como animais e plantas sentem o mundo à sua volta e recordam suas sensações.
Referências
 Mijoon Lee et. al. Exolytic and endolytic turnover of peptidoglycan by lytic transglycosylase Slt of Pseudomonas aeruginosa [Reversões exolítica e endolítica do peptidoglicano pela transglicosilase lítica Slt da Pseudomonas aeruginosa]. Proceedings of the National Academy of Sciences April 24, 2018. 115 (17) 4393-4398; published ahead of print April 9, 2018. www.pnas.org/cgi/doi/10.1073/pnas.1801298115
Mijoon Lee et. al. Exolytic and endolytic turnover of peptidoglycan by lytic transglycosylase Slt of Pseudomonas aeruginosa [Reversões exolítica e endolítica do peptidoglicano pela transglicosilase lítica Slt da Pseudomonas aeruginosa]. Proceedings of the National Academy of Sciences April 24, 2018. 115 (17) 4393-4398; published ahead of print April 9, 2018. www.pnas.org/cgi/doi/10.1073/pnas.1801298115
Calvin K. Lee et al. Multigenerational memory and adaptive adhesion in early bacterial biofilm communities [Memória multigeracional e adesão adaptativa nos primórdios de biofilmes de comunidades bacterianas]. PNAS April 24, 2018. 115 (17) 4471-4476; published ahead of print March 20, 2018. DOI:10.1073/pnas.1720071115


Boletim de Notícias: Homem pode ter vindo à América antes do degelo de Bering | Direto da Ciência
[…] A bactéria que cola, costura e se lembra do que faz Renato Pincelli, Hypercubic […]