Nova espécie de vida foi descoberta fora da Terra
Descobriram uma espécie nova de vida fora da Terra!

Desculpa desapontar, não vamos falar de ETs, mas de uma nova espécie bacteriana descoberta na Estação Espacial Internacional (ISS). Por que esta é uma descoberta fantástica? Quais seriam possíveis aplicações dessa descoberta? E mais… você deve imaginar como é para um cientista descrever uma nova espécie de um animal ou de uma planta que possuem várias características visíveis… Mas você já parou para pensar como é descrita uma espécie nova de bactéria? Tudo isso hoje, aqui, neste post, no Meio de Cultura.
Faz parte da vigilância da ISS, com o objetivo de manter as condições sanitárias e a saúde dos astronautas, que seja realizada a busca por microrganismos em diferentes áreas da estação espacia.

De forma muito resumida, os astronautas passam paninhos de poliéster estéreis numa área de 1m2 para coletar amostras e partículas. Esses paninhos são armazenados e enviados de volta à Terra. O material coletado é desassociado do paninho com solução salina e, então, plaqueado em meio de cultura R2A e incubado por 7 dias a 25 oC. As colônias crescidas são isoladas e armazenadas para futuras análises.

Essas análises envolveram diversos tipos de testes:
- Observação da morfologia da colônia (forma, tamanho, cor, produção de pigmento, etc.)
- Observação morfológica da bactéria ao microscópio (forma, agrupamento e coloração de Gram)
- Teste de motilidade (observar ao microscópio, se as bactérias apresentam movimento)
- Testes fisiológicos e bioquímicos, como crescimento em diferentes temperaturas de incubação, meios com diferentes pH e fontes de substratos (açucares); capacidade de produção de diferentes enzimas (catalase, oxidase); produção ou degradação de diferentes substâncias; e
- Testes genéticos (sequenciamento e análise filogenética para estabelecer relação de proximidade com espécies já conhecidas)
É muita coisa, né?
Nesse artigo, foram identificadas 4 amostras da família Methylobacteriaceae. Essas bactérias são conhecidas por estarem associadas a plantas, tendo papel importante na promoção do crescimento vegetal.
A primeira amostra foi isolada de um filtro HEPA – que possui um alto poder de filtração e é utilizado para filtrar o ar da ISS. Ele havia retornado à Terra em maio de 2011. Essa bactéria foi identificada como uma pertencente à espécie Methylorubrum rhodesianum.

Mas ela não é o foco desse post.
A grande surpresa foram as outras 3 amostras que foram coletadas em diferentes pontos da ISS entre os anos de 2015 e 2016.
Os locais foram: 1) um painel de um laboratório onde são realizados estudos de microgravidade; 2) no painel da cúpula da ISS, que é utilizada para observar as operações do lado de fora da ISS; e 3) a superfície da mesa de jantar da tripulação (mas que também usada para realizar alguns experimentos). Essas três amostras foram chamadas, respectivamente: IF7SW-B2, IIF1SW-B5 e IIF4SW-B5.
A equipe (que incluía pesquisadores estadunidenses e indianos em colaboração com a NASA) ao observar os resultados daqueles diversos experimentos fisiológicos, bioquímicos e genéticos concluíram que as três amostras eram de uma mesma espécie do gênero era Methylobacterium. As análises filogenéticas estabeleceram que essas amostras eram muito relacionadas à espécie M. indicum, mas que, apesar disso, havia diferenças significativas entre as amostras da ISS e as outras espécies já conhecidas do gênero.
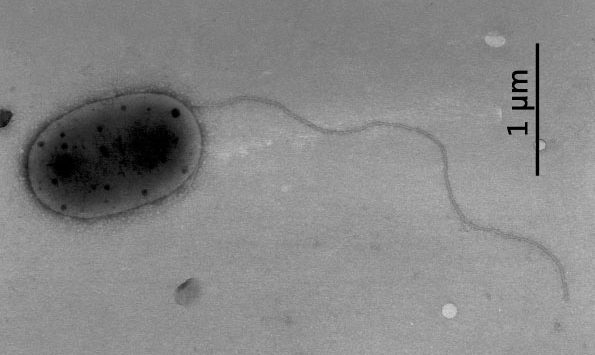
E, assim, Bijlanii e colaboradores (os autores do artigo), concluíram que as amostras da ISS eram diferentes o bastante para serem classificadas como uma nova espécie. Assim, estão propondo uma nova espécie bacteriana: Methylobacterium ajmalii. Esse nome foi escolhido em homenagem ao cientista indiano Seyed Ajmal Khan, um importante pesquisador da biodiversidade.

A descoberta, por si só, já é fantástica, pois demos mais um pequeno passo para conhecermos a nossa biodiversidade microbiana que é imensa. Além, disso, trata-se de um microrganismo capaz de crescer em condições estressantes (a ISS). Os pesquisadores acreditam que essas bactérias chegaram ali, provavelmente, devido ao início do cultivo de plantas a bordo da estação espacial – e, como falamos antes, esses microrganismos possuem há uma relação de simbiose com plantas.
O potencial biotecnológico dessa descoberta também é significativo, uso dessas bactérias na Terra quanto para missões espaciais (tanto na ISS, como em outros planetas… Isso porque as bactérias do Methylobacterium são microrganismos ubíquos (são encontrados no ar, no solo, na água, em sedimentos, e podem estar como células livres ou associados a tecidos de plantas). Além disso, estão envolvidas na fixação de nitrogênio, na solubilização de fosfato, na tolerância a estresse abiótico, na promoção do crescimento de plantas, e possuem atividade de biocontrole de patógenos vegetais.

A análise genética dessas amostras mostrou a presença de genes que permitem à bactéria ajudar no crescimento vegetal (produção de vitaminas e captação de ferro livre no solo), na proteção contra patógenos e a suportarem estresse osmótico (as plantas conseguem crescer em locais com concentrações elevadas de sais). Só alguns exemplos de genes que foram encontrados na nova espécie:
- Isopentenil-tranferase de tRNA (miaA): reportado como essencial para produção e secreção de citocinas (como a zeatina), além disso é responsável pela isopentenilação de alguns tRNAs.
- Genes para múltiplos componentes da via de síntese de vitaminas (cobalamina, biotina, tiamina, riboflavina), que indicam potencial para promover crescimento de vegetais.
- Genes associados à produção de sideróforos – que são moléculas envolvidas na captura de ferro livre no solo, tornando-o solúvel e permitindo a absorção pelo organismo.
Resultados dessas análises de vigilância da ISS são frequentemente são publicados em revistas científicas… Já temos resultados de diversas dessas análises, mas muitas outras amostras já foram coletadas na ISS! Estima-se que cerca de 1.000 ainda estão aguardando para serem analisadas e/ou um para serem enviadas para a Terra! É inegável que este é um processo muito lento (a análise dessas amostras, coletadas entre 2011 e 2016 só foram publicadas em 2021!)…
Seria muito interessante que, na própria ISS, os astronautas tivessem condições de coletarem, processarem e analisarem as amostras! – ainda que fossem análises preliminares.
E é aí que a gente termina esse post se perguntando: também seriam essas bactérias astronautas?
Antes de finalizar, finalmente, vamos ver como foi feita a descrição de Methylobacterium ajmalii – a amostra escolhida para a descrição (espécime tipo da espécie) foi a IF7SW-B2 (coletada painel do laboratório):
DESCRIÇÃO DE Methylobacterium ajmalii sp. nov.
Methylobacterium ajmalii (aj.ma’li.i. N.L. gen. n. ajmalii nomeado em homenagem a Ajmal Khan, um renomado cientista indiano da biodiversidade). As células são bastonetes Gram-negativos, móveis, aeróbios, que apresentam reações de oxidase e catalase positivas. Células possuem 1,6-1,8 µm de largura e 2,2-3,2 µm de comprimento. Colônias em ágar R2A apresentaram-se com pigmentação róseo-avermelhadas, circulares, convexas e lisas, com diâmetro de aproximadamente 0,6-1,0 mm após 3 dias de incubação em ágar R2A. o crescimento ocorre em temperatura entre 25-30 oC (ótimo, 30 oC), em pH 6.0-8.0 (ótimo, pH 7.0) e na presença de NaCl 0-1,0% (p/v) (ótimo, 0%). Nos testes comerciais API ZYM, a linhagem é positiva para fosfatase alcalina, esterase (C4), esterase lipase (c8), leucina arilamidase, tripsina, fosfatase ácida, e naftol-AS-BI-fosfohidrolase, mas negativa para outras atividades enzimáticas. As células utilizam ácido adípico, D-glicose, D-maltose, D-manitol, D-manose, L-arabinose, ácido málico, N-acetil-glicosamina, gluconato de potássio e citrato trissódico para crescimento, mas não outros substratos do teste API 20NE. Células foram capazes de realizar fermentação fraca de inulina e D-melezitose, como observado no teste API 50 CH. Ubiquinona Q-10 é a quinona isoprenoide respiratória predominante. O principal ácido graxo é “summed feature 8*” (que compreende os ácidos graxos C18:1 ω7C e/ou C18:1 ω6c). Os principais lipídios polares são difosfatidilglicerol, fosfatidiletanolamina, fosfatidilcolina e fosfatidilglicerol. O conteúdo C+G do DNA genômico da linhagem tipo é de 71,07 mol%.
*Em alguns casos, dois ou três ácidos graxos não foram possíveis de serem identificados individualmente pela metodologia empregada no estudo e, por isso, eles foram agrupados em “summed features”. No caso, a M. ajmalii apresenta um percentual elevado da summed feature 8 (C18:1 ω7C e/ou C18:1 ω6c), que a diferencia significativamente do perfil de ácidos graxos quando comparada às amostras das outras espécies.
Agora, para quem gosta da parte mais técnica, seguem mais alguns detalhes sobre os TESTES REALIZADOS
- Crescimento em diferentes temperaturas (7, 25, 30, 37 e 45 oC)
- Crescimento em diferentes pH (4.0-10.0, com intervalos de 1.0)
- Halotolerância (concentrações 0-10% de NaCl no meio de cultura)
- Motilidade (método “hanging drop”, observação no microscópio)
- Catalase (adição de solução de Peroxido de hidrogênio a 3%)
- Oxidase (papel filtro com substrato tetrametil-p-fenilenodiamina)
- Testes comerciais API 20 NE, 50 CH e ZYM
- Análise de diversos ácidos graxos celulares
- Perfil de lipídios polares
- Quinona respiratória isoprenoide
- Análise filogenética baseada no sequenciamento do rRNA 16S. MLSA de 6 genes housekeeping (atpD, recA, dnaK, rpoB, ginI e gyrB) e genoma completo (comparação com as 45 espécies do gênero Methylobacterium, além de outras espécies de diferentes gêneros e famílias – como grupos externos)


Referência:
Bijlani et al. (2021) Methylobacterium ajmalii sp. nov., isolated from the International Space Station. Frontiers in Microbiology. doi: 10.3389/fmicb.2021.639396
Aproveite e nos siga em nossas redes sociais: Twitter, Instagram e Facebook! Ah! E se for fazer comprinhas na Amazon, use nosso link!



boa tarde. eu sou estudante de bioinformatica e gostaria de saber se o genoma destas bactérias fora depositado no ncbi ou outro banco. e se não for muito abuso, existiria como disponibilizar o link dos genomas caso estes foram sequenciados. muito obrigado.
Oi, Fernando,
Conferi no artigo referenciado na postagem:
Bijlani et al. (2021) Methylobacterium ajmalii sp. nov., isolated from the International Space Station. Frontiers in Microbiology. doi: 10.3389/fmicb.2021.639396. Disponível em:
No finalzinho, tem esse trecho:
Data Availability Statement
The 16S rRNA gene sequences of Methylobacterium sp. IIF1SW-B5, and Methylobacterium sp. IIF4SW-B5 are submitted under accession numbers KY218843 and KY218865, respectively. The WGS and the raw data deposited under BioProject accession number PRJNA634337. The WGS accession numbers are mentioned in Table 1. The WGS was also deposited in GeneLab under GeneLab dataset (GLDS-300; https://genelab-data.ndc.nasa.gov/genelab/accession/GLDS339 300). The version described in this paper is the first version.
Acho que para mais detalhes você pode consultar o artigo ou entrar e contato diretamente com os autores do trabalho.
Abraço!