Agora pra não confundir Lorenz com Tinbergen..
Agora pra não confundir Lorenz com Tinbergen..
1892 – A General Electric Company é criada com a fusão da Edison General Electric Company and the Thomson-Houston Company.[A GE é talvez a maior empresa de tudo-que-se-pode-usar-num-lab-e-fora-dele. Desde aparelhos megacomplexos até reagentes e kits]
1912 – O navio RMS Titanic naufraga por volta das 02h20min após chocar cerca de três horas antes com um iceberg no Atlântico Norte.[“Nem deus afunda este navio” disse o engenheiro. E não afundou mesmo, afinal foi um iceberg]
1923 – A insulina se torna disponível para uso em larga escala por pacientes que sofrem de diabetes. [Era ainda purificada de vacas. Hoje em dia ela é produzida por células transgênicas – muito mais barata e segura por isso.]
Nascimentos:
1452 – Leonardo da Vinci, artista e cientista italiano [Provavelmente tinha transtorno em déficit de atenção e hiperatividade (TDAH) e mesmo assmi um gênio]
- 1552 – Pietro Cataldi, matemático italiano (m. 1626).
- 1641 – Robert Sibbald, físico escocês (m. 1722).
- 1707 – Leonhard Euler, matemático (m. 1783). [Fez muita coisa, inclusive o sudoku]
- 1710 – William Cullen, físico e químico escocês (m. 1790).
- 1772 – Étienne Geoffroy Saint-Hilaire, naturalista francês (m. 1844).
- 1794 – Jean Pierre Flourens, fisiologista francês (m. 1867).
- 1809 – Hermann Grassmann, matemático alemão (m. 1877).
- 1874 – Johannes Stark, físico alemão laureado com o Prêmio Nobel (m. 1957). [Descreveu o Efeito Doppler. Quem assiste Big Bang Theory sabe]
1896 – Nikolay Nikolayevich Semyonov, quimício russo laureado com o Prêmio Nobel de Química (m. 1986).
1907 – Nikolaas Tinbergen, ornitólogo holandês, laureado com o Prêmio Nobel de Medicina (m. 1988). [Pai da etologia. E a referência na wiki português é um desgosto.]





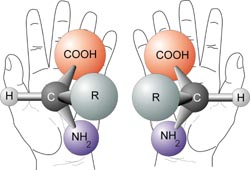
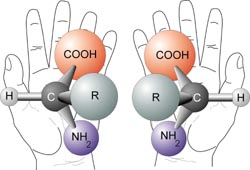 A nomenclatura D ou L vem da comparação estrutural com a molécula quiral do gliceraldeído. Quiral é o nome dado a moléculas que possuem um átomo de carbono ligado a 4 grupos químicos diferentes, de modo que sua estrutura não é sobreponível à sua imagem num espelho. É a distribuição espacial dos grupos químicos em torno desse carbono quiral que denomina, em comparação às formas do gliceraldeído, os aminoácidos L ou D.
A nomenclatura D ou L vem da comparação estrutural com a molécula quiral do gliceraldeído. Quiral é o nome dado a moléculas que possuem um átomo de carbono ligado a 4 grupos químicos diferentes, de modo que sua estrutura não é sobreponível à sua imagem num espelho. É a distribuição espacial dos grupos químicos em torno desse carbono quiral que denomina, em comparação às formas do gliceraldeído, os aminoácidos L ou D.
 Para isso, basta participar do projeto Folding@Home, idealizado pela Universidade Stanford.
Para isso, basta participar do projeto Folding@Home, idealizado pela Universidade Stanford.