Mais um acidente a favor do pesquisador
Em 1986, o pesquisador Richard Jorgensen estava trabalhando com petúnias quando aconteceu um acidente. Jorgensen desejava criar uma petúnia roxa, porém muito mais roxa do que o normal. Para alterar a cor, ele procurou super-expressar a enzima chalcona-sintase (CHS) introduzindo um gene quimérico de CHS, uma enzima limitante da via responsável pela coloração das flores [1]. Mas a alteração genética realizada teve um resultado totalmente inesperado e grande parte das pétalas se tornaram brancas e não roxo escuro como ele desejava.
O mistério só foi desvendado em 1998 e conferiu o Prêmio Nobel a dois pesquisadores americanos, gerou especulações sobre a cura de dezenas de doenças e mais recentemente se tornou uma nova ferramenta para a biologia sintética. Andrew Fire e Craig Mello descobriram que Jorgensen havia esbarrado no que eles vieram a chamar de RNA de interferência (RNAi), um mecanismo de silenciamento gênico.
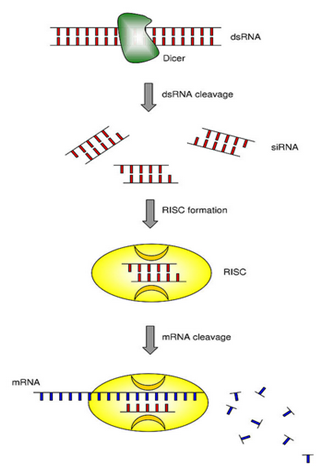
O gatilho para o mecanismo de silenciamento gênico por RNAi ocorre quando um RNA de dupla fita (dsRNA) se forma. Ao ser identificado um dsRNA, a enzima Dicer corta o dsRNA em fragmentos menores que se ligam ao complexo protéico RISC (RNA-induced silencing complex). Em seguida, apenas uma das fitas de RNA permanece presa ao complexo, que serve para ir em busca de fitas de mRNA que sejam complementares [2]. Quando um mRNA complementar é detectado, ocorre o pareamento com o RNA preso ao complexo e ele é então clivado e degradado. Como o mRNA não pode ser traduzido, o gene tem sua expressão reduzida (Figura 1). O silenciamento é uma poderosa ferramenta que agora encontrou uma aplicação na biologia sintética, por meio da evolução dirigida.
O sonho da biologia sintética de construir sistemas que funcionem de modo previsível e robusto frequentemente entra em conflito com a complexidade dos sistemas biológicos. Além de sua complexidade, o comportamento dos microrganismos dependem de um contexto, o que também dificulta o uso de partes padronizadas [3]. Logo, métodos de evolução dirigida possuem grande utilidade, já que em princípio dispensam informações detalhadas de estrutura, funcionalidade e de mecanismos de um sistema [4].
Um dos primeiros experimentos sobre evolução realizado em laboratório foi feito por William Dallinger, em 1880. Dallinger conseguiu que seus microrganismos que cresciam a 18ºC passassem a crescer a 70ºC, no entanto o experimento levou 7 anos e envolveu aumentar a temperatura de sua incubadora gradualmente até que eles fossem capazes de sobreviver. Hoje existem diversos métodos de evolução dirigida e um pouco menos demorados, entre eles o RAGE – RNAi assisted genome evolution.
RAGE é um método utilizado em Saccharomyces cerevisiae e é bastante útil quando se deseja obter fenótipos complexos. Fenótipos complexos, como a tolerância ao ácido acético, dependem da alteração de múltiplos genes e são de grande interesse para a indústria na produção de combustíveis e outros compostos químicos. O uso dessa técnica reduz a expressão de genes (knockdown) em escala genômica e possibilita identificar genes que até então não se imaginava terem papel em determinadas funções.
Para que tais genes possam ser identificados, é necessário criar uma biblioteca de RNAi. A biblioteca é criada fragmentando o DNA genômico com uma enzima de restrição e clonando os fragmentos em um plasmídeo com promotores convergentes, necessário para que RNAs de fita dupla sejam formados. Como S. cerevisiae não possui a via de RNAi, também é necessário inseri-la [6].
Via inserida e biblioteca criada o processo de evolução pode começar. Quando o knockdown de um gene for promissor, tal gene pode ser integrado e novos ciclos de transformação e screening podem ser feitos repetidamente (Figura 2), até que seu objetivo seja alcançado, ou pelo menos, chegue próximo dele.
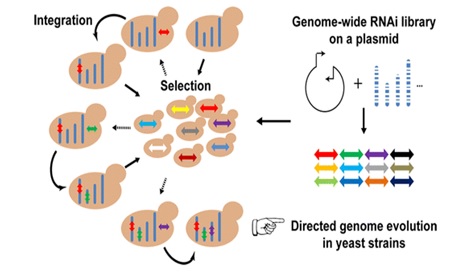
Figura 2: Evolução dirigida pelo método RAGE – Imagem retirada de: http://pubs.acs.org/doi/abs/10.1021/sb500074a
Em 2006, Fire e Melo ganharam o Prêmio Nobel em fisiologia ou medicina por desvendarem o fenômeno observado por Jorgensen. Muitas descobertas acidentais fazem parte da história da ciência, como a penicilina, o raio X e o microondas, por exemplo. O método de evolução dirigida utilizando RNAi pode também em breve facilitar a vida de muitos pesquisadores que buscam aprimorar seus microrganismos.
Referências
- NAPOLI, C.; LEMIEUX C.; JORGENSEN R. lntroduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. The Plant Cell, Vol. 2, p. 279-289.
- CLARK D. P. Molecular Biology. Vol. 2, chapter 11 (2010)
- DOUGHERTY, M. J.; ARNOLD, F. H. Directed evolution: new parts and optimized function. Current Opinion in Biotechnology, 2009, 20:1–6
- COBB, R. E; SUN,N.; ZHAO H. Directed evolution as a powerful synthetic biology tool. Methods (2012)
- SI, T.; LUOZ, Y.; BAO, Z.; ZHAO, H. RNAi-Assisted Genome Evolution in Saccharomyces cerevisiae for Complex Phenotype Engineering. ACS Synth. Biol. (2014)