FAQ #8 Como ver se a transformação gênica deu certo?
Já colocamos nossos plasmídeos nos bichinhos, mas ainda não acabou. Essa é a hora de saber se tudo que fizemos até aqui deu certo!
Primeiro, colocamos eles na placa de seleção com antibiótico e só aqueles que realmente incorporaram o plasmídeo vão sobreviver porque ganharam um gene amigo de resistência ao antibiótico. Agora pegamos uma parte das células sobreviventes e fazemos um teste usando a PCR (você lembra dessa técnica, né?).
Como testar com a PCR?! Roubamos os plasmídeos dessas células e tentamos multiplicar o pedaço de DNA que foi grudado nele adicionando primers, nucleotídeos e enzimas sob aquecimento e desaquecimento. Se o nosso plasmídeo modificado tiver sido incorporado pelos micro-organismos haverá a multiplicação do pedacinho e podemos vê-los em uma eletroforese. Caso contrário, nada foi copiado e nada vai aparecer na eletroforese. Então temos o sinal de que alguma coisa não deu certo por aqui e as chances de erros deste teste são bem pequenas.
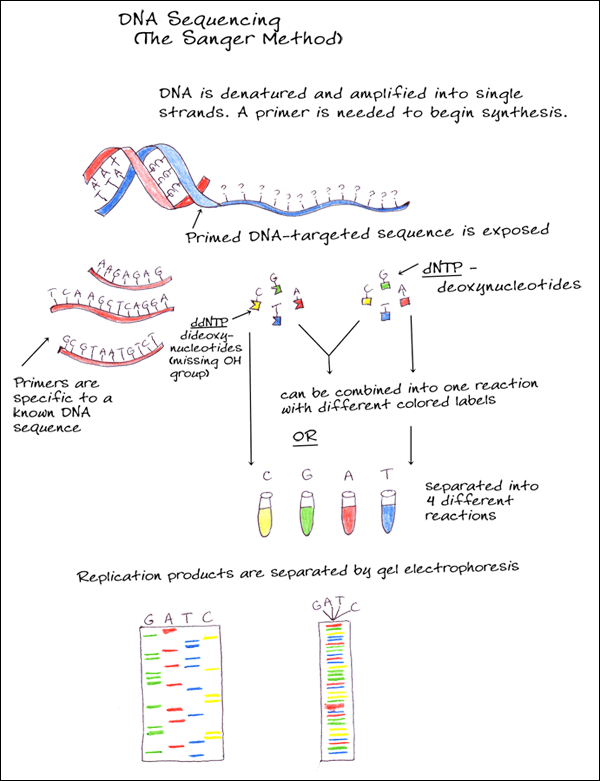
Depois vamos para um teste melhor ainda, o sequenciamento. Este teste diz exatamente qual é a sequência de pares de base da molécula do plasmídeo e acabam de vez suas dúvidas se a transformação deu certo ou não! O sequenciamento começa parecendo um processo de duplicação normal de DNA. Mas além de cadeias molde, enzimas e nucleotídeos normais, há nucleotídeos especiais sintetizados (ddNTP’s) que possuem duas boas propriedades: emitem luz e interrompem o prologamento da cadeia a partir de onde foram adicionados. Então, imagine uma molécula de DNA que possui um par de base A-T em um determinado comprimento e considere que a base A pertence à cadeia molde e a base T à cadeia complementar. Se esta base T for um nucleotídeo especial temos como identificar “quem é” e “qual sua posição” na cadeia, pois ele emitirá uma cor específica para Timina e o comprimento de sua cadeia está interrompido na posição exata (nº 2 em verde). 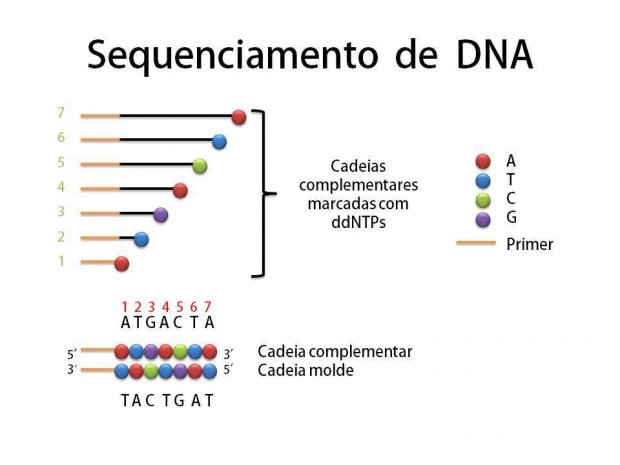 Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Para saber mais sobre sequenciamento genético, clique aqui.
Por Otto Heringer e Viviane Siratuti.
FAQs da Bioengenharia – Introdução
Preocupados com os novos amantes da biologia sintética, estamos procurando materiais de introdução no assunto e encontramos algumas coisas interessantes! 🙂
Por isso vamos colocar uma sequência de posts explicativos, partindo de uma visão geral e seguindo por perguntas mais específicas. É um pequeno tutorial-FAQ sobre alguns fundamentos que quando entendidos ajudam bastante a entender algumas de nossas discussões e outros posts deste blog. Serve para ajudar principalmente quem não é da área de biológicas ou quem ainda não se aprofundou muito neste assunto! Se continuarem com dúvidas, perguntem tá?!
Lembrem-se de ativar as legendas dos vídeos, caso necessitem! Todos possuem legendas originais em inglês e alguns em português. Você pode colocá-las traduzidas quando só houver em inglês (não muito recomendado, rs). Nos vídeos do JOVE existe a opção de colocar os textos em português. Se você não sabia que o YouTube fazia isso por você, dá uma olhada aqui (dica: barrinha inferior do vídeo).
Antes de mais nada, você sabe o que são e quais as relações entre nossas principais moléculas orgânicas (DNA, RNA e proteínas), certo? Se sua resposta é não, assista um desses vídeos!
[youtube_sc url=”http://www.youtube.com/watch?v=h3b9ArupXZg#t=0″]
[youtube_sc url=”http://www.youtube.com/watch?v=tMr9XH64rtM”]
Indo além da Estrutura
OK, agora vamos além desses conceitos básicos dos vídeos. Mais que entender a estrutura e a dinâmica dessas biomóleculas, como os cientistas a alteram? Em linhas gerais, como se faz para modificar geneticamente um organismo?
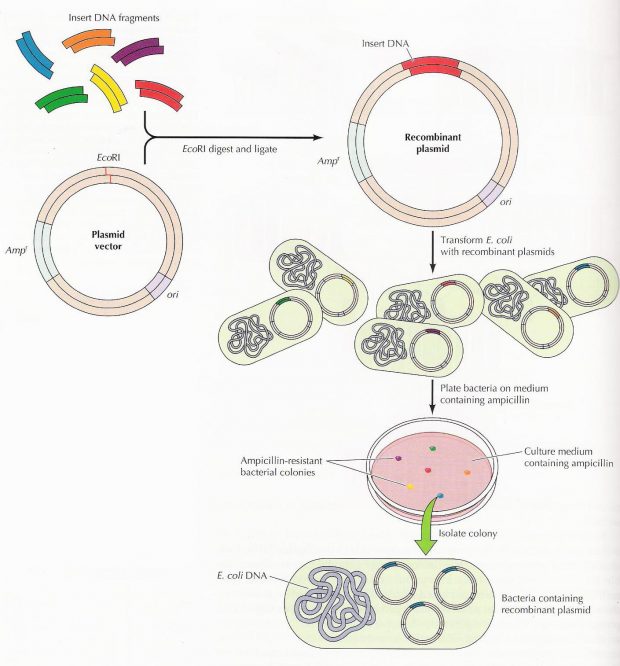
Vamos lá! Primeiro pegamos um pedacinho codificante de DNA (gene) de outro organismo que tenha certa característica que desejamos. Para isso, extraímos o DNA e depois multiplicamos só esse pedaço que nos interessa através de uma técnica chamada PCR (afinal, a eficiência das transformações é pequena e quanto mais material melhor). Precisamos também extrair e abrir os plasmídeos (DNA circular) do organismo escolhido para receber este gene e assim receber as novas características (já que o plasmídeo é nosso principal veículo de introdução dos genes nas células). Para abrir estes plasmídeos usamos enzimas de restrição que cortam as moléculas de DNA em lugares específicos e chamamos isso de digestão. Depois que tivermos vários plasmídeos e genes, podemos grudá-los com a ajuda de enzimas de ligação. E então, para saber se nosso novo plasmídeo deu certo usamos uma técnica de separação por carga e tamanho de molécula, a eletroforese em gel. Nela, as moléculas se acumulam em certas posições que nos dizem seus tamanhos (mergulhadas em um polímero) e assim podemos identificar aquelas que correspondem ao que estimamos. Além disso, fazemos testes com a técnica PCR e o sequenciamento genético. Se os testes disserem que tudo que fizemos deu certo, recuperamos o material e colocamos nossos novos plasmídeos nos organismos, geralmente utilizando choque térmico ou elétrico. Esse passo, introdução de plasmídeos, é a chamada transformação. Mas para selecionarmos somente aqueles organismos que realmente foram transformados usamos antibióticos que matam as células que não ganharam os nossos novos plasmídeos, já que não possuem os genes de resistência que foram colocados neles. E no final, cada célula que sobreviveu se multiplicará, formando colônias de organismos geneticamente modificados. Agora dá uma olhada no esquema de novo e vê se entendeu até aqui!
Por Otto Heringer e Viviane Siratuti.
Problemas com PCR!
Após desenhar o seu primer, montar a sua reação e programar o termociclador, você pode enfrentar problemas para otimizar as sua condições de PCR. Algumas simples ações podem resultar em um produto de PCR específico. Estas são as questões mais comuns aqui no laboratório:
1. Estou tendo (muitos) fragmentos inespecíficos maiores do que o esperado. O que fazer?
– Diminuir o tempo de anelamento da reação;
– Aumentar a temperatura de anelamento (aumento 2 graus a cada teste);
– Diminuir o tempo de extensão;
– Colocar menos primers;
– Checar e colocar menos DNA;
2. Estou obtendo fragmentos inespecíficos menores do que o esperado. O que fazer?
– Aumentar a temperatura de anelamento;
– Aumentar o tempo de anelamento;
– Aumentar a temperatura de extensão;
– Aumentar a temperatura de extensão para 74-78 oC;
– Colocar menos primers e/ou menos DNA.
3. A reação estava funcionando antes, mas agora eu não obtenho nenhum produto.
– Tenha certeza que todos os componentes estão na reação (tampão, DNA, primers…);
– Teste um novo Master Mix ou uma nova solução de dNTP (são sensíveis a descongelamentos consecutivos);
– Utilize um estoque novo de primers;
– Diminua sua temperatura de anelamento 6-10 oC, se não obtiver nenhum fragmento verifique todas os componentes.
Nos próximos posts vou comentar sobre técnicas de PCR: como RT-PCR, deleção de genes…
Como desenhar um primer? E como montar uma reação de PCR?
 No post passado eu comentei sobre a técnica de PCR e de como programar um PCR. Neste post eu vou um pouco mais adiante e vou falar sobre como desenhar um iniciador (primer) e como montar uma reação de PCR.
No post passado eu comentei sobre a técnica de PCR e de como programar um PCR. Neste post eu vou um pouco mais adiante e vou falar sobre como desenhar um iniciador (primer) e como montar uma reação de PCR.
O desenho do primer tem que ser feito com muito cuidado para garantir o futuro do projeto, erros no desenho podem ser percebidos apenas muito tempo depois, após muito esforço e dinheiro já ter sido gasto no projeto. Por isso, realize essa tarefa com muito cuidado, tendo em vista todo o projeto.
A primeira coisa é pegar a sequência de interesse no GenBank, como por exemplo para o gene codificador da enzima xilose isomerase de Escherichia coli. Conheça o gene, busque artigos sobre ele, veja os genes que estão envolta, como ele é regulado,… Depois salve a sequência do gene e mais ou menos 200 bp envolta dele em um documento word. Encontre o start códon, o stop códon, promotor,… e decida a sua estratégia de clonagem.
Para desenhar primers para amplificar o gene inteiro você precisará de regiões envolta do gene. Além disso existem uma série de regras a que você precisa ficar atento para desenhar o primer como: composição de G-C, tamanho do primer, temperatura de anelamento, formação de dímeros e estruturas secundárias e muito mais. Para este trabalho, eu recomendo utilizar um software, o FastPCR. Um software livre (gratuito) e amigável.
É preciso apenas colocar a sua sequência de interesse e pedir para que ele desenhe os primers, depois você precisa checar se aquela sequência amplifica a sua região de interesse. Para a encontrar o primer reverso na sua sequência tem uma ferramenta para obter o reverso complementar da sequência, já que as sequencias de primers são dadas no sentido 5´ – 3´.
Finalmente, você pode fazer um PCR in silico, para isso, volte para o Genbank e copie toda a sequência do genoma da bactéria desejada (isso pode demorar alguns minutos) e passe para o FastPCR para fazer o PCR in silico com os primers desenhados. Com isso, você pode verificar possíveis regiões amplificadas pelo seu par de primers.
Depois de mandar sintetizar os primers, você pode fazer um programa como mostrado no post anterior. Para a reação PCR você deve seguir o protocolo da sua Taq polimerase comprada. Vale a pena ressaltar que para amplificações de genes que serão expressos posteriormente, recomendo uma Taq polimerase de alta fidelidade.
No próximo post vou comentar sobre erros e soluções comuns que ocorrem com reações de PCR.
O que é um PCR? Como montar um programa de PCR?
 Polymerase chain reaction ou reação da polimerase em cadeia (PCR) é técnica de biologia molecular utilizada para amplificar (aumentar o número) de um ou de alguns pedaços de DNA em várias ordens de magnitude, gerando milhões de cópias de uma sequência particular de DNA.
Polymerase chain reaction ou reação da polimerase em cadeia (PCR) é técnica de biologia molecular utilizada para amplificar (aumentar o número) de um ou de alguns pedaços de DNA em várias ordens de magnitude, gerando milhões de cópias de uma sequência particular de DNA.
Utilizando um termociclador (aparelho que permite mudanças rápidas de temperatura), uma DNA polimerase, um par de iniciadores (primers) e dNTP´s (deoxinucleotídeos, que formarão a nova molécula de DNA) é possível sintetizar in vitro DNA a partir de uma fita molde. Veja o vídeo no youtube.
Diz a lenda que Kary Mullis teve a idéia do PCR em um daqueles momentos de ócio produtivo enquanto surfava nas ondas do Sul da Califórnia. Um ano mais tarde, 1983, a sua idéia virou realidade através do desenvolvimento da primeira máquina de PCR. Dez anos mais tarde, 1993, Mullis também foi surfar após receber a notícia que havia sido ganhador do prêmio Nobel pela sua invenção. Essa é uma ótima desculpa para não trabalhar aos finais de semana no laboratório! (rs)
A partir do programa de PCR básico abaixo é possível desenhar um programa específico para seu iniciador e fragmento a ser amplificado. No passo (step) 2, ocorre a denaturação do DNA, No passo 3, ocorre a ligação do iniciador ao seu DNA, para isso é preciso calcular a temperatura de anelamento do seu iniciador. Existe um cálculo baseado na composição dos iniciadores (Ta = 2(A + T) + 4(C + G) – 5), mas existem alguns softwares que calculam para você (porém esta temperatura não deve ser abaixo de 50oC, para evitar ligações inespecíficas). Após a ligação dos iniciadores ao DNA, irá ocorrer a síntese de DNA pela DNA Polimerase, normalmente se utiliza a Taq polimerase que possui uma temperatura ideal de 72oC. O tempo de elongamento da sua sequência depende do tamanho dela, calcula-se cerca de 1 min para cada 1000 pares de base (não menos de 30 segundos). Finalmente, este ciclo será repetido 30 vezes.
Programa básico de PCR:
Step Temperature (°C) Time (min)
1 95 2:00
2 95 0:30
3 55 0:30
4 72 1:00
5 Go to step 3, 30 times
6 4 forever
No próximo post vou comentar sobre o desenho de iniciadores (primers) e como montar uma reação de PCR!
BioBricks: fabricação de uma parte padrão.
 Até agora comentamos sobre a possibilidade de juntar diferentes componentes utilizando o padrão técnico de montagem dos BioBricks. Mas neste post vou comentar sobre a fabricação de uma parte padrão. No futuro, eu pretendo comentar sobre a síntese de DNA sintético, mas agora vou explicar como montar um BioBrick utilizando o método de PCR. O aparelho para realizar um PCR, o termociclador, está presente na maioria dos laboratórios de biologia molecular, e hoje se apresenta como uma ferramenta básica para este tipo de atividade. Existem centenas de livros e sites na Internet que podem te familiarizar com a técnica. Primeiramente, um BioBrick pode ser construído via PCR se existe algum molde de DNA do qual o BioBrick possa ser amplificado (por ex., um gene de alguma bactéria) ou se a parte é pequena o suficiente que possa ser criada através do alinhamento e extensão do primer (iniciador). Nos dois casos é necessário adicionar na extremidade 5′ dos primers as sequências referentes aos sítios das enzimas de restrição presentes nos BioBricks, os sufixos e prefixos (ver post anterior).
Até agora comentamos sobre a possibilidade de juntar diferentes componentes utilizando o padrão técnico de montagem dos BioBricks. Mas neste post vou comentar sobre a fabricação de uma parte padrão. No futuro, eu pretendo comentar sobre a síntese de DNA sintético, mas agora vou explicar como montar um BioBrick utilizando o método de PCR. O aparelho para realizar um PCR, o termociclador, está presente na maioria dos laboratórios de biologia molecular, e hoje se apresenta como uma ferramenta básica para este tipo de atividade. Existem centenas de livros e sites na Internet que podem te familiarizar com a técnica. Primeiramente, um BioBrick pode ser construído via PCR se existe algum molde de DNA do qual o BioBrick possa ser amplificado (por ex., um gene de alguma bactéria) ou se a parte é pequena o suficiente que possa ser criada através do alinhamento e extensão do primer (iniciador). Nos dois casos é necessário adicionar na extremidade 5′ dos primers as sequências referentes aos sítios das enzimas de restrição presentes nos BioBricks, os sufixos e prefixos (ver post anterior).
A construção de BioBricks contendo sequências codificadoras de proteínas requer um sufixo e um prefixo um pouco mais especializados por duas razões:
1. o prefixo é alterado para garantir o espaçamento entre o sítio de ligação do ribossomo e o códon ATG de início.
2. BioBricks que codificam proteínas possuem, por padronização, dois códons de sequência TAA de parada.
Ao construir os primers, basicamente, é necessário “copiar e colar” a seguinte sequência de 31 bp no fim 5′ no seu primer upstream (ou iniciador universal) desenhado para o seu fragmento de DNA de interesse:
5′ —> 3′
GTT TCT TCG AAT TCG CGG CCG CTT CTA G ATG…
start codon (no caso de um gene)
E “copiar e colar” os seguintes 35 bp no fim 5′ do seu primer downstream (iniciador reverso):
5′ —> 3′
GTT TCT TCC TGC AGC GGC CGC TAC TAG TA TTA TAA….
duplo stop codon!
A estas sequências deve-se somar aproximadamente 20 bp de sequência de primer relativa a região codificadora. Para este tipo de clonagem se utiliza uma Taq polimerase com alta fidelidade e um kit de clonagem TOPO-TA cloning.
Claro que todas as outras precauções para se desenhar um primer ainda são válidas, como a verificação de sítios de restrição nas sequências. Para mais detalhes, veja o protocolo disponibilizado para a fabricação de BioBricks.