A Indústria Química do Futuro

Fig. 1 – “Bioreator”, arte de K-Kom – DeviantART.
“Um Químico é um mexedor de sopa, um operador fedorento.
Um Zimologista é alguém que ajuda a manter bilhões de pessoas vivas. Eu sou um especialista em cultura de leveduras. (…)
Este laboratório mantém a New York Yeast funcionando. Não há um dia, nenhuma maldita hora, que nós não tenhamos culturas de cada linhagem de levedura na empresa crescendo em nossas caldeiras. Nós checamos e ajustamos os fatores dos requisitos de comida. Nós garantimos que estão se reproduzindo bem. Nós ‘dobramos’ a genética, começamos as novas linhagens e as ‘limpamos’, descobrimos suas propriedades e moldamos elas novamente.(…)
Vinte anos atrás a Saccharomyces olei Benedictae era só uma linhagem com gosto horrível de sebo e boa para nada. Ela ainda tem gosto de sebo, mas seu conteúdo de lipídios foi forçado de 15 para 87 por cento. Se você usou a Esteira Expressa hoje, apenas se lembre que ela é lubrificada estritamente com S. o. Benedictae, linhagem AG-7. Desenvolvida aqui mesmo nesta sala.”Trecho de “As Cavernas de Aço” (1953), de Isaac Asimov – tradução livre.
![]() Dia 2 de Janeiro é aniversário do “bom doutor” Isaac Asimov, que já nos anos 50 descrevia um planeta Terra apinhado de pessoas em enormes conjuntos de construções urbanas, as “Cavernas de Aço”, título do livro que faz parte de sua série de romances sobre robôs que o fez ser um ícone da ficção científica. Mas um fato pouco lembrado é sua visão de como seria a indústria química do futuro, que se reflete até os dias de hoje. Antes de escritor, Asimov era bioquímico e portanto não é à toa que no mesmo ano em que foi descoberta a estrutura do DNA, ele já conseguisse prever as possibilidades industriais da manipulação genética como no trecho acima de seu livro. Nele um “zimologista” defende a importância de seu trabalho em criar leveduras que possam produzir os mais variados tipos de químicos, num futuro onde o petróleo mundial já se esgotou e a única fonte de matéria-prima é biomassa vegetal.
Dia 2 de Janeiro é aniversário do “bom doutor” Isaac Asimov, que já nos anos 50 descrevia um planeta Terra apinhado de pessoas em enormes conjuntos de construções urbanas, as “Cavernas de Aço”, título do livro que faz parte de sua série de romances sobre robôs que o fez ser um ícone da ficção científica. Mas um fato pouco lembrado é sua visão de como seria a indústria química do futuro, que se reflete até os dias de hoje. Antes de escritor, Asimov era bioquímico e portanto não é à toa que no mesmo ano em que foi descoberta a estrutura do DNA, ele já conseguisse prever as possibilidades industriais da manipulação genética como no trecho acima de seu livro. Nele um “zimologista” defende a importância de seu trabalho em criar leveduras que possam produzir os mais variados tipos de químicos, num futuro onde o petróleo mundial já se esgotou e a única fonte de matéria-prima é biomassa vegetal.

Fábrica da BASF em 1886 em Ludwigshafen, onde é a sede da empresa até os dias de hoje. Pelo menos olhando de longe, não é muito diferente das fábricas de hoje em dia, não? – Fonte: WIkimedia
Essa “bioindústria do futuro” nunca foi tão real como hoje. E já estava na hora. O cerne da maior parte da indústria química mundial ainda é a mesma desde quando o Brasil ainda era um império, mais ou menos na mesma época em que o petróleo começa a ser usado como principal fonte para fabricar produtos químicos industriais. Naquele tempo era impensável que os pequenos seres humanos poderiam causar um impacto ambiental tão grande num mundo tão vasto. Mas está ficando cada vez mais claro para a população de 7,7 bilhões de pessoas que esse modelo de 200 anos precisa mudar, ou ainda vamos ter que aceitar conviver com catástrofes climáticas.
Todo esse tempo de existência dá um privilégio enorme para esse sistema de produção permanecer desse jeito. Foi só com uma verdadeira revolução científica que a indústria de hoje ficou pronta para começar a mudar: a revolução da decodificação genética. Quando os cientistas terminaram de montar o quebra-cabeça que explicava como o DNA guardava instruções para as reações químicas que coisas vivas conseguem fazer, um universo de possibilidades se abriu. Tudo isso culminou nos anos 2000, com uma nova abordagem do que um dia foi conhecido como engenharia genética, a Biologia Sintética. Tratando células da mesma maneira como engenheiros elétricos tratam circuitos eletrônicos, os avanços da aplicação dessa nova disciplina foram muito mais rápidos do que a manufatura de químicos e hoje a bioindústria não apenas existe mas já prospera. A manufatura de químicos por rotas biológicas já está em estágio comercial (ver Figura 3) e a OCDE (o “clube dos países ricos”) estima que em 2030 produtos derivados dessa indústria corresponderão até 2,7% do PIB de seus países membros – para se ter uma ideia, é um valor próximo da riqueza média que toda a agricultura, pesca e extração de madeira geram nesses países hoje em dia.
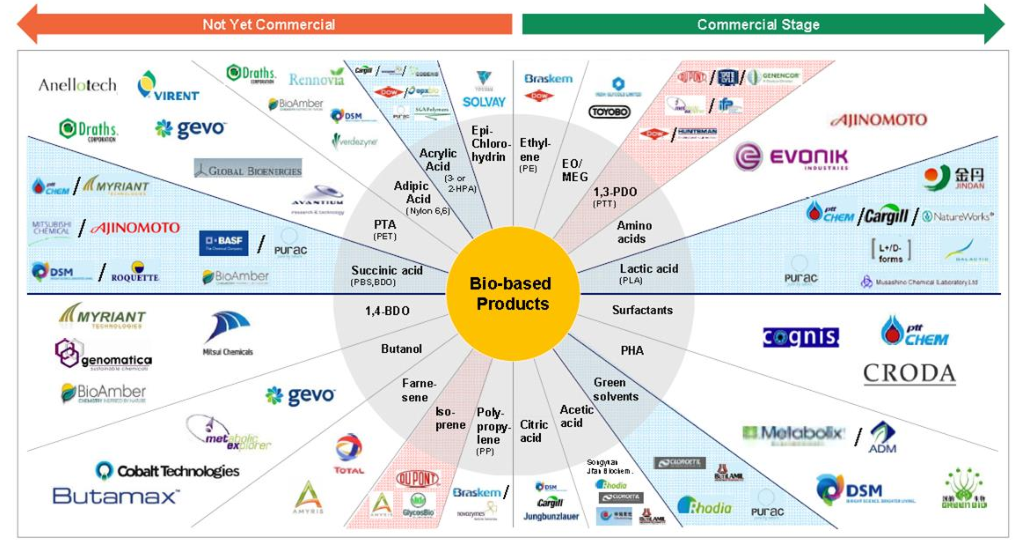
Figura 3: Exemplos de casos de sucesso de manufatura de químicos por rotas biológicas. Retirado de “Industrialization of Biology”, National Research Council, 2015. (Clique para ver melhor)
Boa parte das pessoas está completamente alheia a essas mudanças, muito porque falta algum tempo até verificarmos as estimativas para 2030 e ainda estamos vivendo só começo da mudança de “mexedores de sopa” para “zimologistas” na indústria. Até agora a Biologia Sintética foi a disciplina que melhor atendeu as expectativas do que seria o futuro da bioindústria, mas ainda estamos longe de produzirmos tudo a partir de microrganismos. Hoje a fronteira com o futuro está na imprevisibilidade e complexibilidade dos sistemas biológicos. Apesar da analogia ser válida, “programar” microrganismos não é a mesma coisa que computadores: são tantas variáveis ainda mal compreendidas dentro de uma simples célula que as coisas simplesmente não funcionam como deveriam e muitas vezes os cientistas não sabem exatamente o porquê. Pensando em focar esforços, vários pesquisadores convocados pelo conselho nacional de pesquisa norte-americano montaram um plano para os próximos 10 anos com os desafios a serem superados para uma “manufatura avançada de químicos” a nível industrial (ver Figura 4). Dentre os principais desafios estão o barateamento dos processos de uso de matérias-primas renováveis, aumento da eficiência dos processos de ganho de escala de bioprodução, desenvolvimento de ferramentas mais ágeis de construção de sistemas genéticos e “domesticação” de microrganismos ainda não explorados para uso industrial.
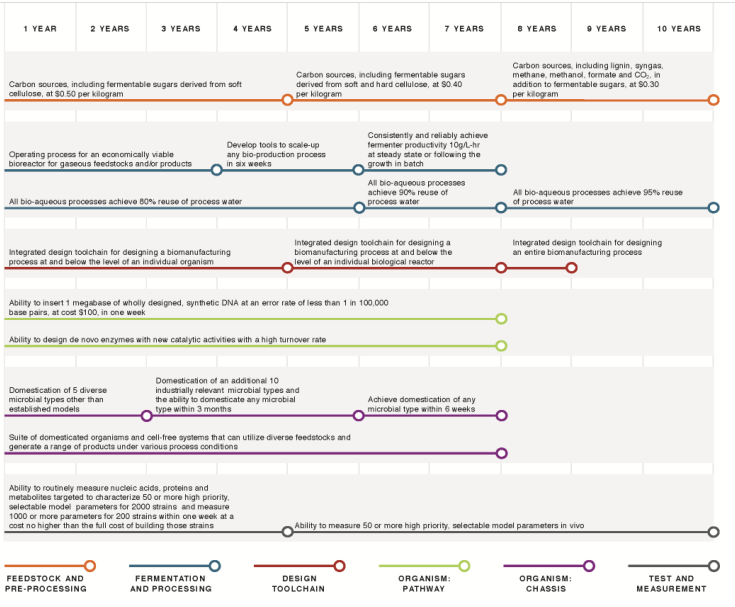
Figura 4: Planejamento de desafios a serem superados nos próximos 10 anos para concretização do potencial industrial da biotecnologia. Fonte: “Industrialization of Biology”, National Research Council, 2015. (Clique para ver melhor)
Parte dos executores desse plano são novos de laboratórios de biotecnologia chamados de “biofoundries”. São basicamente laboratórios altamente automatizados (Figura 5), capazes de realizar uma quantidade sem precedentes de experimentos e fazer medições, quase 24 horas por dia, 7 dias por semana. A estratégia é usar a “força bruta” das máquinas para conseguir lidar com uma quantidade enorme de variáveis, o que seria impraticável em outras condições.

Figura 5: “Biofoundry” versus um Laboratório de Biotecnologia comum. As bancadas de trabalho da Biofoundry são tomadas por robôs que automatizam todos os processos com baixa intervenção humana, enquanto um laboratório comum apenas produz conforme o número de pessoas que é possível caber numa mesma bancada! – Fonte: Ginkgo Bioworks (imagem da esquerda) e Wikimedia (imagem da direita). (Clique para ver melhor)
Em alguns lugares do mundo, como nos EUA, já se inicia um processo de estratificação de um ecossistema de empresas de biotecnologia e indústria: enquanto “biofoundries” fazem o “design” dos microrganismos (como a Ginkgo Bioworks, Genomatica e Zymergen), grandes empresas investem em bioprodução de commodities químicas (como Braskem, Dow, Basf) e moléculas de alto valor agregado, ao mesmo tempo que startups surgem barateando sequenciamento e síntese de DNA (como a Twist Biosciences) e viabilizando softwares necessários para toda a automação funcionar bem (como a Teselagen – que NÃO é uma empresa hipster reinventando a tecelagem, como pode parecer). Como a matéria prima desse tipo de indústria não é o petróleo, seria possível por exemplo existir um sistema de produção mais distribuído, como acontece hoje com as microcervejarias regionais: a manufatura acontece em muitas fábricas de pequena escala, usando fontes locais de material para servir de alimento para os microrganismos.
Nesse futuro muito próximo, a riqueza da biodiversidade é o código de DNA. É como se todo organismo, seja ele microscópico ou gigantesco como um Jatobá, fosse uma máquina biotecnológica que a própria natureza produziu, com “programas” para fabricação de tudo o que o organismo é capaz de fazer. Bastaria então fazer o “download” desse programa, estudá-lo, e fazer “upload” dele em espécies de microrganismos industriais.
O jeito que a indústria química fabrica produtos hoje em dia é baseado em “rotas de síntese”. Ou seja, para se chegar em uma molécula, é preciso planejar todas as reações químicas, que devem acontecer do jeito certo, para no final se ter em grande quantidade o produto que se deseja. E essas rotas não surgem “do nada”, são resultados de anos de pesquisa teórica e prática; uma criação verdadeiramente humana. A visão da “bioindústria” química do futuro aproveitaria as “rotas” programadas nos “softwares” da Natureza. Dentro desse imaginário, a riqueza da biodiversidade está em descobrir novas rotas “prontas” inclusive para moléculas químicas que já existem. Daí a importância da “domesticação” de novas espécies para uso industrial. Hoje por enquanto é possível se contar com os dedos de apenas uma mão a quantidade de espécies que são mais usadas para bioprodução industrial – e sim, a levedura S. cerevisiae sugerida por Asimov está entre elas. Quantas espécies de microrganismos ainda não conhecidos pela ciência podem existir em uma grama de solo amazônico, por exemplo? Enquanto engenheiros biológicos se esforçam para pressionar essas poucas espécies a produzir mais “bioproduto”, podem haver centenas de espécies que, se exploradas geneticamente, poderiam produzir muito mais e muito mais facilmente do que a melhor cepa que já se conseguiu construir.
É claro que ainda existe toda uma indústria química para se “biotecnologizar”, mas já é um grande feito ter as principais commodities químicas sendo bioproduzidas e comercializadas de maneira competitiva com a indústria petroquímica. Isso mostra o peso dos imaginários sociotecnológicos que criamos: mesmo com grandes desafios a serem resolvidos, as consequências políticas e sociais dessas expectativas atraíram e atraem investimento suficiente para tornar real aquilo que a pouco tempo atrás era mera ficção científica. Esse imaginário de uma biotecnologia futurista já tem até organização política representativa no Brasil: a “frente parlamentar da bioeconomia”, que defende os interesses das bioindústrias emergentes, em articulação com o agronegócio.
Asimov com certeza sabia que uma posição visionária precisa de expectativas familiares o suficiente para produzir um futuro reconhecível. A série “Black Mirror” (se você não conhece, fica a dica!) usa essa mesma receita. Se essas expectativas forem muito diferentes da experiência coletiva ninguém a levará a sério, e talvez seja por isso que as histórias de Asimov parecem envelhecer pouco com o passar das décadas: elas se baseiam em imaginários sociais sólidos que existem até hoje, e que são provavelmente tão importantes quanto as próprias tecnologias para dar forma ao futuro.
E aí, já imaginou? É assim que o futuro começa.
Referências
- National Academies Press (2015).
Industrialization of Biology: A Roadmap to Accelerate the Advanced Manufacturing of Chemicals
Report of National Research Council of the National Academies Epub ISBN: 0-309-31655-3 - ASIMOV, Isaac. The caves of steel. 1954. New York: Spectra, 1991.
- HILGARTNER, Stephen; MILLER, Clark; HAGENDIJK, Rob (Ed.). Science and democracy: making knowledge and making power in the biosciences and beyond. Routledge, 2015.
- https://www.camara.leg.br/noticias/560651-camara-lanca-frente-parlamentar-mista-da-bioeconomia/
Especulações e Biologia Sintética
 Minha vontade de discutir e aprender biologia, especialmente tudo aquilo que envolve a palavra DNA já me acompanha antes mesmo de entrar na faculdade. A solução que encontrei para suprir essa vontade e viagens por esse mundo, até então feitas apenas acompanhado de meu computador foi prestar uma prova de seleção para dar aulas de biologia no cursinho da UNESP.
Minha vontade de discutir e aprender biologia, especialmente tudo aquilo que envolve a palavra DNA já me acompanha antes mesmo de entrar na faculdade. A solução que encontrei para suprir essa vontade e viagens por esse mundo, até então feitas apenas acompanhado de meu computador foi prestar uma prova de seleção para dar aulas de biologia no cursinho da UNESP.
Comecei dar aulas em 2012 e o melhor, da matéria que mais gosto, citologia e genética, afinal, eu queria falar de células, DNA, Mendel e não sobre insetos que possuem três pares de patas e um de antenas.
Certo dia, véspera de feriado e com chuva, resolvi passar um filme para discussão. O filme escolhido foi GATTACA. Ao final do filme, um aluno com os olhos arregalados que aparentavam demostrar grande interesse pelo assunto, levanta a mão e pergunta: “Esse tipo de previsão é possível?”
[youtube_sc url=”http://www.youtube.com/watch?v=L8yYjKa36No”]
GATTACA trata-se de um filme hollywodiano de ficção científica, infelizmente não poderemos prever a expectativa de uma pessoa ao nascer, mesmo uma década após a conclusão do Projeto Genoma. Hoje sabemos que os organismos dependem de dois tipos de informação, uma delas é o código genético e a outra são estímulos recebidos do meio ambiente em que vivem, sobre o qual não podemos fazer previsões.
O Projeto Genoma Humano, considerado por muitos a bola de cristal do futuro, foi cercado de promessas e esperanças da mídia e de Wall Street para o lançamento de medicamentos personalizados e previsões sobre riscos de doenças como Alzheimer e cânceres. Tal projeto prometia revolucionar a medicina, ao estilo do filme de ficção científica.
Pouco mais de dez anos já se passaram desde sua conclusão, uma batalha inciada em 1990 travada entre a iniciativa pública e a privada, que chegou ao fim depois de três bilhões de pares de bases sequenciadas e três bilhões de dólares gastos, além de uma briga entre o famoso James Watson e Craig Venter, marinheiro nas horas vagas. Para Nicholas Wade do NYT, o projeto falhou e não trouxe os benefícios tão esperados.
Embora as promessas aguardadas por muitos não chegaram, ele trouxe avanços significativos para a área da biologia. O Projeto Genoma Humano foi um projeto interdisciplinar, contando com a participação de cientistas de diversas áreas como computação, matemática, biologia entre outras, além da colaboração de laboratórios ao redor do mundo, inclusive do Brasil. Ele contribuiu significativamente para o avanço na área de biocomputação e nas técnicas de sequenciamento de DNA, além de permitir a democratização da informação por meio do acesso gratuito ao sequenciamento humano e de diversos outros seres vivos. Genes agora não precisam mais ser estudados de forma individual, hoje dispomos de uma extensa lista de partes biológicas que tem permitido o avanço na área de biologia de sistemas, que promete revolucionar o modo de tratar doenças e o sistema de saúde conhecido atualmente.
Tal fato nos faz refletir sobre as promessas em torno da área de biologia sintética, a qual acredita-se ser capaz de solucionar muitos de nossos problemas atuais como o elevado preço do petróleo, desastres ambientais e uma população cada vez maior para se alimentar. Ao invés de manipular alguns genes de cada vez, como se tem feito a mais de três décadas, graças a descoberta das enzimas de restrição, o campo da biologia sintética vai além e tem como objetivo recodificar e redesenhar circuitos genéticos. Parece não haver limites do que podemos fazer, no entanto tais circuitos não se comportam como circuitos eletrônicos. Sistemas assim costumam ter muita interferência e comportamento imprevisivel, além de estarem sujeitos a mutações. Segundo o cientista Christopher Voigt, “A célula é uma espécie de burrito, tudo dentro dela está misturado” e sujeito a uma variedade de reações bioquímicas, tal complexidade tem limitado o tamanho dos circuitos criados. Drew Endy, um dos pioneiros do ramo, acredita que a biologia sintética só atingirá seu potencial quando os cientistas forem capaz de prever com exatidão como um circuito genético se comportará dentro de uma célula. Para isso tem-se aplicado estratégias de engenharia, como a padronização, dissociação e a abstração.
Outro problema enfrentado é a caracterização das partes biológicas. Embora o Registry of Standard Biological Parts possua mais de cinco mil partes disponíveis, menos da metade tem seu funcionamento confirmado. Caracterizar algumas partes biológicas pode levar anos e não gerar dados para uma publicação. Na cultura do “publique ou pereça”, o que temos é uma grande quantidade de trabalhos, porém com poucos dados reproduzíveis e úteis.
Embora o custo de sintese e sequenciamento tenham caido de forma significativa facilitando o avanço da área, não podemos negar que ainda há muitas pedras no caminho. Nosso conhecimento ainda é limitado mesmo para compreender por completo o comportamento de organismos mais simples como bactérias e fungos unicelulares. Assim como o projeto genoma a biologia sintética também está rodeada por especulação e não podemos dizer onde estaremos em dez anos. Mas uma coisa é certa, aprendemos a ler agora estamos escrevendo código genético.
Referências
- The Promise and Perils of Synthetic Biology – Jonathan B. Tucker and Raymond A. Zilinskas
- Synthetic biology: from hype to impact – Timothy S. Gardner
- Five hard truths for synthetic biology – Roberta Kwok
- Initial impact of the sequencing of the human genome – Eric S. Lander
- http://web.mit.edu/newsoffice/2012/complex-biological-circuit-1007.html
Polimerase Por Segundo
![]() A Biologia é imprecisa por natureza, e vice versa. Isso é uma grande dificuldade ao se fazer design de sistemas biológicos sintéticos; aquilo que é muito bonito no papel às vezes nunca pode ser feito por motivos obscuros e por excesso de ruído dos sinais do sistema. Não dá pra prever. Na tentativa de deixar dispositivos sintéticos mais previsíveis, a Biologia Sintética tenta padronizar não somente partes biológicas, mas também os sinais que a compõem a dinâmica de seu sistema. Esses sinais são justamente a passagem de informação entre DNA e o fenótipo desejado, mas… como diabos deixar isso mais preciso e medir a velocidade dessa passagem de informação? Como medir “Polimerases Por Segundo”?
A Biologia é imprecisa por natureza, e vice versa. Isso é uma grande dificuldade ao se fazer design de sistemas biológicos sintéticos; aquilo que é muito bonito no papel às vezes nunca pode ser feito por motivos obscuros e por excesso de ruído dos sinais do sistema. Não dá pra prever. Na tentativa de deixar dispositivos sintéticos mais previsíveis, a Biologia Sintética tenta padronizar não somente partes biológicas, mas também os sinais que a compõem a dinâmica de seu sistema. Esses sinais são justamente a passagem de informação entre DNA e o fenótipo desejado, mas… como diabos deixar isso mais preciso e medir a velocidade dessa passagem de informação? Como medir “Polimerases Por Segundo”?
Padronização da Transmissão de Informação
Independente do que um aparelho elétrico faça, existem sinais “universais” que pertencem a todos eles: variações na diferença de potencial, na corrente, no campo elétrico e etc. A transmissão de informação entre os dispositivos eletrônicos que compõem esse aparelho são dadas justamente através desses sinais, fazendo todo o sistema elétrico funcionar. Em circuitos genéticos, sinais análogos à corrente elétrica são as taxas de transcrição e tradução, ou mais especificamente, a velocidade com que – respectivamente – uma polimerase e um ribossomo “leêm” seus nucleotídeos. O problema é que esses sinais (as taxas de transcrição e tradução) não são bons como transmissores de sinais. Entenda o porquê:
PoPS e RiPS: Qual é o sentido disso!?
Para que um transmissor de sinal seja bom, ele precisa facilitar com que dispositivos possam ser facilmente combinados em um sistema – além de ser algo “universal”, como foi dito anteriormente. Foi aí então que, usando experiências da engenharia, os biólogos sintéticos cunharam o termo “PoPS” (Polimerase Per Second – Polimerase Por Segundo) e “RiPS” (Ribossome Per Second – Ribossomo Por Segundo). Muitos pesquisadores acham que a criação desses novos termos é como “reinventar a roda”: qual seria a grande diferença entre isso e as clássicas taxas de transcrição e tradução? A diferença é a abrangência da nova medida. Quando se trata de um sítio operador, um RBS, um RNAm e o próprio gene sendo “lido”, não há diferença alguma em se medir uma taxa de transcrição e o “PoPS” ou uma taxa de tradução e o “RiPS”. Mas faz sentido se medir a taxa de transcrição de um sítio terminador por exemplo!? Esse elemento de DNA, que teoricamente não é transcrito (é ele quem justamente para a transcrição), ainda pode eventualmente ter um “leak” e permitir a passagem de uma polimerase. Usar a expressão “… a taxa de transcrição de um sítio terminador …” não faz sentido nenhum, mas acontece. Se usarmos PoPS, que por definição é o número de vezes que uma RNA polimerase passa por um ponto específico de uma molécula de DNA por unidade de tempo, ainda há sentido, pois nessa definição não importa qual a região do DNA a Polimerase passa. É esse tipo de generalidade que permite o fácil uso e novas combinações de dispositivos sintéticos.
Hierarquia de Abstração
Com a criação de sistemas fáceis de se integrar, os engenheiros biológicos podem se beneficiar de métodos largamente praticados em qualquer campo da engenharia, como a hierarquia de abstração. Com isso é mais simples se lidar com a complexidade de sistemas biológicos quando se omite informações desnecessárias. Desse modo (ver imagem abaixo), alguém trabalhando no nível de abstração das partes biológicas não precisa se preocupar com o design e síntese do DNA que usará, do mesmo modo, alguém trabalhando no nível sistêmico precisa pensar em apenas quais dispositivos incluir e como conectá-los para realizar uma função desejada, sem precisar se preocupar com os outros níveis de abstração.

Imagem retirada de: D. Baker, G. Church, J. Collins, D. Endy, J. Jacobson, J. Keasling, P. Modrich, C. Smolke, and R. Weiss. ENGINEERING LIFE: Building a FAB for biology. Scientific American, pages 44–51, June 2006.
Como medir PoPS?
A maioria dos sistemas criados e estudados hoje em dia em Biologia Sintética envolve controle transcricional da atividade genética, o que faz do PoPS a variável mais difundida na área, principalmente pelas pesquisas envolvendo lógica booleana em sistemas genéticos (portanto não é muito comum encontrar “RiPS” em artigos por aí).
Não existe um método direto para se medir PoPS, mas é possível chegar em seu valor indiretamente através de medições de fluorescência de genes reporter. É possível – se você puder encontrar os parâmetros na literatura ou medí-los – encontrar o PoPS de um dispositivo em cinco passos:
Cinco Passos Para o PoPS
1. Ligação de um Gene Repórter como Output
Antes de mais nada, será preciso de um fluorímetro (é claro) e demum espectofotômetro para medir densidade celular. Como exemplo, vamos observar a parte BBa_F2620:
Esse BioBrick tem como “entrada” a substância de quorum sensing 3-oxohexanoil-homoserina lactona e tem como “saída” PoPS. Em presença de 3OC6HSL, o gene que produz o fator de transcrição luxR promove a transcrição de genes após o Lux pR, na parte final do BioBrick BBa_F2620. Para mensurar o quão ativo o luxpR fica, liga-se outro BioBrick no final do dispositivo para mudar o output do sistema colocando-se o BBa_E0240 – a ORF (Open Reading Frame) do GFP (Green Fluorescent Protein):
Assim tem-se uma nova parte, o BioBrick BBa_T9002:
2. Medição da Fluorescência e Absorbância e Subtração do Background
Para medir a fluorescência do GFP e a absorbância da amostra de células, é preciso criar dois controles: um da absorbância (A) e outro da fluorescência (G). O controle da fluorescência será o próprio BBa_T9002 sem ser induzido pela substância de quorum sensing (G_não-induzido), enquanto o controle da absorbância é feita da maneira trivial, verificando somente a absorbância do meio de cultura (A_background). Para se obter os reais valores de Fluorescência induzida por 3OC6HSL e da densidade celular, basta então subtrair esses valores de background com os valores medidos durante a indução pela substância de quorum sensing:
3. Correlação com a Curva Padrão
Com as correções em mãos, outro procedimento trivial a ser feito é encontrar a curva padrão de fluorescência versus GFP e de absorbância versus número de células. Por exemplo, experimentos feitos em laboratório chegaram a essas retas de correlação de valores:
Em que UFC é “Unidade Formadora de Colônia” – o número de células na amostra. E “GFP” seria o número de moléculas de GFP medidas.
4. Interpolar a Curva de GFP versus Tempo Obtida na Medição
A síntese total de GFP por célula (S_célula) é dada pela taxa de produção de GFP total (S_total) dividida pelo número de células (UFC):
Para encontrar a derivada de [GFP] por tempo, basta plotar os dados de GFP obtidos por tempo e interpolar com uma função logística (provavelmente) para obter a equação que melhor descreve a variação de GFP no tempo.
5. Colocar os Valores Nessa Equação Aqui
Depois de determinada a função Scélula, basta colocá-la nessa fórmula e encontrar o PoPS:
Em que:
a = Taxa de maturação do GFP – 1/s
GammaM = Constante de degradação do RNA – 1/s
GammaI = Constante de degradação do GFP imaturo – 1/s
Rô = Constante de síntese proteica por RNAm (RiPS) – [Proteína]/[RNAm].s
PoPS = Polimerase por segundo – [mRNA]/[DNA].s
CUIDADO: Conteúdo Matemático – Prossiga com Cuidado (Ou não…)
Chega-se nessa expressão através de um pequeno sistema de equações diferenciais:
As equações expressam uma dinâmica simplificada de um sistema de transcrição e tradução de uma informação genética. Para chegar na expressão de PoPS, basta substituir a última equação na segunda e isolar M. Com a expressão resultante, basta substituir a variável M na primeira equação e sua derivada em dM/dt.
ATENÇÃO: Aqui acaba o conteúdo matemático. Está tudo bem agora.
E essa é a história de como você pode encontrar o PoPS – essa variável estranha! – no seu próprio laboratório (ou ao menos entender do que se trata). Assim como um circuito elétrico, que pode ser montado da melhor maneira possível e mesmo assim não funcionar por razões obscuras, sistemas biológicos têm muito mais esse problemático costume de não se comportar como esperado. Contudo essa abordagem mais generalista da atividade transcricional de uma célula é uma boa maneira de se tentar enfrentar o grande desafio de se deixar a biologia mais “engenheirável” e mais “precisa”. Não que essa seja a coisa mais fácil do mundo, mas ela nunca será se ninguém tentar. E estamos aos poucos conseguindo.
Referências:
- Bio FAB Group, Baker D, Church G, Collins J, Endy D, Jacobson J, Keasling J, Modrich P, Smolke C, & Weiss R (2006). Engineering life: building a fab for biology. Scientific American, 294 (6), 44-51 PMID: 16711359
- http://partsregistry.org/Part:BBa_F2620:Experience/Endy/Data_analysis
- http://openwetware.org/wiki/Endy:Measuring_PoPS_on_a_plate_reader
Synbio: uma revolução da… Informação?
Nossos tempos andam bem corridos. Com certeza a pessoa mais atabalhoada da década de 80 diria o mesmo se pudesse se comparar com uma pessoa “normal” desse mundo entupido com mais de 7 bilhões de pessoas. Não que hoje em dia o tempo tenha sofrido uma alteração devido a uma curvatura do espaço-tempo causada pelo enorme número de pessoas acumuladas nas cidades – o que aliás seria bem forçado; a não ser que uma enorme massa vinda do espaço venha para cá, mas aí não haveriam mais pessoas para perceber o tempo -, o mesmo 1 minuto da década de oitenta ainda é o mesmo do nosso tempo – mesmo que hipotéticamente os indivíduos das diferentes épocas percebessem o tempo de maneiras relativamente diferentes por algum motivo desconhecido, cada um em sua época ainda contaria os mesmos 60 segundos para contabilizar 1 minuto -, o que muda mesmo é como percebemos esse tempo passando. Quando muitas coisas acontecem temos muitos referenciais para distinguir um momento diferente de outro, e em um mundo com muitas informações sendo captadas (ou pelo menos emitidas) de uma maneira muito rápida, a nossa sensação de passagem de tempo é bem maior. Temos muitas coisas para distinguir como referenciais a todo momento e de uma maneira muito rápida. Essa é a cara do nosso tempo que deixa o homem mais “sem tempo” dos anos 80 no chinelo: Informação.
Informação por Um, Informação por Todos
Isso mesmo. Uns europeus sissudos do CERN, ripongas do vale do silício, e um bando de asiáticos espertos conseguiram mudar o mundo de uma maneira irreparável criando a internet, popularizando o computador (tornando-o pessoal) e deixando-os muito menores e mais baratos. Um grande fato é que a maioria dessas coisas já existiam antes do grande povo (nós!) botar as mãos nessas tecnologias. Em grande centros de pesquisa, é claro. Exatamente por isso que a palavra revolução se encaixa muito bem quando dizemos que o mundo passou por uma revolução da informação: em sua maioria, revoluções políticas – como a francesa, a bolivariana, a russa e etc – um poder passa de um lugar limitado para uma enorme massa de pessoas. O mesmo acontece com a informação: ela é o poder e está completamente esparramada por aí. Não é à tôa que muitas questões políticas foram incendiadas e até mesmo criadas com tudo isso, é só observar melhor a revolução árabe, as mobilizações sociais estudantis em toda américa e, sem precisar observar tanto assim, a resposta da poderosa massa virtual “anonyma” à famigerada PIPA que não voa e à SOPA que não é de comer.
Informação Biológica sem Fronteiras
A biologia sintética, depois de vários anos de gestação em centros de pesquisas por aí afora, nasce justamente como filha desse mundo informacional. É por isso “a cara” da mãe, e aproveita de uma maneira diferente os recursos de nosso admirável mundo novo (admirável mesmo!) em relação às ciências mais velhas, que se anabolizaram com a proliferação de bancos de dados online, colaborações sem fronteiras e acesso rápido e fácil à dados que antigamente você só conseguiria encontrar em missões desafiadoras em bibliotecas enormes pelo mundo. Em vez de ser apenas um ótimo recurso de desenvolvimento e pesquisa, o enorme fluxo de informações dos tempos atuais moldou a característica mais importante da biologia sintética que a torna inegavelmente original: a padronização, o que a diferencia de outras disciplinas como engenharia metabólica, genética, biologia de sistemas, molecular… e por aí vai. Com partes biológicas padronizadas, tanto a troca de informações genéticas em pesquisas como o redesign dessas informações tona-se muitíssimo mais rápido e fácil. A informação flui, torna-se mais útil, cria enormes possibilidades. Assim como a internet deu poder à nós, simples usuários, de sabermos de tudo (ou quase tudo) com apenas uns cliques, a biologia sintética facilitou a troca de informações de tal forma que qualquer um, tendo os equipamentos certos, pode criar “máquinas geneticamente modificadas” em seu quintal. Imagine então com centros de pesquisa!? O limite é a criatividade: basta juntar as partes certas do jeito certo e você tem uma nova aplicabilidade.
Quem mexeu na minha vida!?

"Growth Assembly Project": um esboço de como no futuro as indústrias poderão seus produtos pré-projetados no cultivo daquilo que hoje chamaríamos de "matéria prima" (clique na imagem para ir ao site do projeto).
Mas onde então os sinais da biologia sintética no nosso dia a dia!? Vai demorar um pouco para que todo o modo de produção de produtos industriais, até mesmo commodities, mude (veja imagem acima). A grande mídia vislumbrou com grande alarde pouco tempo atrás a bactéria “chassi perfeito” sintetizada em laboratório pelo grupo de Craig Venter, que seria capaz de ser transformada, de uma maneira super otimizada, em que você quiser, dispensando por exemplo as pesquisas que custam em fazer uma espécie de microrganismo servir adequadamente para produção de uma determinada coisa ou em se comportar de uma determinada maneira. Esse foi um grande avanço que contribuirá ainda mais no futuro quando a Mycoplasma Laboratorium for mais acessível. Não diria portanto que synbio é uma grande mudança do mundo em que vivemos agora, mas é pelo menos uma grande revolução premeditada. Quem sabe no futuro todos os grandes problemas da vida (fome, pobreza) sejam resolvidos com a própria vida, só que um pouco mais sintética.
“O que é Biologia Sintética?”: Reloaded
Como já foi postado aqui, “o que é biologia sintética?” é uma pergunta pouco difícil de definir. Synbio é uma área de intersecção entre Biologia de Sistemas, Engenharia Genética, Biofísica, Biocomputação, Engenharia Metabólica, Biologia Molecular e áreas relacionadas afins. Portanto, não é nada fácil dizer que “aquilo” ou “isto” é biologia sintética porque apontar para algo em Synbio é também apontar para outras áreas mais veteranas no mundo da ciência.
Mas então que diabos é isso tudo!? Uma grande “mistureba generalizada de todas as coisas”? Sim, mas não só isso.
Antes de falar do grande aspecto original que a Biologia Sintética tem – que é o detalhe “revolucionário” da coisa toda – deve-se fazer reconhecer algo que ela faz bem melhor do que a engenharia genética: o Design. O conceito de Design na engenharia de um novo sistema sintético é bem amplo, indo desde um design de proteínas (como proteínas quimera, como light switches), passando pelo próprio design do código genético (similar à escrita de um código de programação de computador mesmo), até ao design de todo o sistema biológico baseado na construção gênica feita (um exemplo bem visível disso é do projeto do iGEM com objetivos de criar uma bactéria resolvedora de Sudoku).
A reinvenção das capacidades do design de um sistema biológico foram possíveis não somente devido à novas tecnologias, mas sobretudo devido a uma nova abordagem com a cara dos nossos tempos super-informacionais: construção (biológicas, claro) automatizada e padronização das construções (os biobricks, por exemplo). Isso facilita enormemente o desenvolvimento de diversos dispositivos sintéticos e designs. Além disso, outra fundamentação da biologia sintética seria a abstração, a capacidade de abstrair problemas biológicos em uma visão mais simples, com base em toda a teoria já existente em outras áreas, como computação e a engenharia elétrica (não é à toa que Tom Knight e Randy Rettberg, dois engenheiros elétricos, fizeram parte da criação do iGEM).
Fica muito mais fácil entender tudo isso nas palavras do próprio Drew Andy – um dos pesquisadores que ajudou a fazer o parto da Biologia Sintética – nesse vídeo bem informal filmado em sua própria sala no MIT. Vale a pena conferir! 🙂
(OBS: é bem informal mesmo!)
Scinamate Synthetic Biology
Vídeo legal sobre SynBio
[youtube=http://www.youtube.com/watch?v=xOx3B2Z_qqE]
[twitter-follow screen_name=’MnlimasBio’]
O que é a vida!?
 Talvez essa seja a pergunta – ou pelo menos variantes dela – que todos os que têm coragem de se perguntar, se perguntam pelo menos uma vez na vida.
Talvez essa seja a pergunta – ou pelo menos variantes dela – que todos os que têm coragem de se perguntar, se perguntam pelo menos uma vez na vida.
Bem, o mínimo que podemos concluir desse metaquestionamento é que, se nos perguntamos isso, é porque estamos vivos e ponto final. O que seria uma das conclusões que você poderia tirar da frase que Descartes ficou convencidamente orgulhoso de ter elaborado: “Penso, logo existo!”. Mas esse é um site sobre biologia sintética, e como tal devemos tagarelar sobre como subjugar a Biologia ainda mais à nosso favor (com todo o respeito Biologia!), e não sobre essas coisas incertas de filósofos!
É aí que o pobre estudante, simpatizante, ou entusiasta do assunto se engana (i.e., leitor do blog). Toda a ciência, por mais complexa e rebuscada que se torne, chega a um ponto em que deve olhar para as suas entranhas e dizer: “Mas o que significa isso mesmo!?”.
Todas não, vai! A matemática já é bastante crescidinha para esse tipo de coisa. Já superou suas crises e faz tempo que vem descrevendo perfeitamente a realidade bem antes de a maioria das pessoas conseguir interpretar sua abstração como algo mais tangível.
A Física por exemplo, passou séculos podendo ser entendida sem maiores preocupações filosóficas, até chegar nos problemas quânticos. Nessa fase, uma pergunta puramente filosófica à olhos leigos – e também para um certo número considerável de não-leigos – poderia ser: “Como uma coisa pode e não pode estar no mesmo lugar ao mesmo tempo?” ou “Como um gato pode estar vivo e morto ao mesmo tempo dentro de uma caixa!?”. Mas aí a Matemática, com toda sua experiência, dá um tapinha nas costas da Física (a que muitos atribuem o parentesco de filha) e a ajuda em seus problemas existenciais, mesmo que muitos seres humanos não tenham a capacidade de compreender à primeira vista como ela ajudou. Mas isso é só um detalhe.
 E a Biologia!? Ela vem sofrendo de um problema existencial há muito tempo, e agora as coisas vêm piorando; coitada. Ela já estava combalida por causa de uma velha história indecisa sobre considerar um vírus como vida ou como “pseudovida”, e agora ainda vêm uns astrobiólogos aí com o papinho de “temos que acabar com os nossos preconceitos sobre o que é vida para conseguir procurar e encontrar adequadamente vida fora da Terra”. E que ainda por cima, têm a audácia de comprovar que o querido ácido nucleico, a peça chave na propagação de toda a vida, pode ser de uma maneira bem diferente (ok, nem tanto diferente) da que conhecíamos como “normal”. Isso sem falar nesses pesquisadores aqui (do post anterior) que conseguiram fazer em laboratório uma bactéria com um nucleotídeo considerado tóxico, que na prática, é quase o mesmo que fazer o que o pessoal da Nasa fez, mas sem o estardalhaço incompreendido na mídia (ou seria a mídia incompreendendo o estardalhaço?).
E a Biologia!? Ela vem sofrendo de um problema existencial há muito tempo, e agora as coisas vêm piorando; coitada. Ela já estava combalida por causa de uma velha história indecisa sobre considerar um vírus como vida ou como “pseudovida”, e agora ainda vêm uns astrobiólogos aí com o papinho de “temos que acabar com os nossos preconceitos sobre o que é vida para conseguir procurar e encontrar adequadamente vida fora da Terra”. E que ainda por cima, têm a audácia de comprovar que o querido ácido nucleico, a peça chave na propagação de toda a vida, pode ser de uma maneira bem diferente (ok, nem tanto diferente) da que conhecíamos como “normal”. Isso sem falar nesses pesquisadores aqui (do post anterior) que conseguiram fazer em laboratório uma bactéria com um nucleotídeo considerado tóxico, que na prática, é quase o mesmo que fazer o que o pessoal da Nasa fez, mas sem o estardalhaço incompreendido na mídia (ou seria a mídia incompreendendo o estardalhaço?).
“Mas o que eu tenho a ver com essas neuras das ciências!?”, poderia se perguntar a pessoa mais desavisada. Eu diria que tudo.
As sociedades mais predominantes no mundo, apesar de terem o pensamento científico seguindo uma lógica singular, têm uma cultura extremamente plural. Fato. Isso quer dizer então, à priori, que existem mais maneiras diferentes dessas culturas verem a ciência do que o contrário; e as maneiras de influenciar a ciência são bem maiores quando ela abre margem para discussão de seus conceitos incertos, como “o que é vida!?”.
Aos olhos de uns, as peripécias do grupo de Craig Venter (que criaram a “célula artificial” Mycoplasma laboratorium) podem ser “brincar de Deus”, aos olhos de outros pode ser apenas “a linhagem de bactéria geneticamente mais ‘enxuta’ que já se conseguiu criar”. É nesse modelamento extremo da vida que a opinião sobre “o que é a vida!?” faz muito a diferença. Em geral essa pluralidade de visões é benéfica, pois ajuda a regularizar muita coisa potencialmente perigosa e não-ética. Mas por causa da falta de informação, pode gerar muito preconceito e dificultar várias pesquisas que poderia ajudar muita gente. Vide por exemplo o uso das células tronco: qual é sua opinião? Agora você consegue ver a implicação na sua vida do título desse post!?
nesse modelamento extremo da vida que a opinião sobre “o que é a vida!?” faz muito a diferença. Em geral essa pluralidade de visões é benéfica, pois ajuda a regularizar muita coisa potencialmente perigosa e não-ética. Mas por causa da falta de informação, pode gerar muito preconceito e dificultar várias pesquisas que poderia ajudar muita gente. Vide por exemplo o uso das células tronco: qual é sua opinião? Agora você consegue ver a implicação na sua vida do título desse post!?
Parece que a Biologia vai ter que ficar filosofando ainda um bom tempo sobre suas entranhas para responder algumas perguntas, mas isso talvez seja só um lembrete da sábia mãe Filosofia: de que pragmatismo em excesso e falta de questionamento geralmente não caem bem para o estômago das ciências.
O que é Biologia Sintética?
 Este post deveria ser um dos primeiros artigos para um site que se diz especializado em biologia sintética. Mas confesso para vocês que definir o que é biologia sintética, para mim, não foi (é) uma tarefa fácil. Como podem ser vistos nos posts no blog, existem vários aspectos da synbio que permitem diferentes definições de acordo com o ponto de vista de quem está fazendo biologia sintética. Por exemplo, um engenheiro interessado em criar dispositivos computacionais sintéticos em uma bactéria, ou um biotecnólogo interessado em produzir biocombustíveis ou um biólogo querendo montar uma bactéria a partir de simples elementos químicos. Claramente, todas estas áreas estão conectadas, mas criar uma definição que consiga embarcar todas as possibilidades não é trivial. Por isso, digo que este post é orgânico, que deve mudar à medida que o meu conhecimento sobre o assunto se aprofunda.
Este post deveria ser um dos primeiros artigos para um site que se diz especializado em biologia sintética. Mas confesso para vocês que definir o que é biologia sintética, para mim, não foi (é) uma tarefa fácil. Como podem ser vistos nos posts no blog, existem vários aspectos da synbio que permitem diferentes definições de acordo com o ponto de vista de quem está fazendo biologia sintética. Por exemplo, um engenheiro interessado em criar dispositivos computacionais sintéticos em uma bactéria, ou um biotecnólogo interessado em produzir biocombustíveis ou um biólogo querendo montar uma bactéria a partir de simples elementos químicos. Claramente, todas estas áreas estão conectadas, mas criar uma definição que consiga embarcar todas as possibilidades não é trivial. Por isso, digo que este post é orgânico, que deve mudar à medida que o meu conhecimento sobre o assunto se aprofunda.
Uma característica que une todos os biológos sintéticos é a vontade de tornar o processo de engenharia de sistemas biológicos mais fácil e confiável. Dentro desse contexto, existem quatro diferentes níveis de atuação da biologia sintética:
(i) partes biológicas: sendo o DNA a linguagem de programação, as partes biológicas são uma sequência de dados (AGCTA…) que possuem funções determinadas. Por exemplo, uma sequência de DNA que faz a célula mudar a cor de verde para amarelo. Estas partes são descritas, catalogadas e respeitam determinado padrão físico de montagem (leia mais sobre os biobricks). Espera-se que com o tempo se possam descrever as características de inúmeras partes biológicas para serem utilizadas para a construção de dispositivos, sistemas,…. Essa é uma das funções da Secretária de Partes Biológicas Padrão do MIT.
(ii) dispositivos sintéticos: são compostos por partes biológicas capazes de processar sinais. Processam inputs em outputs. Para a construção de dispositivos robustos e eficientes são necessárias partes que funcionem de uma maneira previsível. Veja mais sobre dispositivos sintéticos.
(iii) sistemas sintéticos: são um conjunto dispositivos capazes de captar sinais, processar informações e realizar funções determinadas, como por exemplo, uma célula capaz de captar algum sinal do ambiente e decidir se irá realizar uma determinada função como combater uma célula tumoral, produzir determinado metabólito etc.
(iv) por útimo, existe a arquitetura sintética de populações em que, por ex, cada microrganismo possui um dispositivo diferente, sendo necessário que estes dispositivos trabalhem em conjunto para realizar determinada função. Trabalhar em conjunto, como uma população que precisa trabalhar com sincronismo, é o caso dos osciladores. Para isso, é necessário dominar mecanismos robustos de comunicação célula-célula.
A biologia sintética pode ser aplicada em praticamente todas áreas da biologia molecular, biotecnologia e engenharia genética. Porém existem algumas áreas que se destacam como sendo próprias da biologia sintética:
1. Construção de uma célula mínima: identificação das partes básicas para construção de uma célula.
2. Reconstrução de células: tendo como objetivo central a construção de formas de vida artificiais a partir de elementos químicos.
3. Construção de novos códigos genéticos.
5. Construção de células capazes de realizar funções diferentes daquelas encontradas na natureza, como a obtenção de novas rotas bioquímicas de produção de novos compostos. .
Essa última, talvez seja a que mais tenha impacto nas nossas vidas cotidianas a curto-prazo, através do desenvolvimento de remédios mais baratos e com a produção de combustíveis e químicos utilizando recursos renováveis.
















