Bactérias na guerra contra o câncer
 Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Atualmente, desvendar o sistema de comunicação utilizado por células cancerígenas tem sido foco de inúmeras pesquisas. Como em qualquer guerra moderna, destruir o sistema de comunicação inimigo pode causar grandes danos. E na guerra contra o câncer provavelmente não será diferente. Impedir as células de se comunicarem pode evitar que elas adotem estratégias inteligentes tais como ficarem dormentes durante quimioterapia ou até mesmo matar células irmãs para obtenção de alimento. Uma vez entendido a linguagem utilizada por estas células, podemos utilizar isto ao nosso favor, interferindo nas mensagens e fazendo com que células dormentes sejam acordadas durante a quimioterapia ou até mesmo induzir as células cancerígenas a matarem umas as outras.
Finalmente, uma possível estratégia futura seria recrutar bactérias para derrotar o câncer. Elas poderiam ser utilizadas para “ensinar” as células do sistema imunológico a reconhecer e matar as células cancerígenas. É importante ressaltar que ainda compreendemos muito pouco sobre os mecanismos envolvidos no câncer e novas abordagens são necessárias para superá-lo. Entretanto, quem sabe num futuro próximo, estaremos em uma era de guerra cibernética biológica onde bactérias serão inteligentemente projetadas para derrotar o câncer.
Referências:
“Bacterial survival strategies suggest rethinking cancer cooperativity” Eshel Ben-Jacob, Donald S. Coffey, Herbert Levine. Trends in Microbiology. 2012.
“Bacterial linguistic communication and social intelligence” Eshel Ben-Jacob, Israela Becker, Yoash Shapira, Herbert Levine. Trends in Microbiology. 2004
Transposons: pedaços de DNA que mudam de endereço no genoma.
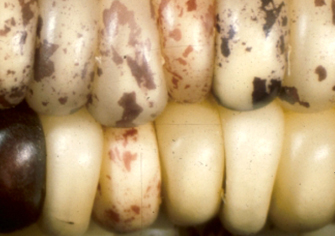
Mudança no padrão das cores do milho devido a interação entre os genes responsáveis pelo pigmento e elementos transponíveis.
Você sabia que alguns trechos de DNA têm a capacidade de mover-se no genoma, saindo de um cromossomo e se inserindo em outro? Pois é, e esta descoberta foi feita há muito tempo atrás por uma brilhante cientista, antes mesmo da descoberta da estrutura do DNA por Watson e Crick. Barbara McClintock ao observar a relação entre os padrões das cores do milho e algumas quebras cromossomais, percebeu que algumas destas quebras ocorriam com uma freqüência muito mais alta do que o esperado e, surpreendente, sempre no mesmo cromossomo. Após uma série de experimentos, a cientista percebeu que esta quebra ocorria devido a inserção de pedaços de DNA vindos de outros cromossomos que alteravam a expressão dos genes de pigmento. Confiante nos seus experimentos, Barbara então propôs que havia elementos transponíveis no genoma, ou seja, os genes não tinham um endereço fixo, mas tinham um mecanismo para movimentar-se dentro do genoma.
Se nos dias de hoje esta ideia parece bastante surpreendente, imagine como foi a recepção desta ousada ideia no começo da década de 50, quando a cientista começou a publicar seus resultados. Eles foram ignorados e ridicularizados pelos seus contemporâneos, levando a cientista a parar de publicar suas descobertas sobre o tema. Somente no início da década de 70 alguns elementos transponíveis foram identificados em bactéria validando assim a teoria de Barbara. Entretanto, o reconhecimento completo de uma das mais importantes descobertas da biologia somente aconteceu no início da década de 80, quando a cientista foi homenageada com o prêmio Nobel.
Atualmente vários estudos já relacionaram os elementos transponíveis, chamados de transposons, a uma série de doenças e como grande fonte de variabilidade genética. Hoje sabe-se que boa parte do genoma é composto destes elementos e dois mecanismos principais já foram identificados. O primeiro deles é um mecanismo de recortar-e-colar, onde alguns pedaços do DNA simplesmente saem de um lugar e movem-se para outra parte do genoma. O outro mecanismo é do tipo cortar-e-colar, gerando mais de uma cópia no genoma. Este último mecanismo é chamado de retrotransposons e consiste em quase metade do genoma humano e será tema de um próximo post.
FAQ #8 Como ver se a transformação gênica deu certo?
Já colocamos nossos plasmídeos nos bichinhos, mas ainda não acabou. Essa é a hora de saber se tudo que fizemos até aqui deu certo!
Primeiro, colocamos eles na placa de seleção com antibiótico e só aqueles que realmente incorporaram o plasmídeo vão sobreviver porque ganharam um gene amigo de resistência ao antibiótico. Agora pegamos uma parte das células sobreviventes e fazemos um teste usando a PCR (você lembra dessa técnica, né?).
Como testar com a PCR?! Roubamos os plasmídeos dessas células e tentamos multiplicar o pedaço de DNA que foi grudado nele adicionando primers, nucleotídeos e enzimas sob aquecimento e desaquecimento. Se o nosso plasmídeo modificado tiver sido incorporado pelos micro-organismos haverá a multiplicação do pedacinho e podemos vê-los em uma eletroforese. Caso contrário, nada foi copiado e nada vai aparecer na eletroforese. Então temos o sinal de que alguma coisa não deu certo por aqui e as chances de erros deste teste são bem pequenas.
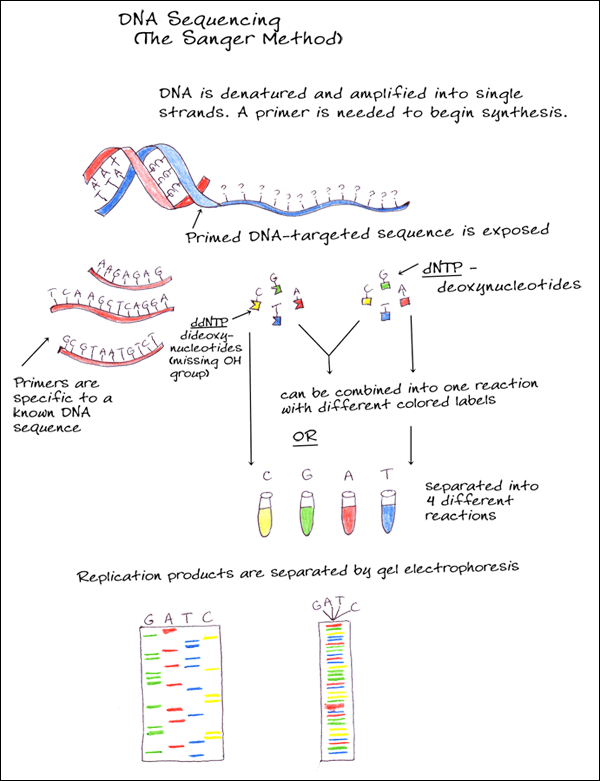
Depois vamos para um teste melhor ainda, o sequenciamento. Este teste diz exatamente qual é a sequência de pares de base da molécula do plasmídeo e acabam de vez suas dúvidas se a transformação deu certo ou não! O sequenciamento começa parecendo um processo de duplicação normal de DNA. Mas além de cadeias molde, enzimas e nucleotídeos normais, há nucleotídeos especiais sintetizados (ddNTP’s) que possuem duas boas propriedades: emitem luz e interrompem o prologamento da cadeia a partir de onde foram adicionados. Então, imagine uma molécula de DNA que possui um par de base A-T em um determinado comprimento e considere que a base A pertence à cadeia molde e a base T à cadeia complementar. Se esta base T for um nucleotídeo especial temos como identificar “quem é” e “qual sua posição” na cadeia, pois ele emitirá uma cor específica para Timina e o comprimento de sua cadeia está interrompido na posição exata (nº 2 em verde). 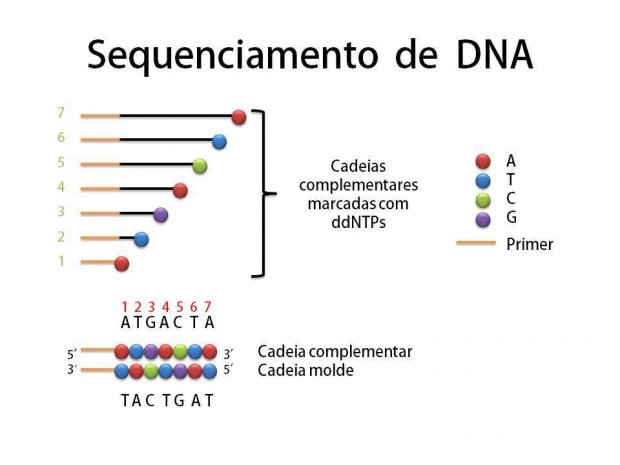 Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Para saber mais sobre sequenciamento genético, clique aqui.
Por Otto Heringer e Viviane Siratuti.