Os verdadeiros revolucionários
Ninguém duvida que Apple, Tesla e Google sejam sinônimos de inovação e tecnologia. E que seu sucesso está diretamente ligado a mentes brilhantes como Steve Jobs e Elon Musk, visionários de primeira hora. Mas os investimentos necessários para desenvolver a tecnologia que está presente num iPhone ou num Tesla com certeza espantaria a maior parte dos investidores do mercado, impacientes e ávidos por lucros no espaço de tempo mais curto possível.
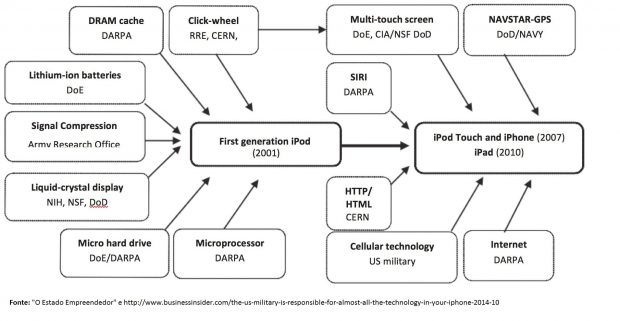
Pouca gente sabe que foi o dinheiro do contribuinte americano ao longo de décadas que ajudou estas empresas a serem o sucesso que são e forneceu as bases para o surgimento dos produtos revolucionários de algumas das empresas mais inovadoras do mundo. Essa é a ideia defendida pela economista Mariana Mazzucato no seu livro “O Estado Empreendedor”.
Apesar das críticas, às vezes merecidas, de serem estruturas pesadas e burocráticas, as agências do governo americano financiaram pesquisas que trouxeram para a indústria de consumo a tela touch screen, o display de cristal liquido, o SIRI e ajudaram Steve Jobs a fazer da sua empresa a marca icônica que ela é.
Mas não são apenas as agência de defesa que produzem inovações. A partir de 1983 as empresas de biotecnologia se beneficiaram de um belo empurrão dado pelo Governo. Naquele ano foi aprovado nos Estados Unidos o Orphan Drug Act, decreto que fornece incentivos fiscais e subsídios de P&D para o desenvolvimento de medicamentos para o tratamento de doenças raras (doenças que afetam menos de 200 000 pessoas). Sem este apoio eles praticamente não existiriam. Esta iniciativa foi fundamental para o crescimento de empresas como Amgen, Genentech e Genzyme. Hoje os medicamentos para doenças raras são responsáveis por mais de 70% da receita das principais empresas de biotecnologia.
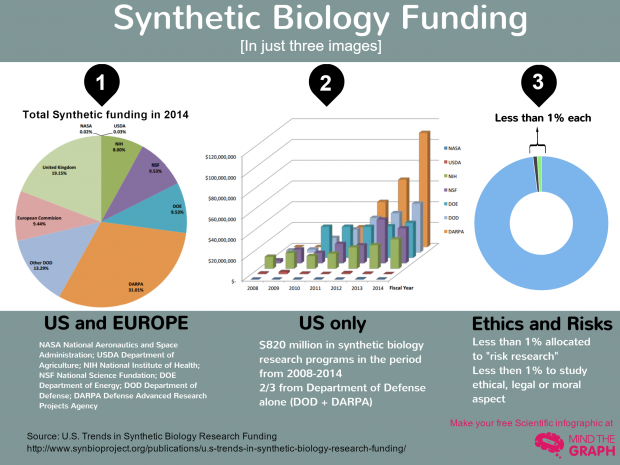
Os produtos inovadores dependem fundamentalmente dos investimentos em pesquisa de base. Se quisermos saber o que o futuro nos reserva, temos que olhar o que está sendo feito hoje neste campo, um exemplo é a biologia sintética. Entre 2008 e 2014 as agências governamentais americanas já investiram quase 1 bilhão de dólares em pesquisas nessa área. O MIT-Broad Foundry é um dos institutos apoiados com recursos da agência de defesa DARPA e, de acordo com Ben Gordon, diretor do Foundry, ele tem o objetivo de trazer soluções para a saúde, a agricultura e a química que são desafiadoras demais para a indústria e para a academia.
Aqui no Brasil, além da pesquisa básica, o Governo tem investido em pesquisas na indústria e na criação de empresas de base tecnológica como forma de aproveitar as pesquisas, as patentes e o conhecimento produzido nas universidades. Entre as agências de fomento está a Financiadora de Estudos e Projetos (FINEP), que por meio do Plano Inova Empresa apoia a inovação em setores que o Governo Federal considera estratégicos. Lançado em 2013, é o projeto mais ambicioso de inovação que o país já teve. Uma verdadeira revolução. Ele conta com R$ 32,9 bilhões que serão destinados a áreas como energia, nanotecnologia e biotecnologia nos próximos anos.
Uma das instituições que receberam recursos do FINEP foi a Embrapa Agroenergia. Em um prédio com 10.000 m2 no Distrito Federal, a instituição trabalha para produzir um futuro sustentável para o planeta e para a economia brasileira. Mais de 50 projetos estão em desenvolvimento, entre eles a pesquisa em processos de conversão da biomassa em bioenergia, biomateriais e químicos renováveis de alto valor agregado. Mas nem sempre os recursos públicos são bem aplicados. O programa Ciência sem Fronteiras é um desses casos. Com mais de 100.000 bolsas, a maior parte delas para alunos de graduação, o programa investe na formação de pessoal altamente qualificado, colocando estudantes e pesquisadores em instituições de excelência no exterior e atraindo jovens talentos para trabalhar no Brasil. Apesar de proporcionar a vivência internacional aos alunos, a iniciativa falha pela falta de um acompanhamento rigoroso das disciplinas e atividades desenvolvidas por eles no exterior. Isso nunca poderia acontecer num programa onde o custo de um aluno pode chegar a 80.000 reais por ano. Rogerio Meneghini, professor da USP e diretor científico da biblioteca virtual SciELO afirma: “No contexto do desenvolvimento da ciência, a ida desses estudantes de graduação ao exterior não vai fazer qualquer diferença”.
Num momento de dificuldades econômicas como a que enfrentamos hoje e viveremos nos próximos anos, é vital utilizar com mais sabedoria os recursos públicos. Gerenciar e avaliar os resultados ficou mais importante do que nunca. Ter a parceira das empresas privadas é essencial. Resumindo: é fundamental não perdermos essa revolução que o Governo começou.
Biotecnologia sem fronteiras: o monopólio da inovação está com seus dias contados
O baixo custo dos computadores, a lei de Moore ainda em vigor e o acesso à internet democratizaram os meios de produção, distribuição e educação. Hoje não somos apenas consumidores passivos, mas também produtores ativos. E na ciência, não é diferente. Vivemos a era “Pro-Am”, em que amadores dedicados, inovadores e conectados trabalham como profissionais, uma realidade na astronomia, na ciência da computação, e agora da biotecnologia. Basta um computador, conexão com a internet e um cartão de crédito para encomendar DNA e testar algumas das mais novas técnicas de clonagem e edição de genoma, até então só acessível a universidades e grandes empresas.
Em 1987, a luz de uma estrela, que explodiu há 168.000 anos chegou a Terra. Foi então que astronômos  amadores junto com profissionais, confirmaram a teoria do que ocorre quando uma estrela explode, uma das maiores descobertas da astronomia do século XX. Hoje muito mais gente participa da ciência que até então dependia de equipamentos sofisticados e caros. Uma dessas tecnologias, foi disponibilizada por John Dobson, responsável por criar um poderoso telescópio usando materias de segunda-mão. Dobson se recusou a lucrar com sua invenção, a qual nunca patenteou. Essa democratização chegou à biologia com o movimento Do-it-yourself Biology (DIYbio) em 2008, em que profissionais e amadores desenvolvem projetos em laboratórios comunitários, constroem equipamentos por uma fração do preço e aproximam a comunidade da discussão sobre organismos geneticamente modificados.
amadores junto com profissionais, confirmaram a teoria do que ocorre quando uma estrela explode, uma das maiores descobertas da astronomia do século XX. Hoje muito mais gente participa da ciência que até então dependia de equipamentos sofisticados e caros. Uma dessas tecnologias, foi disponibilizada por John Dobson, responsável por criar um poderoso telescópio usando materias de segunda-mão. Dobson se recusou a lucrar com sua invenção, a qual nunca patenteou. Essa democratização chegou à biologia com o movimento Do-it-yourself Biology (DIYbio) em 2008, em que profissionais e amadores desenvolvem projetos em laboratórios comunitários, constroem equipamentos por uma fração do preço e aproximam a comunidade da discussão sobre organismos geneticamente modificados.
Em um desses laboratórios comunitários, o BioCurious, na Califórnia, são desenvolvidos projetos que envolvem a recente ferramenta de edição de genoma CRISPR, uma bioimpressora capaz de “imprimir” células de E. coli, e um projeto do iGEM para a produção de queijo “vegan”, com proteínas do leite produzidas por leveduras, e não vacas.
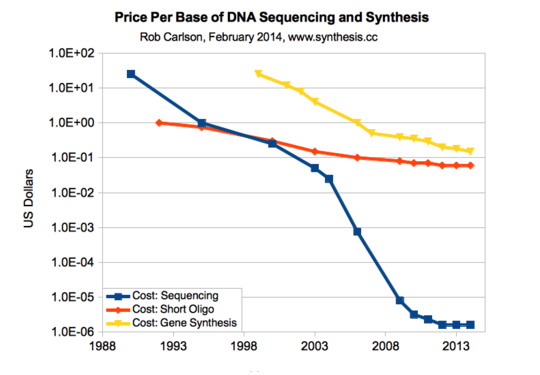
Atualmente é possível comprar ou montar os próprios equipamentos, como o OpenPCR, um termociclador usado para amplificar DNA, a centrífuga OpenFuge e o robô OpenTrons, que permite automatizar seu trabalho de bancada. Em breve, o MiniION™ será realidade, um sequenciador portátil e descartável que pode analisar sua amostra, seja ela de um microrganismo ou sangue, em qualquer lugar do mundo, sem precisar de milhares de doláres em equipamentos ou treinamento. Além de aparelhos mais acessíveis, o preço de síntese por base de DNA (A, T, C, G) já custa apenas alguns centavos e tem caido ano após ano. No futuro, nada impede que etapas custosas de um projeto sejam terceirizadas, ocupando a capacidade ociosa de laboratórios ou serem realizadas por empresas prestadoras de serviços, como já acontece com a síntese e o sequenciamento de DNA na China.
Projetos como esses vão reduzir o custo de se buscar o novo, trazendo soluções acessíveis para a saúde, alimentação e preservação do meio ambiente, quebrando o atual monopólio da inovação presente apenas nas grandes instituições como empresas e universidades. Como em toda abertura democrática, espera-se que muito mais gente se beneficie deste passo da ciência: a biotecnologia sem fronteiras.
Confira a iniciativa acontecendo em Sâo Paulo – http://synbiobrasil.org/st/diy/
Referências:
The Pro-Am Revolution – How enthusiasts are changing our economy and society – Charles Leadbeater and Paul Miller
Time for new DNA synthesis and sequencing cost curves – Rob Carlson
CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
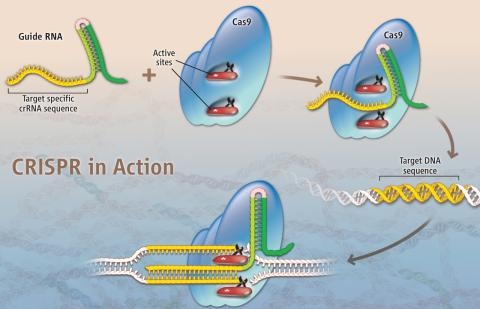 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência:
Bactérias na guerra contra o câncer
 Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Atualmente, desvendar o sistema de comunicação utilizado por células cancerígenas tem sido foco de inúmeras pesquisas. Como em qualquer guerra moderna, destruir o sistema de comunicação inimigo pode causar grandes danos. E na guerra contra o câncer provavelmente não será diferente. Impedir as células de se comunicarem pode evitar que elas adotem estratégias inteligentes tais como ficarem dormentes durante quimioterapia ou até mesmo matar células irmãs para obtenção de alimento. Uma vez entendido a linguagem utilizada por estas células, podemos utilizar isto ao nosso favor, interferindo nas mensagens e fazendo com que células dormentes sejam acordadas durante a quimioterapia ou até mesmo induzir as células cancerígenas a matarem umas as outras.
Finalmente, uma possível estratégia futura seria recrutar bactérias para derrotar o câncer. Elas poderiam ser utilizadas para “ensinar” as células do sistema imunológico a reconhecer e matar as células cancerígenas. É importante ressaltar que ainda compreendemos muito pouco sobre os mecanismos envolvidos no câncer e novas abordagens são necessárias para superá-lo. Entretanto, quem sabe num futuro próximo, estaremos em uma era de guerra cibernética biológica onde bactérias serão inteligentemente projetadas para derrotar o câncer.
Referências:
“Bacterial survival strategies suggest rethinking cancer cooperativity” Eshel Ben-Jacob, Donald S. Coffey, Herbert Levine. Trends in Microbiology. 2012.
“Bacterial linguistic communication and social intelligence” Eshel Ben-Jacob, Israela Becker, Yoash Shapira, Herbert Levine. Trends in Microbiology. 2004
Jamboré Brasil!
Quem diria. A um ano atrás estávamos nós fazendo vaquinha virtual pra levar o Brasil para a competição internacional de máquinas geneticamente modificadas e hoje, ainda na luta, podemos compartilhar o fardo herdado da Unicamp de representar a ciência tupiniquim no iGEM. Que lindo isso.
Mais lindo ainda é que as equipes de Manaus, Belo Horizonte e São Paulo são amiguinhas! Numa das competições mais bizarras do mundo (o iGEM) o conceito de competição também é “distorcido”: ganha mais quem colabora mais – o “distorcido” deveria ser exatamente o contrário na ciência mundial hoje em dia, mas deixa pra lá! E é por isso que nós vamos nos reunir no primeiro encontro nacional de equipes do iGEM: para trocar experiências, fazer networking, se conhecer melhor e conversar bastante sobre coisas nerds, como Biologia Sintética, é claro. Afinal, a gente faz o que a gente ama, não é mesmo!?
Enfim! Nós das equipes do Brasil, que estamos aqui na raça, na gana, na teimosia pra fazer um Brasil e, “de tabela”, um mundo melhor, abrimos esse encontro de jovens interdisciplinares e amantes de biotecnologia para todo mundo! Sim, aqui na USP, em São Paulo! É o “Jamboré”! Porque Jamboree é “nas gringa” [fora do país], aqui é Jamboré!
Local e Data
Tudo vai acontecer neste sábado, dia 17 de Agosto, no Instituto de Química, no famigerado “Queijinho” (ou, Complexo Ana Rosa Kucinski, como foi rebatizado recentemente), sala A2. Veja o mapa aqui:
Visualizar Jamboré! em um mapa maior
Cronograma
As atividades vão ser de manhã e a tarde. Atividades infinitas!
| Horário | Atividade |
| 10H – 10:30H | Abertura: “Biologia Sintética, iGEM e Brasil” |
| 10:30 – 12:00 | Apresentação dos projetos brasileiros no iGEM 2013 |
| 12H – 14H | Almoço |
| 14H – 15:30H | Play-teste de Jogo de Cartas sobre Biologia Sintética |
| 15:30H – 15:50H | Coffee-Break |
| 15:50H – 17H | Mesa Redonda sobre a formação das equipes do iGEM no Brasil |
Pessoas de todas as áreas são bem vindas. Aqui interdisciplinariedade (e discussões estranhas) são nossa especialidade. Não esperamos que você saiba nada de Biologia Molecular ou modelagem matemática, para qualquer dúvida nós vamos estar ali para ajudar. Ou a piorar. Depende do ponto de vista.
O evento é aberto a todos fora e dentro da comunidade USP. Então se quiser um programa nerd de qualidade esse final de semana, venha para a Cidade Universitária!
Sequenciamento em Marte
Tem gente que jura já ter visto discos voadores e até ter entrado em contato com extraterrestres. Verdade ou não, esse é um assunto que desperta curiosidade em muitos e medo em tantos outros. Ao contrário do imaginário popular, é provável que os seres extraterrestres já tenham chegado na Terra há 4 bilhões de anos e que nós evoluimos a partir deles. É isso o que sugere uma teoria conhecida como panspermia.
Talvez não seja mais necessário esperar por alienígenas visitarem nosso planeta ou uma longa discussão sobre a veracidade dos organismos fossilizados encontrados em meteoritos para confirmar a existência de ETs. Em breve teremos acesso ao código genético de organismos marcianos, de acordo com a proposta de dois cientistas, Craig Venter e Jonathan Rothberg. Ambos estão em uma corrida, embora não declarada oficialmente, para sequenciar o DNA de possíveis formas de vida que existam ou existiram em Marte. Por isso, eles querem uma carona até o planeta vermelho para sequenciar possíveis formas de vida que possam encontrar por lá.
 A carona não é para eles, mas para o sequenciador que pretendem enviar. Venter está confiante que
A carona não é para eles, mas para o sequenciador que pretendem enviar. Venter está confiante que
encontrará formas de vida que contenham DNA em Marte, como afirmou em uma conferência da Wired Health no ano passado, em Nova York. Sua equipe está desenvolvendo e testando o que ele chama de “teletransporte biológico”. Um robô que será enviado para Marte capaz sequenciar o DNA encontrado no local, mesmo que seja de uma única célula, e transmitir a sequência do organismo extraterrestre para um computador aqui na Terra. De acordo com ele, testes já estão sendo feitos no deserto de Mojave, onde cientistas simulam as condições de exploração do espaço. Com o DNA digitalizado será possível sintetizá-lo, injetá-lo em uma célula universal receptora e dar vida a um ET. A entrevista completa sobre esse assunto pode ser assistida aqui (11:00).
Correndo em outra frente está Jonathan Rothberg, fundador da Ion Torrent, em um projeto financiado pela NASA chamado SET-G (The Search for Extra-terrestrial Genomes) também pretende sequenciar DNA no planeta vermelho. Para isso será necessário reduzir o tamanho de seu sequenciador de grande sucesso, o Ion Personal Genome Machine, de trinta para apenas três quilos, viabilizando a viagem de milhões de quilômetros.
Mas por que não trazer uma amostra de Marte? Tessi Kanavarioti, químico envolvido no estudo de pedras que vieram da lua na década de 70, garante: “Devido a possibilidade de contaminação, ninguém iria acreditar em você”. Foi o que aconteceu em 1971, quando astronautas da Apollo 12 trouxeram uma câmera de TV que ficou três anos na lua. Nela foi encontrada uma única bactéria Streptococcus mitis. Muitos disseram se tratar de uma contaminação, embora fosse apenas uma única bactéria. O micro-organismo estava dormente e ganhou vida novamente na Terra. Além da contaminação, o sequenciamento em Marte reduziria o tempo para obter essa amostra, caso ela fosse enviada para ser analisada por aqui.
Mas este é um projeto de alto risco. As moléculas de DNA possuem uma meia-vida de 521 anos (tempo que leva para metade das ligações fosfodiéster se romperem), portanto temos que acreditar que exista vida agora em Marte, ou organismos mortos há menos de 1,5 milhões de anos, caso contrário os fragmentos encontrados seriam muito pequenos e não trariam informações úteis. Além disso, nada garante que as formas de vida que possam existir por lá tenham os mesmos componentes do nosso DNA.
O espaço parece um local improvável de abrigar formas de vida como conhecemos, seja por conta da baixa temperatura, pouco oxigênio ou elevada radiação. Mesmo sem foguetes podemos encontrar organismos que vivem em condições que não consideramos favoráveis. Esses organismos são os extremófilos, ou seja, eles adoram condições extremas de pH, salinidade, temperatura ou radiação. Um caso interessante é a bactéria Deinococcus radiodurans, encontrada vivendo dentro de reatores nucleares. É provável que esses sejam os tipos de organismos que possamos encontrar em outros planetas.
 Além de micro-organismos que conseguem sobreviver em condições que consideramos extremas, novas
Além de micro-organismos que conseguem sobreviver em condições que consideramos extremas, novas
evidências obtidas pelo robô Curiosity da NASA sugerem que o planeta já foi habitável. A análise de uma rocha sedimentar mostrou a presença de enxofre, nitrogênio, hidrogênio, oxigênio, fósforo e carbono, o que aumenta as chances de um possível sequenciamento dar certo.
Caso seja encontrado DNA, teremos mais evidências para sustentar a hipótese de que a vida não teve origem aqui na Terra e que ela pode ter evoluído de forma diferente em outros planetas. Uma próxima viagem para Marte está planejada pela NASA para 2018, mas nem Venter nem Rothberg tem lugar garantido ainda. O que torna a corrida ainda mais emocionante.
Referências:
- The Biological Big Bang: Panspermia and Origins of Life. Edited by Chandra Wickramasinghe, Ph.D.
- http://www.nature.com/news/dna-has-a-521-year-half-life-1.11555
- http://www.jpl.nasa.gov/news/news.php?release=2013-092
- http://www.technologyreview.com/news/429662/genome-hunters-go-after-martian-dna/
Experiência em Biologia Sintética – Monique Gasparoto
Entrevista feita por Mira Melke.
A Biologia Sintética é extremamente motivadora. Para provar isso e para mostrar o quão importante e distinta pode ser uma experiência em Biologia Sintética acima do equador convidei uma amiga, companheira dos tempo de Biomol (Ciências Físicas e Biomoleculares) para escrever um pouquinho para a gente.
Quem fala agora é a Monique:
Biologia sintética: impossível não se apaixonar!
Minha história com a Biologia Sintética começou como toda história de amor, umas paquerinhas para cá, um google search para lá, mas nada muito sério. A primeira vez que ouvi falar da área foi em 2009, quando nem havia descrições em português. O amor adormeceu enquanto eu me desdobrava para ser aprovada em todas as disciplinas do curso de Ciências Físicas e Biomoleculares da USP de São Carlos, do qual atualmente sou aluna do último ano. Envolvi-me em outra área de pesquisa, o mundo continuou a andar, mas quando eu menos esperava fui me reencontrar com minha paixonite dos tempos de caloura.
Como bolsista do programa Ciências sem Fronteiras, passei um ano na Boston University e além da incrível experiência de intercâmbio, tive a oportunidade de trabalhar no laboratório do professor Doug Densmore (CIDAR) e fazer parte do time do iGEM da Boston University. Eu não poderia sonhar em um lugar mais incrível para me aproximar da Synbio: estar em Boston onde as primeiras bases da área foram lançadas, fazer pesquisa em um laboratório exclusivamente de Biologia Sintética – em que todo mundo tem o site do Registry nos favoritos(!), assistir a palestras e seminários dos pesquisadores referência da área, como o Jim Collins, com quem dividíamos espaço de laboratório , visitar o Headquarters do iGEM e muitos outros aspectos me fizeram ter a certeza de que a Synbio veio para ficar não só na minha vida, mas certamente na de todos os que a conhecem.

O projeto que desenvolvemos para a competição trabalhava os três pilares do iGEM: construção, caracterização e compartilhamento das informações do Registry. Para isso introduzimos na competição o método de Clonagem Modular (MoClo) descrita por Weber et al, propusemos um protocolo de caracterização padrão para circuitos com proteínas fluorescentes usando citometria de fluxo e esboçamos uma página comum a ser usada no Registry em que as informação sobre as partes poderiam ser geradas automaticamente a partir do Clotho, uma plataforma para Biologia Sintética desenvolvida pelo meu orientador, Doug Densmore. Mais detalhes vocês podem conferir na nossa Wiki.
Foi um período de aprendizado intenso, porque era a primeira vez que o Densmore Lab apoiava um time de WetLab, a tradição dos anos anteriores era o time de software. Éramos dois alunos de graduação orientados por três alunos de doutorado e uma pós-doc, e nunca imaginei participar de um ambiente tão colaborativo e estimulante. É claro que parte disso é devido à excelente estrutura do laboratório e às facilidades dos meios de pesquisa, quem não ficaria feliz e contente com sequenciamentos de DNA que ficam prontos no mesmo dia e enzimas que chegam ao laboratório em no máximo 48h após a encomenda!? Mas o diferencial dessa experiência veio da oportunidade de vivenciar um ambiente de apaixonados por Biologia Sintética e perceber como eles desenvolvem suas pesquisas: com muita competência, muito estudo e muita motivação!
O que mais me cativa nessa área da ciência que agrega à biologia molecular conceitos e ferramentas da engenharia é que tão importante quanto o conhecimento técnico, é a inovação e a criatividade. Características que eu pude testemunhar de perto em todos aqueles que participaram do iGEM, e que ficaram ainda mais nítidas quando na fase final da competição em Boston, times do mundo inteiro, desde do Leste Asiático até a América do Sul se reuniram para sonhar, discutir e compartilhar suas propostas para tornar o mundo melhor, “one part at a time”.
Talvez não haja outro grupo de (malucos) cientistas que acredite tanto que seus projetos e conhecimentos podem mudar o mundo. Aí está o brilho da Synbio, que uniu pesquisadores de fronteira que não queriam mais ficar confinados às suas especialidades, mas decidiram sair de sua zona de conforto e ousar e empreender em grupos multidisciplinares. A ousadia desses biólogos sintéticos é tão grande que são capazes de investir cifrões de patrocínio e meses de trabalho em uma competição em que o grande prêmio, aos olhos dos mais céticos, é somente um BioBrick gigante. É como dizem por aí, a biologia sintética tem razões que a própria razão desconhece.
Microalgas na Biologia Sintética
![]() Na penúltima reunião do Clube de Biologia Sintética foi discutido em que pé andam as pesquisas envolvendo microalgas – uma das milagrosas fontes energia sustentável e fixação de CO2 – no contexto da Biologia Sintética. O apresentador da vez, João Molino, com base nos conhecimentos que vem adquirindo no seu doutorado na Farmácia (FCF) aqui na USP, nos deu um review dos trabalhos com microalgas usadas em Biologia Sintética, além de falar um pouco de como as microalgas são incríveis para converter energia solar em bioprodutos e – consequentemente – fixar CO2. Com isso ele sugere no final algumas oportunidades que poderíamos usar para projetos do iGEM do ano que vem.
Na penúltima reunião do Clube de Biologia Sintética foi discutido em que pé andam as pesquisas envolvendo microalgas – uma das milagrosas fontes energia sustentável e fixação de CO2 – no contexto da Biologia Sintética. O apresentador da vez, João Molino, com base nos conhecimentos que vem adquirindo no seu doutorado na Farmácia (FCF) aqui na USP, nos deu um review dos trabalhos com microalgas usadas em Biologia Sintética, além de falar um pouco de como as microalgas são incríveis para converter energia solar em bioprodutos e – consequentemente – fixar CO2. Com isso ele sugere no final algumas oportunidades que poderíamos usar para projetos do iGEM do ano que vem.
Vídeo com “Pipotecnica”
Como tivemos problemas envolvendo a transmissão da reunião (que já era feita de maneira precária), resolvemos gravar novamente a apresentação, só que desta vez utilizando uma nova pirotecnia dos vídeos da internet, o Popcornmaker (veja mais sobre ele nessa palestra de 4min no TED). Junto ao vídeo irão aparecer muitos links e informações extras diretamente da wikipédia em inglês (nunca substimem a wikipédia em inglês!), portanto se quiser saber mais sobre alguma informação “ao vivo” durante o vídeo, cheque os links! [clique na imagem abaixo para ir ao vídeo em outra aba]
Anotações Pessoais
Apesar do custo/benefício das pesquisas de microalgas na indústria não ser muito bom – segundo o que o Mateus me contou outro dia – suas características são muito provocativas para serem usadas como solução ecológica para muitos problemas e melhorar bastante processos de produção de bioprodutos já existentes. Ela faz coisas simplesmente incríveis. Como o João mostra no vídeo, ela é campeã na produção de galões/acre de óleo, além de poder viver em ambientes completamente isolados, como em uma garrafa fechada por exemplo – diga aí, qual ser vivo que você encontra no seu dia a dia (sem contar microalgas né…) que consegue viver muito bem e por muito tempo num ambiente completamente fechado e sem ar! Ela também fixa CO2 que é uma beleza, produz hidrogênio (hidrogênio cara!) e ainda pode ser usada como biorremediador (e de fato é naturalmente) para limpar áreas contaminadas!
Além de ser muito interessante biotecnologicamente, as microalgas são um grande gargalo na biologia sintética devido à falta de BioBricks e de elementos de DNA padronizados, como suas sequências terminadoras. O que é bem legal para o Registry of Parts e para o iGEM: partes inéditas! O time do chile do iGEM deste ano foi um dos primeiros a conseguir transformar cianobactérias (“parentes” das microalgas) com sucesso utilizando BioBricks na competição, o que é um bom indício para se trabalhar com microalgas.
O Grande Desafio
Apesar disso tudo, trabalhar com microalgas é algo bem desafiador, muito por causa do item mais valioso que se tem em laboratório: tempo. Um processo inserção de vetor nas células que duraria apenas (no máximo!) 2 dias de trabalhando com E.coli, com nossas amigas verdinhas duraria cerca de uma a duas semanas (se não me engano, segundo o que o João me contou). Para se fazer um projeto desse tipo estaríamos um pouco limitados para o pouco tempo do iGEM, a não ser que nos organizássemos muito bem (ainda estamos trabalhando nesse quesito). Mas o interessante é que aparentemente elas são bem geneticamente estáveis quando se tratando do vetor inserido; pelo o que o João nos contou, algumas microalgas transformadas duram anos com o seu novo pedaço de DNA. O processo de transformação também é aparentemente tranquilo e sem muito mistério.
Seguindo com os nossos objetivos de criar um projeto para o iGEM, muitas ideias surgiram da potencialidade de trabalhar com microalgas. Particularmente, comecei a pensar num sistema em que as microalgas “alimentassem” uns extremófilos, para que eles produzissem um efeito desejado com suas habilidades únicas da natureza – habilidades extremas! Mas discorro sobre isso em futuros posts.
Referência Principal
Durante o vídeo, muitas referências interessantes apareceram com ajuda do Popcornmaker, mas a referência principal que guiou o overview que o João nos fez é essa aí embaixo:
- Wang B, Wang J, Zhang W, & Meldrum DR (2012). Application of synthetic biology in cyanobacteria and algae. Frontiers in microbiology, 3 PMID: 23049529
Brasil no iGEM
 Para quem acha, não somos o primeiro grupo a ambicionar ir a um evento do iGEM. Lá pelos idos de 2009 o Brasil participou pela primeira vez da competição representado pela Unicamp, conseguindo trazer aqui para o lado de baixo do hemisfério uma medalha de ouro já “de cara”, equiparando-se às universidades mais renomadas do mundo, como Cambridge, Harvard e outras.
Para quem acha, não somos o primeiro grupo a ambicionar ir a um evento do iGEM. Lá pelos idos de 2009 o Brasil participou pela primeira vez da competição representado pela Unicamp, conseguindo trazer aqui para o lado de baixo do hemisfério uma medalha de ouro já “de cara”, equiparando-se às universidades mais renomadas do mundo, como Cambridge, Harvard e outras.
Sob o projeto intitulado “Microguards”, o time brasileiro criou um mecanismo de transformação de E.Coli’s e de leveduras (S. Cerevisiae) em “micro guardas”, que atacam bactérias contaminantes de biorreatores de etanol, no caso as bactérias do gênero Lactobacillus (do tipo daquelas que regulam a sua ação intestinal quando você bebe o seu leite fermentado – Yakult, Chamyto, e etc -). Usar um mecanismo como esse na indústria brasileira evitaria o atual desperdício de milhares de litros de álcool que deixam de ser produzidos por causa do açúcar consumido pelos microorganismos invasores nos bioreatores de etanol. Cerca de 5 à 10 % da produção é desperdiçada por causa desses ladrõezinhos.
O Mecanismo
Reconhecimento
 “ColiGuards”
“ColiGuards”
Criando uma espécie de “sistema imune de bioreatores”, duas linhagens de E.coli’s são criadas no meio de cultivo: a linhagem natural chamada de workers linage e a linhagem de “microguards”, ou killers linage; a população dos dois tipos de linhagem varia dependendo do grau de contaminação do meio, ou seja, quando não há contaminantes, muito poucas bactérias workers transformam-se em killers e vice-versa.
A transformação em killers é ativada por, um metabólito secundário de quorum sensing, AI-2 (se quiser saber mais clique aqui), que é liberado tanto pela bactéria contaminante quanto pelas E.Coli’s selvagens, é reconhecido pelas E.Coli workers, que não produzem AI-2, o que as induz a diferenciarem-se em killers. Além disso elas podem detectar os contaminates através da conjugação bacteriana com os microorganismos invasores, abilidade que só as killers passam a ter no processo de diferenciação, utilizando-a apenas naqueles microorganismos que possuem um plasmídeo diferente do seu (veja “recognition by conjugation” aqui).
 “YeastGuards”
“YeastGuards”
Para as Leveduras o mecanismo de reconhecimento da presença de contaminantes é um pouco mais simples: a produção de lactato provinda do consumo de açúcar dos Lactobacillus é utilizada para ativar gatilhos gênicos – o que somente é possível através da sensibilização das leveduras ao lactato, utilizando uma permease expressa pela levedura para facilitar sua incorporação à célula – que induzem um ataque aos contaminantes.
Diferenciação
Os brasileiros aproveitaram o elegante design feito pelo time francês da universidade de Paris no iGEM de 2007 para criação de um sistema de diferenciação das E.Coli’s em microguards. Com a atuação de uma recombinase (a cre recombinase), parte dos genes do plasmídeo que transforma o microorganismo sofre excisão, tornando-se um pequeno pedaço de DNA circular com baixa taxa de atividade e sem origem de replicação, enquanto os outros genes que permaneceram no plasmídeo passam a se tornar ativos devido ao seu reposicionamento na fita de DNA logo após um promotor, como pode ser visto na imagem logo abaixo:

Lox71 e lox66 são os sítios do DNA onde atuam as cre recombinases (atuação simbolizada pelo raio vermelho). Após sua ação, os genes após as regiões de terminação (T) são reposicionados - no caso do exemplo o gene dapA - próximos ao promotor que antes era da região excisionada (na imagem o promotor Tet), e por isso tornam-se ativos.
O controle populacional dos microorganismos killers é mantido através do gene ftsK da imagem acima: um gene que produz uma proteína determinante na divisão celular, sendo uma “máquina literal de segregação de cromossomos”. Dessa maneira, evita-se que a população de killers cresça demais e se torne um “tiro pela culatra”, diminuindo a produção de etanol.
Outro aspecto bastante interessante da construção das ColiGuards é o mecanismo de controle de uma população basal de killers em um biorreator não contaminado. Eles usaram uma criativa construção feita por outro time do iGEM, o de Caltech de 2008, que se aproveitou de uma “falha” da atividade da DNA-polimerase para randomizar a expressão de um gene em uma população bacteriana. Essa “falha” é a característica que a polimerase possui de ignorar ou repetir algumas sequências de nucleotídeos que são repetidas longamente no código, produzindo uma cópia de DNA com um número variável dessas repetições (fenômeno chamado de SSM, do inglês: slipped-strand mispairing), a consequência disso é que a sequência codificadora (o gene) pode ser deslocada da posição correta em relação ao seu start codon se as repetições não forem múltiplas de 3 (porque cada códon é constituído de 3 nucleotídeos), e assim a tradução não é feita corretamente. Veja figura abaixo:

As letras maiúsculas correspondem cada uma a um aminoácido, traduzido por um códon (grupo de três nucleotídeos). ATG é o start codon e TAA é um codon de parada formado pelo deslocamento da sequência codificadora. Repare que após uma SSM, uma repetição foi omitida.
Assim, as replicações de DNA que tiverem as repetições múltiplas ATGC múltiplas de três, terão o ajuste correto da região codificadora em relação ao start códon e a tradução correta da cre recombinase será possível:

Como esse tipo de erro da DNA-polimerase não é tão frequente, apenas uma pequena parte da população se diferenciará em killer se as repetições forem colocadas antes do gene da cre recombinase.
Para diferenciar bactérias workers na vizinhança de contaminantes, todos os microguards possuem uma outra cópia do gene recombinase, mas controlado por um promotor sensível ao AI2 liberado pelos Lactobacillus e pelas Coliguards quando detectam a presença dos invasores (figura ao lado).
Mecanismo de Ataque
A construção desses mecanismos de ataque aproveitou-se da maior diferença estrutural entre as E.coli’s e Leveduras, e o Lactobacillus: a coli é uma bactéria Gram negativa (possui duas membranas celulares, existindo entre elas uma fina parede celular) e o invasor é Gram positivo (possui apenas uma parede celular no exterior e uma membrana celular interior do compartimento celular), enquanto a parede celular das leveduras é constituída de carbohidratos, diferentemente dos peptídeoglicanos do contaminante. Sabendo disso, o grupo resolveu expressar substâncias que ataquem a parede celular de peptídeoglicanos exposta dos Lactobacillus e encontraram as lisozimas como a arma perfeita para isso, sendo usadas tanto na secreção como no método kamikaze.
O grande problema dos métodos de secreção é que eles podem se comportar como contaminantes, além da consequente redução do grau de efetividade da enzima durante o tempo devido à seleção natural. A solução encontrada para esse problema foi a conjugação de um plasmídeo (lembrando que somente as killers têm a abilidade de conjugação!) com o gene de uma endonuclease, a colicina, destruindo os Lactobacillus “por dentro”. Para que as próprias E.coli’s não fossem afetadas por esse gene letal, foi inserido um gene de resistência que torna a expressão de colicina inofensiva às Coliguards.
iGEM 2009
Melhor do que descrever como foi o projeto, nada melhor do que as próprias pessoas que participaram do evento para explicarem o que fizeram! Confira a apresentação do grupo brasileiro no Jamboree realizado em 2009 no MIT:
E nesse ano há outro time em associação com a universidade francesa de Saint-Etienne, com o projeto intitulado “Stress Wars”. Confira o que já está sendo feito nesse novo projeto franco-tupiniquim grupo junto aos outros links interessantes logo abaixo:
- UNICAMP-EMSE team 2011: http://2011.igem.org/Team:UNICAMP-EMSE_Brazil
- UNICAMP-Brazil team 2009: http://2009.igem.org/Team:UNICAMP-Brazil
- Caltech team 2008: http://2008.igem.org/Team:Caltech
- Paris team 2007: http://parts.mit.edu/igem07/index.php/Paris