Os verdadeiros revolucionários
Ninguém duvida que Apple, Tesla e Google sejam sinônimos de inovação e tecnologia. E que seu sucesso está diretamente ligado a mentes brilhantes como Steve Jobs e Elon Musk, visionários de primeira hora. Mas os investimentos necessários para desenvolver a tecnologia que está presente num iPhone ou num Tesla com certeza espantaria a maior parte dos investidores do mercado, impacientes e ávidos por lucros no espaço de tempo mais curto possível.
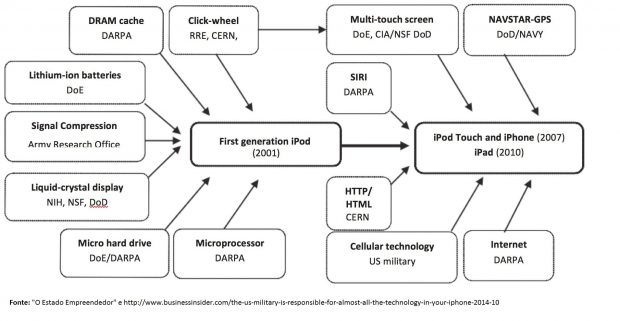
Pouca gente sabe que foi o dinheiro do contribuinte americano ao longo de décadas que ajudou estas empresas a serem o sucesso que são e forneceu as bases para o surgimento dos produtos revolucionários de algumas das empresas mais inovadoras do mundo. Essa é a ideia defendida pela economista Mariana Mazzucato no seu livro “O Estado Empreendedor”.
Apesar das críticas, às vezes merecidas, de serem estruturas pesadas e burocráticas, as agências do governo americano financiaram pesquisas que trouxeram para a indústria de consumo a tela touch screen, o display de cristal liquido, o SIRI e ajudaram Steve Jobs a fazer da sua empresa a marca icônica que ela é.
Mas não são apenas as agência de defesa que produzem inovações. A partir de 1983 as empresas de biotecnologia se beneficiaram de um belo empurrão dado pelo Governo. Naquele ano foi aprovado nos Estados Unidos o Orphan Drug Act, decreto que fornece incentivos fiscais e subsídios de P&D para o desenvolvimento de medicamentos para o tratamento de doenças raras (doenças que afetam menos de 200 000 pessoas). Sem este apoio eles praticamente não existiriam. Esta iniciativa foi fundamental para o crescimento de empresas como Amgen, Genentech e Genzyme. Hoje os medicamentos para doenças raras são responsáveis por mais de 70% da receita das principais empresas de biotecnologia.
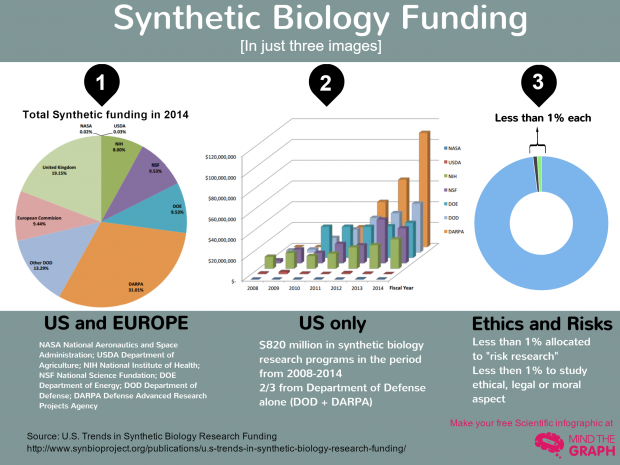
Os produtos inovadores dependem fundamentalmente dos investimentos em pesquisa de base. Se quisermos saber o que o futuro nos reserva, temos que olhar o que está sendo feito hoje neste campo, um exemplo é a biologia sintética. Entre 2008 e 2014 as agências governamentais americanas já investiram quase 1 bilhão de dólares em pesquisas nessa área. O MIT-Broad Foundry é um dos institutos apoiados com recursos da agência de defesa DARPA e, de acordo com Ben Gordon, diretor do Foundry, ele tem o objetivo de trazer soluções para a saúde, a agricultura e a química que são desafiadoras demais para a indústria e para a academia.
Aqui no Brasil, além da pesquisa básica, o Governo tem investido em pesquisas na indústria e na criação de empresas de base tecnológica como forma de aproveitar as pesquisas, as patentes e o conhecimento produzido nas universidades. Entre as agências de fomento está a Financiadora de Estudos e Projetos (FINEP), que por meio do Plano Inova Empresa apoia a inovação em setores que o Governo Federal considera estratégicos. Lançado em 2013, é o projeto mais ambicioso de inovação que o país já teve. Uma verdadeira revolução. Ele conta com R$ 32,9 bilhões que serão destinados a áreas como energia, nanotecnologia e biotecnologia nos próximos anos.
Uma das instituições que receberam recursos do FINEP foi a Embrapa Agroenergia. Em um prédio com 10.000 m2 no Distrito Federal, a instituição trabalha para produzir um futuro sustentável para o planeta e para a economia brasileira. Mais de 50 projetos estão em desenvolvimento, entre eles a pesquisa em processos de conversão da biomassa em bioenergia, biomateriais e químicos renováveis de alto valor agregado. Mas nem sempre os recursos públicos são bem aplicados. O programa Ciência sem Fronteiras é um desses casos. Com mais de 100.000 bolsas, a maior parte delas para alunos de graduação, o programa investe na formação de pessoal altamente qualificado, colocando estudantes e pesquisadores em instituições de excelência no exterior e atraindo jovens talentos para trabalhar no Brasil. Apesar de proporcionar a vivência internacional aos alunos, a iniciativa falha pela falta de um acompanhamento rigoroso das disciplinas e atividades desenvolvidas por eles no exterior. Isso nunca poderia acontecer num programa onde o custo de um aluno pode chegar a 80.000 reais por ano. Rogerio Meneghini, professor da USP e diretor científico da biblioteca virtual SciELO afirma: “No contexto do desenvolvimento da ciência, a ida desses estudantes de graduação ao exterior não vai fazer qualquer diferença”.
Num momento de dificuldades econômicas como a que enfrentamos hoje e viveremos nos próximos anos, é vital utilizar com mais sabedoria os recursos públicos. Gerenciar e avaliar os resultados ficou mais importante do que nunca. Ter a parceira das empresas privadas é essencial. Resumindo: é fundamental não perdermos essa revolução que o Governo começou.
BioWindows – A Microsoft pode estar reinventado o futuro mais uma vez
Considerado uma das pessoas mais importantes do século pela revista Times, Bill Gates dispensa apresentações. No início do ano, ele retirou mais de um bilhão de dólares investidos na Coca-Cola, McDonalds e Exxon. O motivo? Gates pretende focar em empresas que visam além do lucro também melhorar o futuro da humanidade. A Microsoft também está seguindo essa meta. Nos últimos anos a empresa tem trabalhado para acelerar pesquisas que envolvem desde o aquecimento global até a biologia sintética.
Talvez você não saiba, mas Bill Gates investe em biotecnologia há mais de 25 anos. A primeira empresa em que ele apostou foi a ICOS, um laboratório focado no desenvolvimento de medicamentos para o tratamento de doenças inflamatórias que mais tarde foi comprado pela farmacêutica Eli Lilly. Mas os investimentos não pararam por aí. A Microsoft tem uma divisão empenhada em solucionar problemas globais por meio da tecnologia, a Microsoft Research. Nesse laboratório são desenvolvidos softwares capazes de acelerar pesquisas em diferentes campos da ciência.
Na biologia sintética, o design racional de células que desempenhem comportamentos previsíveis permanece um desafio para os pesquisadores, e é nisso que uma das pesquisas da empresa está focando. A Microsoft Research vem trabalhando há mais de dez anos para criar softwares e linguagens de programação que permitam ao pesquisador selecionar tudo aquilo que ele deseja que uma célula sintética execute, sem precisar se preocupar com todas as possíveis combinações de genes e sequências regulatórias disponíveis. A partir dessas informações, o software forneceria as melhores sequências de DNA para que a célula realize tal função, economizando tempo e dinheiro.
Os pesquisadores da empresa desejam que tais programas sejam fáceis de usar, atingindo assim um maior número de usuários possíveis.
Em mais de uma ocasião, Bill Gates comentou que se fosse adolescente nos dias de hoje teria optado por estudar biologia e genética. Em 2012, a receita doméstica de produtos geneticamente modificados nos Estados Unidos foi de U$ 350 bilhões, valor que tem crescido 15% ao ano. Para efeito de comparação, é o equivalente a quase 10% do PIB do Brasil no mesmo período. De acordo com Stephen Emmott, responsável pela ciência computacional da empresa, a Microsoft quer fazer para os softwares de modelagem aquilo que ela fez para os softwares de negócios Excel e Word. Isso mostra que ela pode estar reinventando o futuro mais uma vez.
Cientista coreano conhecido por ressuscitar cachorros tem agora novo desafio
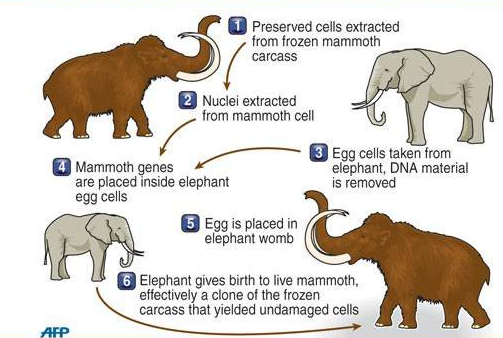
Essa semana o caso do dentista americano acusado de atrair um leão para fora do parque nacional de Zimbábue e matá-lo ganhou bastante repercussão na mídia, principalmente por se tratar de um leão conhecido e monitorado por GPS. Ele se chamava Cecil, e também foi decapitado. O dentista que pagou 50 mil dólares para localizar e matar o animal, continua foragido, assim como inúmeros outros caçadores, que segundo a World Wildlife Fund (WWF), são grandes responsáveis por reduzir o número de animais vertebrados (mamíferos, aves, peixes e etc.) pela metade nos últimos 40 anos. Por isso alguns pesquisadores planejam ressuscitar diversas espécies. Um desses pesquisadores é Hwang Hoo-Suk, que está confiante em fazer um mamute andar na Terra novamente.
O laboratório SOOAM biotech, na Coréia do Sul, é famoso por clonar cachorros por 100 mil dólares, e agora eles planejam ressuscitar os mamutes. Com o derretimento do permafrost, milhares de mamutes estão aparecendo na Sibéria, e muitos deles em excelente estado de conservação, suficiente para alguns já terem até provado a carne de mamutes congelados há mais de 10 mil anos. A carne de mamute é tão comum por lá, que também é usada como isca por caçadores de raposas. O desafio agora é encontrar células que estejam intactas e com DNA viável, pois até agora apenas DNA fragmentado foi encontrado. Caso se consiga isto, o mamute seria clonado usando a mesma técnica utilizada para clonar a ovelha Dolly. Mas essa certamente não será uma tarefa fácil.
Em 2003, cientistas na Espanha ressuscitaram o bucardo, uma cabra dos Pirineus que foi caçada extensivamente e dada como extinta no ano 2000. Utilizando células que haviam sido preservadas, os pesquisadores transferiram o núcleo intacto para um óvulo de cabra, originando 208 embriões. Dos 208 embriões implantados, apenas uma gestação teve sucesso, e em 2003, nasceu o primeiro animal fruto da “desextinção”. Os pesquisadores na Coréia do Sul planejam fazer algo parecido, porém usando os óvulos e a barriga de aluguel de uma elefanta.
Após anos de trabalho, milhares de dólares investidos e 208 embriões, o bucardo sobreviveu por apenas sete minutos. Como o pesquisador George Church coloca em seu livro, “Regenesis“, sete minutos pode parecer pouco, mas basta lembrar que o primeiro voo dos irmãos Wright durou 12 segundos. Sessenta e seis anos depois, o homem pisou na Lua. Mesmo que a “desextinção” se torne viável, a melhor alternativa por muito tempo continuará sendo a preservação do meio ambiente.
Assista ao documentário (com legenda!).
[youtube_sc url=”https://www.youtube.com/watch?v=xmlpSOHc5A4″]
Cabras™, porcos™, peixes™ e muito mais com marca registrada
Você pode não saber, mas o Brasil já é o segundo maior produtor de transgênicos do mundo, ficando atrás apenas do Estados Unidos. Até agora as alterações genéticas em escala comercial estiveram restritas apenas às plantas, com destaque para a soja e o milho. Mas não são apenas novos vegetais que os pesquisadores desejam colocar no mercado, animais geneticamente modificados aguardam aprovação para serem comercializados enquanto outros ainda passam por diversos testes. Confira cinco pesquisas que podem oferecer imensas vantagens para os consumidores, os produtores e até para o meio ambiente.
EnviropigTM
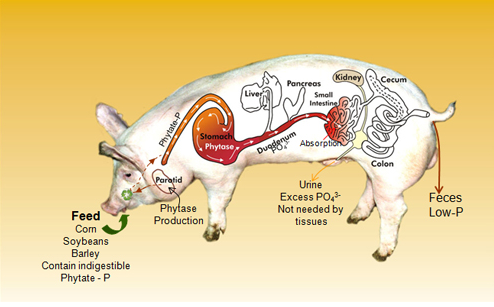
Com quase um bilhão de porcos no mundo, uma grande preocupação que se tem é o destino dos dejetos ricos em fósforo e nitrogênio. O EnviropigTM criado na Universidade de Guelph, no Canadá, foi concebido para ser um porco “eco-friendly”.
Grande parte do fósforo presente nos grãos e sementes que compõem a alimentação dos porcos estão na forma de fitato, um composto que eles não conseguem digerir e portanto são excretados. Para contornar esse problema, foi introduzido em seu genoma o gene da enzima fitase, o que torna possível aos porcos a digestão e absorção do fósforo desses alimentos. Dessa forma, o Enviropig excreta até 70% menos fósforo nas fezes.
A pesquisa começou em 1995 e já recebeu patentes nos EUA e China, mas ainda não foi aprovado para consumo.
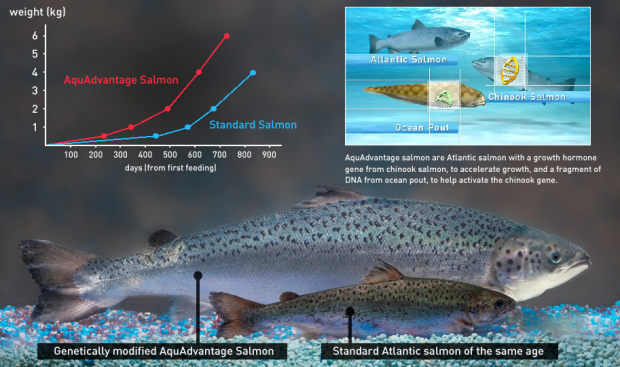
AquAdvantage®
O salmão AquAdvantage® deve ser o primeiro animal transgênico a ser aprovado para consumo pela Food and Drug Administration (FDA). Ele é igual ao salmão do atlântico em tamanho, aparência e gosto, exceto pelo fato de ter em seu genoma o gene de hormônio do crescimento do salmão do pacífico e DNA do peixe-carneiro americano.
Essas alterações permitem que o salmão da empresa AquaBounty Technologies cresça duas vezes mais rápido que o salmão selvagem e consuma 25% menos alimento durante sua vida. Os peixes são estéreis e criados apenas em cativeiro. A pesquisa teve início em 1989 e, embora tenha-se concluído que o salmão não apresente riscos, ele ainda está em revisão pela FDA.
Porcos ricos em ômega-3
O consumo de alimentos ricos em omega-3 é recomendado por possuir poder anti-inflamatório e reduzir os riscos de doenças cardiovasculares. No entanto, nem todos os seres humanos têm acesso a esse tipo de alimento presente em grande quantidade nos peixes marinhos. Para oferecer uma carne alternativa ao peixe, rica em ômega-3, a solução até agora era alimentar os animais com linhaça, peixes e outros produtos marinhos, o que altera as características sensoriais da carne.
Para conseguir uma carne rica em ômega-3 sem alterar a alimentação dos animais, pesquisadores nos Estados Unidos criaram porcos com o gene fat-1 do verme Caenorhabditis elegans. O gene fat-1 permite que os porcos consigam converter ômega-6 em ômega-3. A pesquisa foi publicada na revista Nature Biotechnology mas ainda não há previsão de comercialização.
Cabras Transgênicas
A diarréia é responsável pela morte de mais de meio milhão de crianças todos os anos. Uma pesquisa que teve início em 1999 na Universidade da Califórnia – Davis (UCD), tem como objetivo obter um leite com poder anti-microbiano produzido com cabras que receberam o gene humano da enzima lisozima, proteína abundante no leite materno.
A pesquisa que começou nos Estados Unidos agora é feita em parceria com a Universidade do Ceará. O leite produzido pelas cabras transgênicas já mostrou efeitos terapêuticos em porcos, animais que têm um sistema digestivo parecido com o nosso. Os próximos passos serão os testes clínicos em humanos.
Porcos “editados”
Utilizando ferramentas de edição de genoma (Zinc Finger Nucleases – ZFNs e Transcription Activator-Like Effector Nucleases – TALENs), pesquisadores do Instituto Roslin, no Reino Unido, criaram porcos resistentes ao virus da febre suína Africana, capaz de matar os porcos europeus em menos de 24 horas.
Para tornar os porcos europeus resistentes foi necessário que uma única letra no genoma fosse alterada. A alteração foi feita com base no porco selvagem africano que é resistente ao virus, porém incapaz de cruzar com o porco europeu.
Os testes com os porcos devem começar esse ano e se tiverem sucesso serão submetidos à aprovação pela FDA.
Capazes de causar menor impacto ambiental, resistir a doenças e serem mais saudáveis, os animais geneticamente modificados podem ter um importante papel na alimentação da população mundial, que deve atingir 9 bilhões em 2050. Mesmo ainda enfrentando a oposição de ativistas, muitos pesquisadores acreditam que os animais modificados por ferramentas de edição de genoma devem ter sua aprovação acelerada pelas agências reguladoras. Caso isso aconteça, o Brasil não será apenas um dos maiores produtores de plantas™ geneticamente modificadas, mas também de animais™.
5 empresas que estão utilizando a biotecnologia para mudar o mundo
Todos os dias, mais de 90 milhões de barris de petróleo são produzidos e mesmo assim este número continua a crescer. O consumo deve chegar próximo de 100 milhões em 2020. A queima e o refino do petróleo são grandes responsáveis pela poluição, o aquecimento global e danos à saúde. Felizmente muitas empresas estão buscando alternativas renováveis e aqui vão 5 exemplos.
DuPont
A mais velha da lista, a DuPont utiliza microrganismos e açúcar de milho para produzir produtos renováveis de diferentes finalidades, desde vestuário até móveis. Um desses produtos, o Sorona® EP, é um plástico termo resistente atualmente empregado no Toyota Prius.
LanzaTech
Fundada em 2005 e com sede nos EUA, a LanzaTech emprega arqueobactérias que são capazes de transformar a poluição em produtos renováveis, como combustíveis, nylon e borracha.
Gases poluentes ricos em carbono provenientes da indústria, como a siderúrgica, vão para um bioreator, onde os microrganismos fermentadores se encarregam de tranformá-los em etanol e outras moléculas que são utilizadas para produzir plástico, fibras sintéticas e borracha. O etanol da LanzaTech não depende de fontes de alimentos e terras aráveis, como o álcool obtido a partir do milho ou da cana-de-açúcar. Com apenas 10 anos, a empresa já possui 85 patentes e outras 250 pendentes. A primeira fábrica em escala comercial começa a operar ainda esse ano na China, e a companhia aérea Virgin Atlantic deve ser a primeira a voar com o combustível.
Sapphire Energy
Fundada em 2007, e com três plantas na Califórnia e Novo México, a Sapphire Energy é a primeira e única empresa no mundo a utilizar algas para produzir petróleo. Seu petróleo renovável recebeu o nome de Green Crude, e não depende de água potável nem de terras aráveis. As algas capturam dióxido de carbono durante processo, o que faz o Green Crude neutro em emissões de CO2. Com investidores como Bill Gates, a família Rockfeller e a Monsanto, a empresa espera que ele seja competitivo com o petróleo fóssil já em 2018.
NatureWorks
Em média, cada pessoa no mundo irá consumir 45 kg de plástico em 2015, e apenas uma parte disso será reciclado. De olho neste mercado, a NatureWorks criou o Ingeo™, um plástico revolucionário.
Sua fábrica nos EUA utiliza o açúcar proveniente de plantas e leveduras para produzir ácido lático, o responsável por formar o plástico PLA (ácido polilático). Ele é capaz de substituir o PET e o PS, presentes em garrafas, talheres descartáveis e eletrônicos. Ao contrário do plástico derivado do petróleo, ele é facilmente reciclado e gera 60% menos gases do efeito estufa.
Amyris
Criada em 2003 e com cerca de 400 funcionários, a empresa americana emprega biologia sintética para produzir produtos químicos renováveis utilizados em cosméticos, fragrâncias, combustíveis e medicamentos.
Com apoio da fundação Bill & Melinda Gates, a Amyris desenvolveu uma levedura capaz de criar um precursor da artemisinina, o medicamente utilizado no tratamento da malária, doença que mata todos os anos mais de meio milhão de pessoas. Em 2013, a empresa farmacêutica Sanofi iniciou a produção da artemisinina utilizando essa tecnologia.
No Rio de Janeiro e em São Paulo, o combustível da empresa, o Diesel de Cana™, é utilizado diariamente por cerca de 400 ônibus. Em 2014, a GOL fez o primeiro vôo comercial com uma mistura contendo 10% de combustível renovável.
A Revista The Economist do mês passado apontou a biotecnologia como um dos campos que mais poderão contribuir para a evolução humana no futuro. Pelo jeito algumas empresas já saíram na frente.
Biotecnologia sem fronteiras: o monopólio da inovação está com seus dias contados
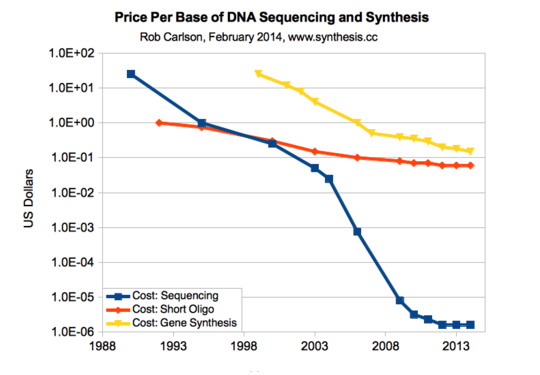
O baixo custo dos computadores, a lei de Moore ainda em vigor e o acesso à internet democratizaram os meios de produção, distribuição e educação. Hoje não somos apenas consumidores passivos, mas também produtores ativos. E na ciência, não é diferente. Vivemos a era “Pro-Am”, em que amadores dedicados, inovadores e conectados trabalham como profissionais, uma realidade na astronomia, na ciência da computação, e agora da biotecnologia. Basta um computador, conexão com a internet e um cartão de crédito para encomendar DNA e testar algumas das mais novas técnicas de clonagem e edição de genoma, até então só acessível a universidades e grandes empresas.
Em 1987, a luz de uma estrela, que explodiu há 168.000 anos chegou a Terra. Foi então que astronômos  amadores junto com profissionais, confirmaram a teoria do que ocorre quando uma estrela explode, uma das maiores descobertas da astronomia do século XX. Hoje muito mais gente participa da ciência que até então dependia de equipamentos sofisticados e caros. Uma dessas tecnologias, foi disponibilizada por John Dobson, responsável por criar um poderoso telescópio usando materias de segunda-mão. Dobson se recusou a lucrar com sua invenção, a qual nunca patenteou. Essa democratização chegou à biologia com o movimento Do-it-yourself Biology (DIYbio) em 2008, em que profissionais e amadores desenvolvem projetos em laboratórios comunitários, constroem equipamentos por uma fração do preço e aproximam a comunidade da discussão sobre organismos geneticamente modificados.
amadores junto com profissionais, confirmaram a teoria do que ocorre quando uma estrela explode, uma das maiores descobertas da astronomia do século XX. Hoje muito mais gente participa da ciência que até então dependia de equipamentos sofisticados e caros. Uma dessas tecnologias, foi disponibilizada por John Dobson, responsável por criar um poderoso telescópio usando materias de segunda-mão. Dobson se recusou a lucrar com sua invenção, a qual nunca patenteou. Essa democratização chegou à biologia com o movimento Do-it-yourself Biology (DIYbio) em 2008, em que profissionais e amadores desenvolvem projetos em laboratórios comunitários, constroem equipamentos por uma fração do preço e aproximam a comunidade da discussão sobre organismos geneticamente modificados.
Em um desses laboratórios comunitários, o BioCurious, na Califórnia, são desenvolvidos projetos que envolvem a recente ferramenta de edição de genoma CRISPR, uma bioimpressora capaz de “imprimir” células de E. coli, e um projeto do iGEM para a produção de queijo “vegan”, com proteínas do leite produzidas por leveduras, e não vacas.
Atualmente é possível comprar ou montar os próprios equipamentos, como o OpenPCR, um termociclador usado para amplificar DNA, a centrífuga OpenFuge e o robô OpenTrons, que permite automatizar seu trabalho de bancada. Em breve, o MiniION™ será realidade, um sequenciador portátil e descartável que pode analisar sua amostra, seja ela de um microrganismo ou sangue, em qualquer lugar do mundo, sem precisar de milhares de doláres em equipamentos ou treinamento. Além de aparelhos mais acessíveis, o preço de síntese por base de DNA (A, T, C, G) já custa apenas alguns centavos e tem caido ano após ano. No futuro, nada impede que etapas custosas de um projeto sejam terceirizadas, ocupando a capacidade ociosa de laboratórios ou serem realizadas por empresas prestadoras de serviços, como já acontece com a síntese e o sequenciamento de DNA na China.
Projetos como esses vão reduzir o custo de se buscar o novo, trazendo soluções acessíveis para a saúde, alimentação e preservação do meio ambiente, quebrando o atual monopólio da inovação presente apenas nas grandes instituições como empresas e universidades. Como em toda abertura democrática, espera-se que muito mais gente se beneficie deste passo da ciência: a biotecnologia sem fronteiras.
Confira a iniciativa acontecendo em Sâo Paulo – http://synbiobrasil.org/st/diy/
Referências:
The Pro-Am Revolution – How enthusiasts are changing our economy and society – Charles Leadbeater and Paul Miller
Time for new DNA synthesis and sequencing cost curves – Rob Carlson
Produção em massa, o jeito chinês de fazer ciência
Depois de 35 anos de impressionante desenvolvimento, a China começa a ser reconhecida não apenas pela sua capacidade de produzir e exportar produtos de baixo custo mas também com alta tecnologia. E não são apenas bens materiais que ela anda produzido em massa, mas agora o sequenciamento do DNA, por exemplo. É o que alguns já estão chamando de “Bio-Google”. Seguindo a filosofia, “sequenciar tudo aquilo que se mexe”, o Beijing Genomics Institute (BGI) possui 50% da capacidade de sequenciamento do mundo e já leu mais de 50.000 genomas nos últimos anos.
O BGI fica localizado em uma antiga fábrica de sapatos em Shenzhen, o Vale do Silício chinês,  e participa de projetos que vão desde sequenciar uma bactéria até a busca por genes ligados à inteligência. Muitos dos estudos são conduzidos por pesquisadores de todas as partes do mundo, e o BGI oferece preços baixos e até de graça para aqueles que compartilham seus resultados.
e participa de projetos que vão desde sequenciar uma bactéria até a busca por genes ligados à inteligência. Muitos dos estudos são conduzidos por pesquisadores de todas as partes do mundo, e o BGI oferece preços baixos e até de graça para aqueles que compartilham seus resultados.
Um desses projetos, chamado 3M, planeja sequenciar 3 milhões de genomas, sendo 1 milhão de plantas e animais, 1 milhão de humanos e 1 milhão de microrganismos. O Instituto participa de outras iniciativas como o controverso sequenciamento de 2000 pessoas com QI elevado para desvendar genes que influenciam na inteligência. Há também o sequenciamento de 10.000 pessoas com autismo, o de 2.000 pessoas obesas e o de 2.000 magras. Além disso, também possui uma parceria com a fundação Bill e Melinda Gates para o desenvolvimento da agricultura e saúde em países subdesenvolvidos. Trabalhando em projetos como esses, o BGI já colaborou em mais de mil publicações em revistas de alto impacto como Nature, Science e Cell.
O Instituto está chamando a atenção do mundo pelo volume de dados que estão sendo produzidos. Segundo Lincoln Stein, pesquisador do Ontario Institute for Cancer Research, a questão agora não é mais o quão próximo estamos de um sequenciamento do genoma que custe U$1.000, mas sim, de uma análise do genoma a U$100.000. Outro desafio é o armazenamento de tudo isso, já que 6 terabytes de dados são produzidos por dia.
Zhang Yong, um dos pesquisador do BGI, acredita que na próxima década o Instituto será capaz de organizar toda essa informação biológica em uma espécie de “Bio-Google”.
Muitos achavam que o BGI estava apenas prestando um serviço, quando em 2013 ele adquiriu um fabricante de equipamentos e software para sequenciamento localizado na Califórnia, a Complete Genomics, e isso já está tirando o sono de muita gente. Agora a pesquisa pode se tornar ainda mais barata, já que equipamentos e reagentes representam grande parte do orçamento.
Enquanto isso no Brasil, a falta de planejamento de médio e longo prazo, a dificuldade em reter pessoas qualificadas, a ausência de uma cultura empreendedora que estimule a criação de empresas que sirvam de sustentação à pesquisa, nos deixa cada dia ainda mais distante dos centros de vanguarda. E isso apenas contribui para elevar nosso custo em P&D, além de nos tornar reféns de equipamentos e reagentes importados, notadamente agora com o dólar passando a barreira dos R$3,00. Talvez em breve nossas amostras sejam analisadas por um equipamento chinês em Los Angeles, Istambul, Shenzhen, ou então, numa facility chinesa ao lado da sua casa.
Mais um acidente a favor do pesquisador
Em 1986, o pesquisador Richard Jorgensen estava trabalhando com petúnias quando aconteceu um acidente. Jorgensen desejava criar uma petúnia roxa, porém muito mais roxa do que o normal. Para alterar a cor, ele procurou super-expressar a enzima chalcona-sintase (CHS) introduzindo um gene quimérico de CHS, uma enzima limitante da via responsável pela coloração das flores [1]. Mas a alteração genética realizada teve um resultado totalmente inesperado e grande parte das pétalas se tornaram brancas e não roxo escuro como ele desejava.
O mistério só foi desvendado em 1998 e conferiu o Prêmio Nobel a dois pesquisadores americanos, gerou especulações sobre a cura de dezenas de doenças e mais recentemente se tornou uma nova ferramenta para a biologia sintética. Andrew Fire e Craig Mello descobriram que Jorgensen havia esbarrado no que eles vieram a chamar de RNA de interferência (RNAi), um mecanismo de silenciamento gênico.
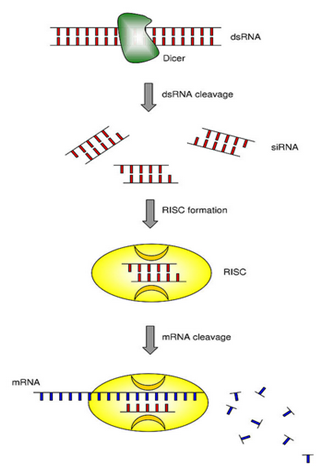
O gatilho para o mecanismo de silenciamento gênico por RNAi ocorre quando um RNA de dupla fita (dsRNA) se forma. Ao ser identificado um dsRNA, a enzima Dicer corta o dsRNA em fragmentos menores que se ligam ao complexo protéico RISC (RNA-induced silencing complex). Em seguida, apenas uma das fitas de RNA permanece presa ao complexo, que serve para ir em busca de fitas de mRNA que sejam complementares [2]. Quando um mRNA complementar é detectado, ocorre o pareamento com o RNA preso ao complexo e ele é então clivado e degradado. Como o mRNA não pode ser traduzido, o gene tem sua expressão reduzida (Figura 1). O silenciamento é uma poderosa ferramenta que agora encontrou uma aplicação na biologia sintética, por meio da evolução dirigida.
O sonho da biologia sintética de construir sistemas que funcionem de modo previsível e robusto frequentemente entra em conflito com a complexidade dos sistemas biológicos. Além de sua complexidade, o comportamento dos microrganismos dependem de um contexto, o que também dificulta o uso de partes padronizadas [3]. Logo, métodos de evolução dirigida possuem grande utilidade, já que em princípio dispensam informações detalhadas de estrutura, funcionalidade e de mecanismos de um sistema [4].
Um dos primeiros experimentos sobre evolução realizado em laboratório foi feito por William Dallinger, em 1880. Dallinger conseguiu que seus microrganismos que cresciam a 18ºC passassem a crescer a 70ºC, no entanto o experimento levou 7 anos e envolveu aumentar a temperatura de sua incubadora gradualmente até que eles fossem capazes de sobreviver. Hoje existem diversos métodos de evolução dirigida e um pouco menos demorados, entre eles o RAGE – RNAi assisted genome evolution.
RAGE é um método utilizado em Saccharomyces cerevisiae e é bastante útil quando se deseja obter fenótipos complexos. Fenótipos complexos, como a tolerância ao ácido acético, dependem da alteração de múltiplos genes e são de grande interesse para a indústria na produção de combustíveis e outros compostos químicos. O uso dessa técnica reduz a expressão de genes (knockdown) em escala genômica e possibilita identificar genes que até então não se imaginava terem papel em determinadas funções.
Para que tais genes possam ser identificados, é necessário criar uma biblioteca de RNAi. A biblioteca é criada fragmentando o DNA genômico com uma enzima de restrição e clonando os fragmentos em um plasmídeo com promotores convergentes, necessário para que RNAs de fita dupla sejam formados. Como S. cerevisiae não possui a via de RNAi, também é necessário inseri-la [6].
Via inserida e biblioteca criada o processo de evolução pode começar. Quando o knockdown de um gene for promissor, tal gene pode ser integrado e novos ciclos de transformação e screening podem ser feitos repetidamente (Figura 2), até que seu objetivo seja alcançado, ou pelo menos, chegue próximo dele.
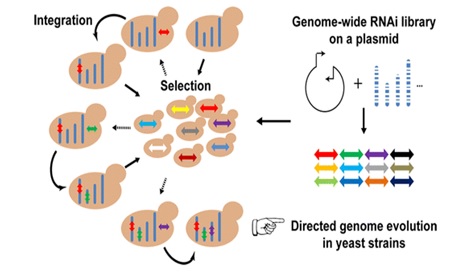
Figura 2: Evolução dirigida pelo método RAGE – Imagem retirada de: http://pubs.acs.org/doi/abs/10.1021/sb500074a
Em 2006, Fire e Melo ganharam o Prêmio Nobel em fisiologia ou medicina por desvendarem o fenômeno observado por Jorgensen. Muitas descobertas acidentais fazem parte da história da ciência, como a penicilina, o raio X e o microondas, por exemplo. O método de evolução dirigida utilizando RNAi pode também em breve facilitar a vida de muitos pesquisadores que buscam aprimorar seus microrganismos.
Referências
- NAPOLI, C.; LEMIEUX C.; JORGENSEN R. lntroduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. The Plant Cell, Vol. 2, p. 279-289.
- CLARK D. P. Molecular Biology. Vol. 2, chapter 11 (2010)
- DOUGHERTY, M. J.; ARNOLD, F. H. Directed evolution: new parts and optimized function. Current Opinion in Biotechnology, 2009, 20:1–6
- COBB, R. E; SUN,N.; ZHAO H. Directed evolution as a powerful synthetic biology tool. Methods (2012)
- SI, T.; LUOZ, Y.; BAO, Z.; ZHAO, H. RNAi-Assisted Genome Evolution in Saccharomyces cerevisiae for Complex Phenotype Engineering. ACS Synth. Biol. (2014)
Por que ATP!?
![]()
A maioria das pessoas que sabe um pouquinho de bioquímica aprende que o ATP é a “moeda energética” da célula. Maravilha. Mas aposto que se você perguntar o porquê disso, a resposta-padrão vai ser: “É porque é uma molécula muito energética, presente em reações de catálise e regulação de diversas enzimas na célula e blá-blá-blá…”. Lindo. Mas existem muitas moléculas muito energéticas no universo, e argumentar que o ATP é a “moeda energética” simplesmente porque as pessoas descobriram que na natureza ele está presente em várias reações enzimáticas acaba sendo um jeito bastante sofisticado de dizer “porque sim, zequinha!”.
[youtube_sc url=”https://www.youtube.com/watch?v=8Wldlug58Kc”]
Como construir um regulador enzimático e transportador energético na célula!?
Seguindo o espírito de engenheirar o metabolismo, como poderíamos, conhecendo as características físico-químicas dos elementos e compostos químicos, construir algo que seja como o ATP: uma coisa que regule de maneira sistêmica as atividades enzimáticas da célula, armazene energia e que seja fácil de se armazenar e ao mesmo de se usar!?
Bem, vamos pensar em energia como dinheiro: digamos que você seja rico, muito rico. Com muito dinheiro, a probabilidade de que você seja roubado é bem maior. Para que você mantenha sua riqueza é preciso que ela seja armazenada de um jeito “estável” (doce ilusão, mas considere isso como verdade, ok?) e por isso você coloca o seu dinheiro no banco. Você precisa proteger seu dinheiro e ainda acessá-lo facilmente – colocar tudo num baú e enterrar numa ilha desconhecida não vai ajudar na funcionalidade do seu dinheiro.
O mesmo funciona para a nossa moeda energética: ela não pode ser um composto cuja energia possa ser facilmente “roubada” por qualquer espécie química, mas também não pode ser pouco reativa a ponto de não conseguir regular a atividade enzimática e a transmissão energética. É preciso ter o controle desse “dinheiro” para gastá-lo facilmente onde você precisa: na manutenção da vida celular.
Então temos três coisas principais para a nossa molécula-moeda-energética: ela precisa ser estável o suficiente para que a energia não se “esvaia” facilmente, não tão estável a ponto de ser difícil usá-la e ainda reagir facilmente com proteínas.
Candidatos
Sabemos experimentalmente que o fosfato do ATP é a molécula-moeda. Mas porque só ela? Será que não tem nenhuma outra molécula que poderia fazer isso? Não poderia existir nenhuma outra coisa (ou coisas) alternativa que pudesse substituir o ATP ou que possa estar fazendo a mesma coisa?
Para encontrar um substituto (ou equivalente) para o ATP é preciso explorar análogos estruturais e energéticos dessa molécula. Selecionei dois possíveis candidatos:
TriSulfonatos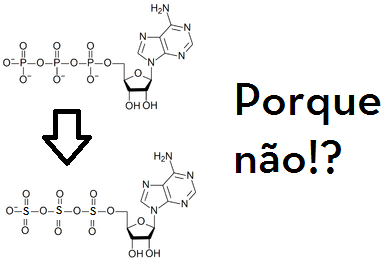
Da mesma forma que o ATP é um TriFosfato, porque ele não poderia ser um trisulfonato? A estrutura é bem similar e as energias das ligações éter (a ligação S – O – S) são bem parecidas também (veja). Além disso, sabe-se que há resíduos de aminoácidos que sofrem sulfonização.
Arsênico
Do mesmo jeito que teorizam as diferentes maneiras da vida ter surgido no universo, porque não 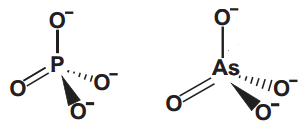 poderíamos ter um “ATP de Arsênico”!? O Arsênico fica logo abaixo do fósforo na tabela periódica e por isso faz o mesmo número de ligações, além de formar arsenato (e com isso diarsenatos e triarsenatos), de estrutura bem parecida com a do fosfato.
poderíamos ter um “ATP de Arsênico”!? O Arsênico fica logo abaixo do fósforo na tabela periódica e por isso faz o mesmo número de ligações, além de formar arsenato (e com isso diarsenatos e triarsenatos), de estrutura bem parecida com a do fosfato.
Que o melhor ganhe
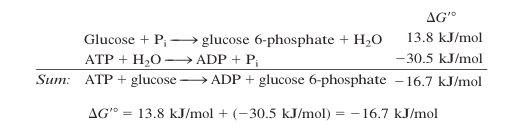
As reações que o ATP faz são reações de hidrólise: um fosfato tem sua ligação éter (O – P) quebrada resultando na fosforilação de alguma coisa. Essa ligação é de alta energia (em termos bioquímicos) e é espontânea (delta G negativo). Reações não termodinamicamente espontâneas que passam por uma etapa de fosforilação acabam se tornando espontâneas na soma geral da energia livre, como por exemplo na fosforilação da glicose:
(Imagem modificada da referência [6])
Apesar da reação de hidrólise ser espontânea, a fosforilação tem uma velocidade de reação suficientemente lenta para garantir a estabilidade que a “moeda energética” precisa para realizar reações úteis (não que hidrolisar e simplesmente gerar calor seja de todo inútil…).
Se eu conseguir mostrar que o arsenato ou o sulfonato são melhores em termos energéticos e cinéticos, um mundo novo de possibilidades bioquímicas se abrirá!
Pesquisei alguns dados na internet e montei a seguinte tabela:
(Dados retirados das refs [1], [2], [3] e [4])
O quão maior for a constante de hidrólise (k) da reação, mais rápida ela será. Com esses dados dá para ver claramente que o fosfato é um “banco energético” bem mais confiável que os arsenato e o sulfato, sendo cerca de 100000 vezes mais estável que estes dois últimos – mantendo sua facilidade de “uso” energético com o delta G de hidrólise negativo. O fosfato é, além disso, bem mais “rico” que os seus concorrentes.
O sulfato, apesar de ser um candidato tentador, é bem mais difícil se “ser um ATP” como o arsenato pode ser. Não consegui encontrar nenhuma molécula como a proposta na imagem anterior, apenas dissulfonatos – e ainda não ligados a uma adenosina! Mesmo que houvesse um “ATP de enxofre”, seria difícil que ele assumisse o papel de controle das atividades enzimáticas porque ele não reage com resíduos de aminoácidos tão bem como o arsenato e o fosfato.
O arsenato já ganhou espaço na mídia a algum tempo atrás com o suposto encontro de uma “vida baseada em arsênico” na terra. Existem de fato várias enzimas que se ligam a arsenato e ele consegue substituir o fosfato em muitos casos, mas causando efeitos tóxicos na célula ao competir com o fosfato para a fosforilação de moléculas durante o catabolismo. Mesmo que houvesse um organismo em que a toxicidade fosse tolerada, a eficiência do processo de “arsenização” seria pífia: muitas moléculas apenas iriam hidrolisar espontaneamente de maneira “inútil”, liberando calor – além de tudo isso, eu nem preciso argumentar a questão a abundância do Arsênico no meio ambiente!
Não é porque sim!
Enfim, agora estou convencido do porquê o ATP é como é: o fósforo e as ligações éter o fazem um composto único, com propriedades flexíveis e ideais para ser o transmissor de energia de compostos mais energéticos do metabolismo até os menos energéticos, fazendo reações termodinamicamente não-espontâneas acontecerem e regulando de maneira sistêmica todo o conjunto de milhares de enzimas das células que dependem da fosforilação para alternarem entre seus estados ativos e inativos.
Portanto, da próxima vez que você não encontrar a resposta que queria, é melhor tentar construir o que você não entende – afinal, qual seria melhor jeito de entender alguma coisa no final das contas!?
Referências
[1] Rosen, B., Ajees, A., & McDermott, T. (2011). Life and death with arsenic BioEssays, 33 (5), 350-357 DOI: 10.1002/bies.201100012
[5] Tabelas de Entalpias de Ligação
[6] Nelson, David L., Albert L. Lehninger, and Michael M. Cox. Lehninger principles of biochemistry. Macmillan, 2008.
CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
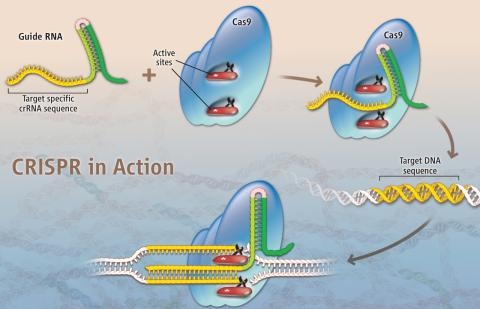 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência: