A Indústria Química do Futuro

Fig. 1 – “Bioreator”, arte de K-Kom – DeviantART.
“Um Químico é um mexedor de sopa, um operador fedorento.
Um Zimologista é alguém que ajuda a manter bilhões de pessoas vivas. Eu sou um especialista em cultura de leveduras. (…)
Este laboratório mantém a New York Yeast funcionando. Não há um dia, nenhuma maldita hora, que nós não tenhamos culturas de cada linhagem de levedura na empresa crescendo em nossas caldeiras. Nós checamos e ajustamos os fatores dos requisitos de comida. Nós garantimos que estão se reproduzindo bem. Nós ‘dobramos’ a genética, começamos as novas linhagens e as ‘limpamos’, descobrimos suas propriedades e moldamos elas novamente.(…)
Vinte anos atrás a Saccharomyces olei Benedictae era só uma linhagem com gosto horrível de sebo e boa para nada. Ela ainda tem gosto de sebo, mas seu conteúdo de lipídios foi forçado de 15 para 87 por cento. Se você usou a Esteira Expressa hoje, apenas se lembre que ela é lubrificada estritamente com S. o. Benedictae, linhagem AG-7. Desenvolvida aqui mesmo nesta sala.”Trecho de “As Cavernas de Aço” (1953), de Isaac Asimov – tradução livre.
![]() Dia 2 de Janeiro é aniversário do “bom doutor” Isaac Asimov, que já nos anos 50 descrevia um planeta Terra apinhado de pessoas em enormes conjuntos de construções urbanas, as “Cavernas de Aço”, título do livro que faz parte de sua série de romances sobre robôs que o fez ser um ícone da ficção científica. Mas um fato pouco lembrado é sua visão de como seria a indústria química do futuro, que se reflete até os dias de hoje. Antes de escritor, Asimov era bioquímico e portanto não é à toa que no mesmo ano em que foi descoberta a estrutura do DNA, ele já conseguisse prever as possibilidades industriais da manipulação genética como no trecho acima de seu livro. Nele um “zimologista” defende a importância de seu trabalho em criar leveduras que possam produzir os mais variados tipos de químicos, num futuro onde o petróleo mundial já se esgotou e a única fonte de matéria-prima é biomassa vegetal.
Dia 2 de Janeiro é aniversário do “bom doutor” Isaac Asimov, que já nos anos 50 descrevia um planeta Terra apinhado de pessoas em enormes conjuntos de construções urbanas, as “Cavernas de Aço”, título do livro que faz parte de sua série de romances sobre robôs que o fez ser um ícone da ficção científica. Mas um fato pouco lembrado é sua visão de como seria a indústria química do futuro, que se reflete até os dias de hoje. Antes de escritor, Asimov era bioquímico e portanto não é à toa que no mesmo ano em que foi descoberta a estrutura do DNA, ele já conseguisse prever as possibilidades industriais da manipulação genética como no trecho acima de seu livro. Nele um “zimologista” defende a importância de seu trabalho em criar leveduras que possam produzir os mais variados tipos de químicos, num futuro onde o petróleo mundial já se esgotou e a única fonte de matéria-prima é biomassa vegetal.

Fábrica da BASF em 1886 em Ludwigshafen, onde é a sede da empresa até os dias de hoje. Pelo menos olhando de longe, não é muito diferente das fábricas de hoje em dia, não? – Fonte: WIkimedia
Essa “bioindústria do futuro” nunca foi tão real como hoje. E já estava na hora. O cerne da maior parte da indústria química mundial ainda é a mesma desde quando o Brasil ainda era um império, mais ou menos na mesma época em que o petróleo começa a ser usado como principal fonte para fabricar produtos químicos industriais. Naquele tempo era impensável que os pequenos seres humanos poderiam causar um impacto ambiental tão grande num mundo tão vasto. Mas está ficando cada vez mais claro para a população de 7,7 bilhões de pessoas que esse modelo de 200 anos precisa mudar, ou ainda vamos ter que aceitar conviver com catástrofes climáticas.
Todo esse tempo de existência dá um privilégio enorme para esse sistema de produção permanecer desse jeito. Foi só com uma verdadeira revolução científica que a indústria de hoje ficou pronta para começar a mudar: a revolução da decodificação genética. Quando os cientistas terminaram de montar o quebra-cabeça que explicava como o DNA guardava instruções para as reações químicas que coisas vivas conseguem fazer, um universo de possibilidades se abriu. Tudo isso culminou nos anos 2000, com uma nova abordagem do que um dia foi conhecido como engenharia genética, a Biologia Sintética. Tratando células da mesma maneira como engenheiros elétricos tratam circuitos eletrônicos, os avanços da aplicação dessa nova disciplina foram muito mais rápidos do que a manufatura de químicos e hoje a bioindústria não apenas existe mas já prospera. A manufatura de químicos por rotas biológicas já está em estágio comercial (ver Figura 3) e a OCDE (o “clube dos países ricos”) estima que em 2030 produtos derivados dessa indústria corresponderão até 2,7% do PIB de seus países membros – para se ter uma ideia, é um valor próximo da riqueza média que toda a agricultura, pesca e extração de madeira geram nesses países hoje em dia.
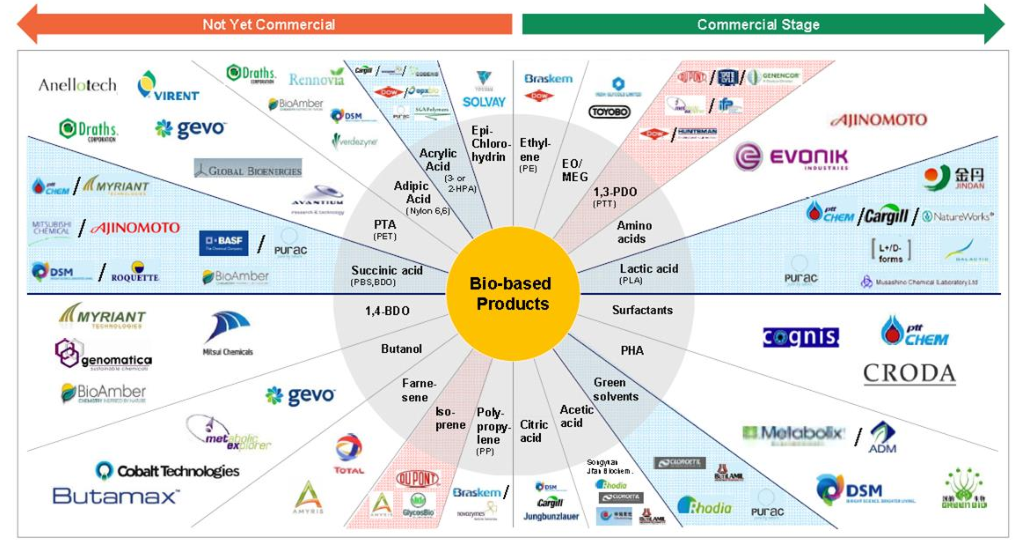
Figura 3: Exemplos de casos de sucesso de manufatura de químicos por rotas biológicas. Retirado de “Industrialization of Biology”, National Research Council, 2015. (Clique para ver melhor)
Boa parte das pessoas está completamente alheia a essas mudanças, muito porque falta algum tempo até verificarmos as estimativas para 2030 e ainda estamos vivendo só começo da mudança de “mexedores de sopa” para “zimologistas” na indústria. Até agora a Biologia Sintética foi a disciplina que melhor atendeu as expectativas do que seria o futuro da bioindústria, mas ainda estamos longe de produzirmos tudo a partir de microrganismos. Hoje a fronteira com o futuro está na imprevisibilidade e complexibilidade dos sistemas biológicos. Apesar da analogia ser válida, “programar” microrganismos não é a mesma coisa que computadores: são tantas variáveis ainda mal compreendidas dentro de uma simples célula que as coisas simplesmente não funcionam como deveriam e muitas vezes os cientistas não sabem exatamente o porquê. Pensando em focar esforços, vários pesquisadores convocados pelo conselho nacional de pesquisa norte-americano montaram um plano para os próximos 10 anos com os desafios a serem superados para uma “manufatura avançada de químicos” a nível industrial (ver Figura 4). Dentre os principais desafios estão o barateamento dos processos de uso de matérias-primas renováveis, aumento da eficiência dos processos de ganho de escala de bioprodução, desenvolvimento de ferramentas mais ágeis de construção de sistemas genéticos e “domesticação” de microrganismos ainda não explorados para uso industrial.
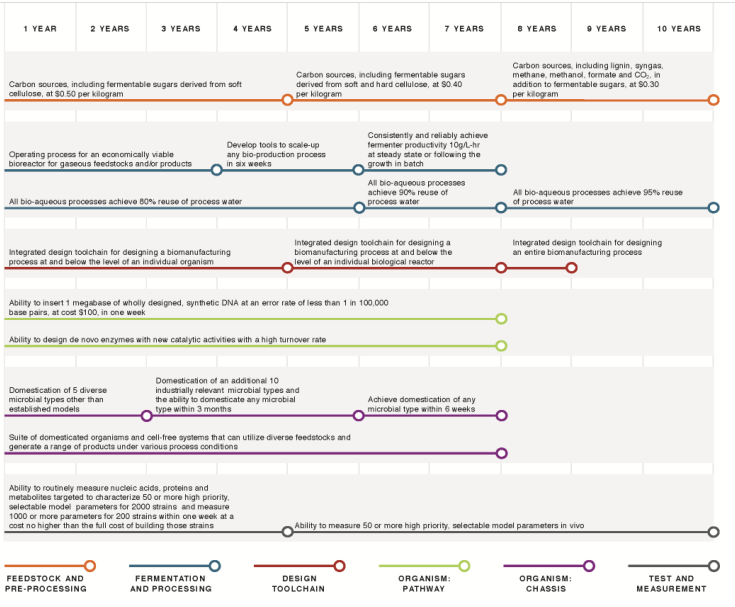
Figura 4: Planejamento de desafios a serem superados nos próximos 10 anos para concretização do potencial industrial da biotecnologia. Fonte: “Industrialization of Biology”, National Research Council, 2015. (Clique para ver melhor)
Parte dos executores desse plano são novos de laboratórios de biotecnologia chamados de “biofoundries”. São basicamente laboratórios altamente automatizados (Figura 5), capazes de realizar uma quantidade sem precedentes de experimentos e fazer medições, quase 24 horas por dia, 7 dias por semana. A estratégia é usar a “força bruta” das máquinas para conseguir lidar com uma quantidade enorme de variáveis, o que seria impraticável em outras condições.

Figura 5: “Biofoundry” versus um Laboratório de Biotecnologia comum. As bancadas de trabalho da Biofoundry são tomadas por robôs que automatizam todos os processos com baixa intervenção humana, enquanto um laboratório comum apenas produz conforme o número de pessoas que é possível caber numa mesma bancada! – Fonte: Ginkgo Bioworks (imagem da esquerda) e Wikimedia (imagem da direita). (Clique para ver melhor)
Em alguns lugares do mundo, como nos EUA, já se inicia um processo de estratificação de um ecossistema de empresas de biotecnologia e indústria: enquanto “biofoundries” fazem o “design” dos microrganismos (como a Ginkgo Bioworks, Genomatica e Zymergen), grandes empresas investem em bioprodução de commodities químicas (como Braskem, Dow, Basf) e moléculas de alto valor agregado, ao mesmo tempo que startups surgem barateando sequenciamento e síntese de DNA (como a Twist Biosciences) e viabilizando softwares necessários para toda a automação funcionar bem (como a Teselagen – que NÃO é uma empresa hipster reinventando a tecelagem, como pode parecer). Como a matéria prima desse tipo de indústria não é o petróleo, seria possível por exemplo existir um sistema de produção mais distribuído, como acontece hoje com as microcervejarias regionais: a manufatura acontece em muitas fábricas de pequena escala, usando fontes locais de material para servir de alimento para os microrganismos.
Nesse futuro muito próximo, a riqueza da biodiversidade é o código de DNA. É como se todo organismo, seja ele microscópico ou gigantesco como um Jatobá, fosse uma máquina biotecnológica que a própria natureza produziu, com “programas” para fabricação de tudo o que o organismo é capaz de fazer. Bastaria então fazer o “download” desse programa, estudá-lo, e fazer “upload” dele em espécies de microrganismos industriais.
O jeito que a indústria química fabrica produtos hoje em dia é baseado em “rotas de síntese”. Ou seja, para se chegar em uma molécula, é preciso planejar todas as reações químicas, que devem acontecer do jeito certo, para no final se ter em grande quantidade o produto que se deseja. E essas rotas não surgem “do nada”, são resultados de anos de pesquisa teórica e prática; uma criação verdadeiramente humana. A visão da “bioindústria” química do futuro aproveitaria as “rotas” programadas nos “softwares” da Natureza. Dentro desse imaginário, a riqueza da biodiversidade está em descobrir novas rotas “prontas” inclusive para moléculas químicas que já existem. Daí a importância da “domesticação” de novas espécies para uso industrial. Hoje por enquanto é possível se contar com os dedos de apenas uma mão a quantidade de espécies que são mais usadas para bioprodução industrial – e sim, a levedura S. cerevisiae sugerida por Asimov está entre elas. Quantas espécies de microrganismos ainda não conhecidos pela ciência podem existir em uma grama de solo amazônico, por exemplo? Enquanto engenheiros biológicos se esforçam para pressionar essas poucas espécies a produzir mais “bioproduto”, podem haver centenas de espécies que, se exploradas geneticamente, poderiam produzir muito mais e muito mais facilmente do que a melhor cepa que já se conseguiu construir.
É claro que ainda existe toda uma indústria química para se “biotecnologizar”, mas já é um grande feito ter as principais commodities químicas sendo bioproduzidas e comercializadas de maneira competitiva com a indústria petroquímica. Isso mostra o peso dos imaginários sociotecnológicos que criamos: mesmo com grandes desafios a serem resolvidos, as consequências políticas e sociais dessas expectativas atraíram e atraem investimento suficiente para tornar real aquilo que a pouco tempo atrás era mera ficção científica. Esse imaginário de uma biotecnologia futurista já tem até organização política representativa no Brasil: a “frente parlamentar da bioeconomia”, que defende os interesses das bioindústrias emergentes, em articulação com o agronegócio.
Asimov com certeza sabia que uma posição visionária precisa de expectativas familiares o suficiente para produzir um futuro reconhecível. A série “Black Mirror” (se você não conhece, fica a dica!) usa essa mesma receita. Se essas expectativas forem muito diferentes da experiência coletiva ninguém a levará a sério, e talvez seja por isso que as histórias de Asimov parecem envelhecer pouco com o passar das décadas: elas se baseiam em imaginários sociais sólidos que existem até hoje, e que são provavelmente tão importantes quanto as próprias tecnologias para dar forma ao futuro.
E aí, já imaginou? É assim que o futuro começa.
Referências
- National Academies Press (2015).
Industrialization of Biology: A Roadmap to Accelerate the Advanced Manufacturing of Chemicals
Report of National Research Council of the National Academies Epub ISBN: 0-309-31655-3 - ASIMOV, Isaac. The caves of steel. 1954. New York: Spectra, 1991.
- HILGARTNER, Stephen; MILLER, Clark; HAGENDIJK, Rob (Ed.). Science and democracy: making knowledge and making power in the biosciences and beyond. Routledge, 2015.
- https://www.camara.leg.br/noticias/560651-camara-lanca-frente-parlamentar-mista-da-bioeconomia/
CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
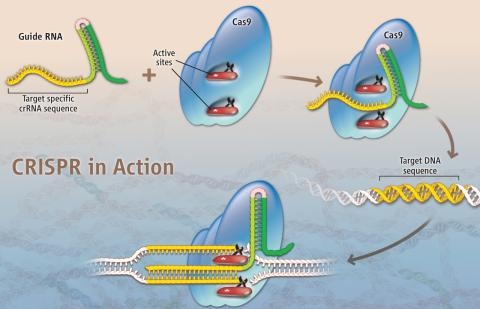 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência:
Transposons: pedaços de DNA que mudam de endereço no genoma.
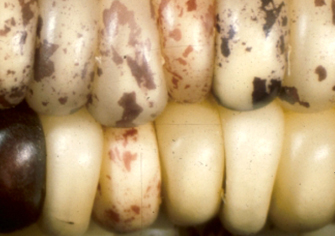
Mudança no padrão das cores do milho devido a interação entre os genes responsáveis pelo pigmento e elementos transponíveis.
Você sabia que alguns trechos de DNA têm a capacidade de mover-se no genoma, saindo de um cromossomo e se inserindo em outro? Pois é, e esta descoberta foi feita há muito tempo atrás por uma brilhante cientista, antes mesmo da descoberta da estrutura do DNA por Watson e Crick. Barbara McClintock ao observar a relação entre os padrões das cores do milho e algumas quebras cromossomais, percebeu que algumas destas quebras ocorriam com uma freqüência muito mais alta do que o esperado e, surpreendente, sempre no mesmo cromossomo. Após uma série de experimentos, a cientista percebeu que esta quebra ocorria devido a inserção de pedaços de DNA vindos de outros cromossomos que alteravam a expressão dos genes de pigmento. Confiante nos seus experimentos, Barbara então propôs que havia elementos transponíveis no genoma, ou seja, os genes não tinham um endereço fixo, mas tinham um mecanismo para movimentar-se dentro do genoma.
Se nos dias de hoje esta ideia parece bastante surpreendente, imagine como foi a recepção desta ousada ideia no começo da década de 50, quando a cientista começou a publicar seus resultados. Eles foram ignorados e ridicularizados pelos seus contemporâneos, levando a cientista a parar de publicar suas descobertas sobre o tema. Somente no início da década de 70 alguns elementos transponíveis foram identificados em bactéria validando assim a teoria de Barbara. Entretanto, o reconhecimento completo de uma das mais importantes descobertas da biologia somente aconteceu no início da década de 80, quando a cientista foi homenageada com o prêmio Nobel.
Atualmente vários estudos já relacionaram os elementos transponíveis, chamados de transposons, a uma série de doenças e como grande fonte de variabilidade genética. Hoje sabe-se que boa parte do genoma é composto destes elementos e dois mecanismos principais já foram identificados. O primeiro deles é um mecanismo de recortar-e-colar, onde alguns pedaços do DNA simplesmente saem de um lugar e movem-se para outra parte do genoma. O outro mecanismo é do tipo cortar-e-colar, gerando mais de uma cópia no genoma. Este último mecanismo é chamado de retrotransposons e consiste em quase metade do genoma humano e será tema de um próximo post.
FAQ #8 Como ver se a transformação gênica deu certo?
Já colocamos nossos plasmídeos nos bichinhos, mas ainda não acabou. Essa é a hora de saber se tudo que fizemos até aqui deu certo!
Primeiro, colocamos eles na placa de seleção com antibiótico e só aqueles que realmente incorporaram o plasmídeo vão sobreviver porque ganharam um gene amigo de resistência ao antibiótico. Agora pegamos uma parte das células sobreviventes e fazemos um teste usando a PCR (você lembra dessa técnica, né?).
Como testar com a PCR?! Roubamos os plasmídeos dessas células e tentamos multiplicar o pedaço de DNA que foi grudado nele adicionando primers, nucleotídeos e enzimas sob aquecimento e desaquecimento. Se o nosso plasmídeo modificado tiver sido incorporado pelos micro-organismos haverá a multiplicação do pedacinho e podemos vê-los em uma eletroforese. Caso contrário, nada foi copiado e nada vai aparecer na eletroforese. Então temos o sinal de que alguma coisa não deu certo por aqui e as chances de erros deste teste são bem pequenas.
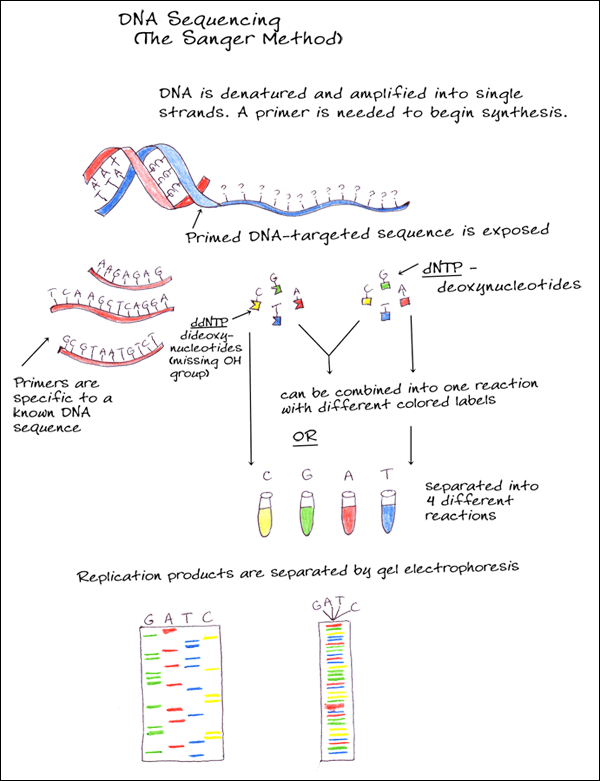
Depois vamos para um teste melhor ainda, o sequenciamento. Este teste diz exatamente qual é a sequência de pares de base da molécula do plasmídeo e acabam de vez suas dúvidas se a transformação deu certo ou não! O sequenciamento começa parecendo um processo de duplicação normal de DNA. Mas além de cadeias molde, enzimas e nucleotídeos normais, há nucleotídeos especiais sintetizados (ddNTP’s) que possuem duas boas propriedades: emitem luz e interrompem o prologamento da cadeia a partir de onde foram adicionados. Então, imagine uma molécula de DNA que possui um par de base A-T em um determinado comprimento e considere que a base A pertence à cadeia molde e a base T à cadeia complementar. Se esta base T for um nucleotídeo especial temos como identificar “quem é” e “qual sua posição” na cadeia, pois ele emitirá uma cor específica para Timina e o comprimento de sua cadeia está interrompido na posição exata (nº 2 em verde). 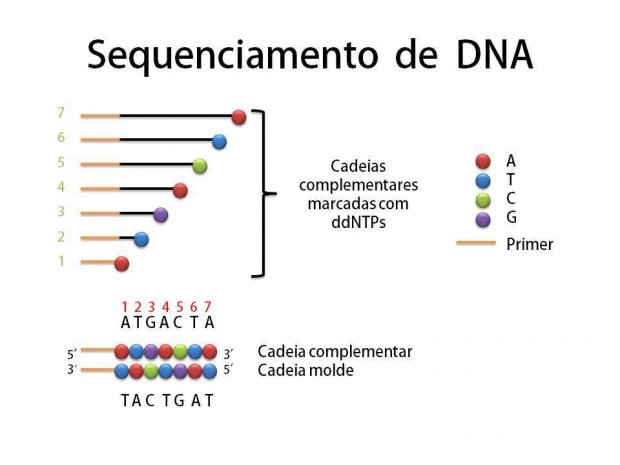 Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Para saber mais sobre sequenciamento genético, clique aqui.
Por Otto Heringer e Viviane Siratuti.
FAQ #7 Como enfiar o plasmídeo nas células?
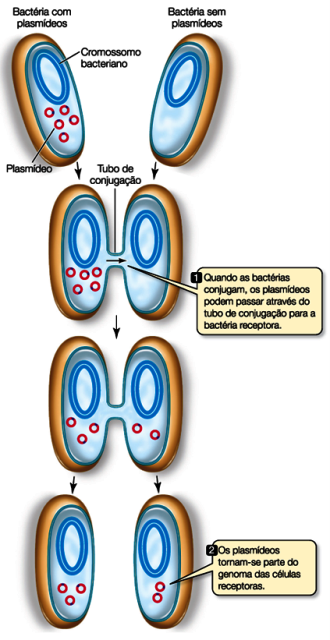
Dar e receber plasmídeos não é nenhuma novidade para algumas células!
As bactérias costumam trocar informações genéticas assim e esse processo é chamado de conjugação, portanto elas já possuem toda maquinaria necessária pra isso! Desse jeito elas aumentam muito a variabilidade e as chances de sobrevivência da população.
Podemos usar esse mecanismo natural para enfiar os plasmídeos, mas na maioria das vezes damos uma ajudinha usando choques térmicos ou elétricos.
Para entender o choque térmico, assista o vídeo do JOVE aqui. E do choque elétrico, aqui.
Por Otto Heringer e Viviane Siratuti.
FAQ #6 Como ver se nossos plasmídeos incorporaram os pedacinhos de DNA?
Sabendo o tamanho do plasmídeo e dos pedacinhos estimamos um tamanho para nosso novo plasmídeo e agora é só conferir se estão como esperado!
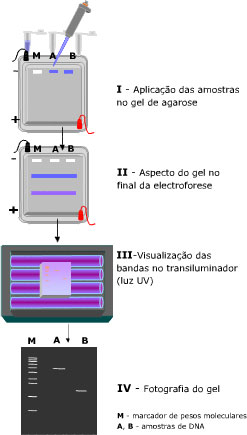
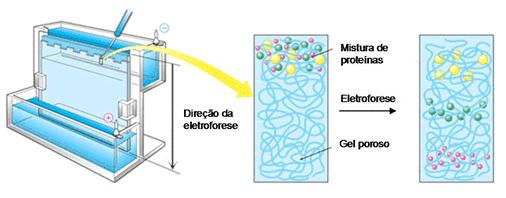
Para isso, existe uma técnica de separação chamada eletroforese em gel, onde as moléculas são “peneiradas” por algum polímero (gel de poliacrilamida ou de agarose) quando se movimentam atraídas ou repulsadas pelos eletrodos de cargas opostas que são colocados nas extremidades do gel. Ou seja, uma molécula com carga negativa caminha para o eletrodo de carga positiva e vice-versa. E as moléculas de mesma carga correm de maneiras diferentes pelo gel de acordo com seus pesos moleculares e tamanhos, as menores e mais “leves” passam com mais facilidade pelo gel enquanto as maiores e mais “pesadas” ficam mais retidas. No final, temos as moléculas separadas ao longo do gel e podemos comparar com a ladder (uma “régua” feita de moléculas com pesos e tamanhos já conhecidos) para saber se nossos plasmídeos estão no tamanho esperado.
Depois de confirmar pela eletroforese que nossos plasmídeos foram modificados, precisamos “salvá-los” do gel para colocar nas células. Basicamente, cortamos o pedaço de gel que está com nossos plasmídeos e colocamos em tubinhos com uma pequena coluna de sílica dentro (imagem aí embaixo). Então as moléculas de DNA se ligam à coluna, fazemos uma lavagem para tirar o gel e depois diluímos o DNA para retirá-lo da coluna!
Para saber mais sobre eletroforese, assista o vídeo do JOVE Science aqui e purificação aqui. 🙂
Por Otto Heringer e Viviane Siratuti.
FAQ #5 Como colocar os pedaços de DNA no plasmídeo?
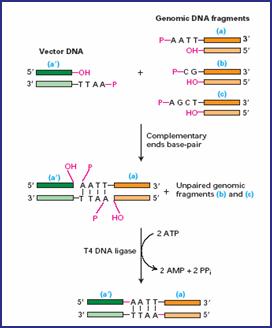
Para abrir os plasmídeos (lembrando que é um DNA circular) e depois grudar os pedacinhos de DNA que extraímos pela Miniprep e multiplicamos pela PCR, usamos as chamadas enzimas de restrição e de ligação. Isso tudo ainda fora das células, ok?!
As enzimas de restrição reconhecem e cortam os plasmídeos em regiões específicas que queremos, isto é, cortam em determinadas sequências de pares de base. Inclusive existem dois tipos de corte, podendo formar extremidades coesivas (cortes em comprimentos diferentes entre as duas fitas) ou cegas (corte reto na dupla fita).
Feito isso, agora usamos as enzimas de ligação para grudar as extremidades dos plasmídeos às extremidades dos pedacinhos, onde as bases se complementam.
Legal saber também que o nome dessas enzimas estão sempre relacionados com os nomes dos organismos onde elas foram encontradas (veja a enzima EcoRI, por exemplo).
Para assistir o vídeo do JOVE Science sobre enzimas de restrição, clique aqui. E enzimas de ligação, aqui.
Por Otto Heringer e Viviane Siratuti.
FAQ #4 Onde colocar esses pedaços de DNA?
“No genoma, né, dêr!”. Certo, mas como? E será que o genoma é o único lugar que podemos colocar esse novo pedacinho de DNA no micro-organismo que queremos modificar? Não! Existe outro lugar também e ele se chama plasmídeo (quem já jogou BioShock vai soltar umas sinapses a mais agora).
O plasmídeo é um DNA circular presente em várias espécies de seres vivos e é responsável por conter informações valiosas envolvendo a sobrevivência do organismo a fatores externos (como por exemplo sua resistência a um antibiótico), além de ser o principal ator da transferência horizontal de informação genética, ou seja, a passagem de um DNA funcional de um ser vivo para outro sem haver hereditariedade (é aí que o jogo BioShock extrapola isso para seres humanos). Adivinha de onde veio a ideia de usar o plasmídeo como transmissor – “vetor” – de informação genética para modificar as células? Veio exatamente desse mecanismo natural de realizar transferência horizontal de genes que vários micro-organismos possuem, então aproveitamos para fazer a transferência das informações genéticas que nós queremos!
E se vamos usar plasmídeos é preciso extraí-los também! Os plasmídeos são moléculas de DNA assim como os pedacinhos obtidos a partir de um genoma, e multiplicados pela PCR, que vamos introduzir no microorganismo, mas aqui há uma etapa importante durante a extração de DNA onde é feita a separação do conteúdo plasmidial do genômico.
O vídeozinho abaixo tem uma animação no ínicio e depois mostra o procedimento do isolamento do plasmídeo em lab!
[youtube_sc url=”http://www.youtube.com/watch?v=8xEDEJ0DHFA”]
Por Otto Heringer e Viviane Siratuti.
FAQ #3 Como copiar o código em laboratório?
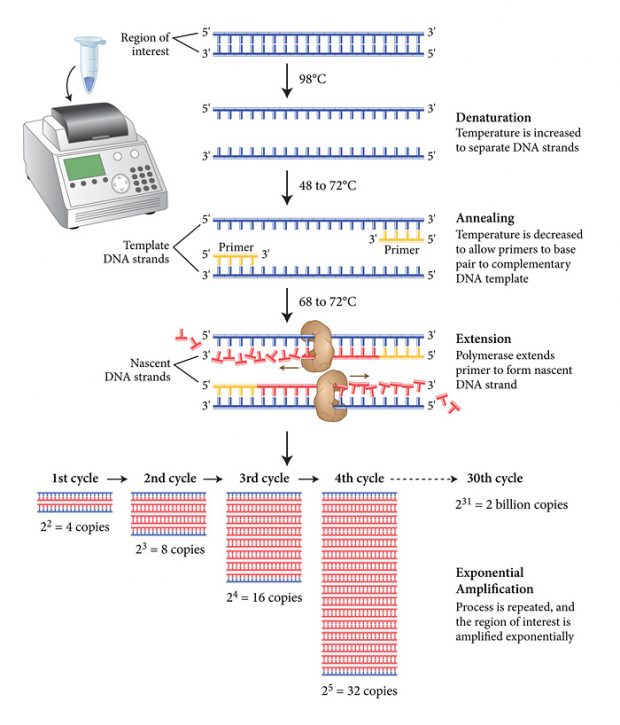
Ahá! Essa é a pergunta que mudou a biotecnologia. Até conseguirmos fazer isso, nós (i.e. humanidade) passamos por um caminho bem interessante envolvendo prêmios nobel e momentos epifânicos. O videozinho explica muito melhor do que esse texto corrido como funciona a metodologia pra se fazer isso, a Reação em Cadeia da Polimerase (Polimerase Chain Reaction), o tão amado e odiado (quando simplesmente não funciona) PCR!
[youtube_sc url=”http://www.youtube.com/watch?v=vmlLj1aLZ7s”]
Encurtando a história: usando uma enzima que trabalha “sentando” no molde de uma fita única de DNA, aquecimento e desaquecimento (para “ligar” e “desligar” essa enzima) e pedacinhos de nucleotídeo, conseguimos fazer milhões de cópias de apenas uma única molécula de DNA – é por isso que o pessoal do CSI (o seriado) consegue uma grande informação genética usando apenas resíduos quase desprezíveis de material biológico.
Por Otto Heringer e Viviane Siratuti.
FAQ #2 Como extrair um pedaço de DNA desejado?
Queremos pegar um pedacinho codificante de DNA (responsável por alguma característica no organismo) de um outro pedaço maior de DNA. OK, mas onde fica esse outro pedaço? Bem, existem pedaços disso praticamente a todo o seu redor. Onde há vida, há informação genética que pode ser “pega”. Extraímos ele basicamente fazemos lavagens usando solventes de diferentes “afinidades de dissolução” (a grosso modo) com as biomoléculas da célula até restar o DNA.
Se você quer saber, existem até kits comerciais para se fazer isso (como por exemplo a imagem abaixo), são os famigerados “kits de miniprep”. “Mini” porque em geral são bastante usados kits que trabalham com pequenos volumes (microlitros), mas também existem os kits de “midiprep” e “maxiprep”, para volumes maiores.
Assista o vídeo caso você queira saber mais do processo (aviso: a realidade às vezes não é algo tão excitante como você imaginava).
[youtube_sc url=”http://www.youtube.com/watch?v=P12DPa-g8Ro”]
Aliás, se você ficou com vontade de extrair DNA em casa, é bem simples, clique aqui.
Por Otto Heringer e Viviane Siratuti.