CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
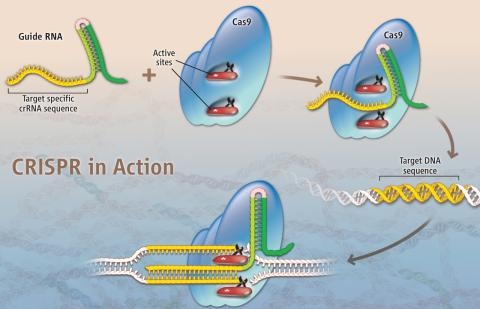 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência:
Bactérias na guerra contra o câncer
 Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Atualmente, desvendar o sistema de comunicação utilizado por células cancerígenas tem sido foco de inúmeras pesquisas. Como em qualquer guerra moderna, destruir o sistema de comunicação inimigo pode causar grandes danos. E na guerra contra o câncer provavelmente não será diferente. Impedir as células de se comunicarem pode evitar que elas adotem estratégias inteligentes tais como ficarem dormentes durante quimioterapia ou até mesmo matar células irmãs para obtenção de alimento. Uma vez entendido a linguagem utilizada por estas células, podemos utilizar isto ao nosso favor, interferindo nas mensagens e fazendo com que células dormentes sejam acordadas durante a quimioterapia ou até mesmo induzir as células cancerígenas a matarem umas as outras.
Finalmente, uma possível estratégia futura seria recrutar bactérias para derrotar o câncer. Elas poderiam ser utilizadas para “ensinar” as células do sistema imunológico a reconhecer e matar as células cancerígenas. É importante ressaltar que ainda compreendemos muito pouco sobre os mecanismos envolvidos no câncer e novas abordagens são necessárias para superá-lo. Entretanto, quem sabe num futuro próximo, estaremos em uma era de guerra cibernética biológica onde bactérias serão inteligentemente projetadas para derrotar o câncer.
Referências:
“Bacterial survival strategies suggest rethinking cancer cooperativity” Eshel Ben-Jacob, Donald S. Coffey, Herbert Levine. Trends in Microbiology. 2012.
“Bacterial linguistic communication and social intelligence” Eshel Ben-Jacob, Israela Becker, Yoash Shapira, Herbert Levine. Trends in Microbiology. 2004
Estratégias para tempos adversos.
Em tempos adversos é comum haver muitas mudanças e isto não acontece somente com os humanos que saem as ruas, fazem guerra ou revolução. Na natureza observamos inúmeras estratégias dos seres vivos para superar tempos difíceis, algumas bastante chocantes do ponto de vista humano, tais como infanticídio ou canibalismo. E no mundo microscópio não é diferente.
Se você pensa que as bactérias morrem pacíficas quando as condições não são favoráveis você está bastante enganado. Até mesmo para estes minúsculos seres unicelulares a vida pode ser complexa e cheias de decisões difíceis e estressantes a serem tomadas. Existem diversas estratégias para lidar com tempos difíceis, tais como escassez de alimentos e danos ao DNA. Uma das estratégias mais radicais e mais utilizadas quando a coisa está realmente feia é formar esporos, uma resposta celular bastante complexa que envolve a ativação de mais de 500 genes ao longo de aproximadamente 10 horas. Este processo termina com a morte da célula mãe e a formação de uma célula filha dormente com a capacidade de resistir a situações extremas como calor, radiação e presença de substancias químicas. Por outro lado, durante este tempo o esporo não pode tirar vantagem imediata de situações favoráveis para se reproduzir.
Esporulação é uma tomada de decisão bastante complexa e que não se inicia simplesmente com a escassez de alimentos, mas é resultado de uma série de passos que podem ser descritas como decisões celulares sobre como lidar com o stress presente. Envolve, por exemplo, uma comunicação entre as bactérias da mesma colonia por um mecanismo chamado de quorum sensing. Além disto, antes da esporulação, comumente as bactérias tentam outras táticas como ativar um flagelo para buscar alimentos, secretar antibióticos e outras substancias na tentativa de destruir outros micróbios competidores. As células também checam uma série de condições internas antes de decidir esporular tais como a integridade do DNA cromossomal.
Mesmo quando a maior parte da colonia decide por esporular, o material gerado pela lise da membranas das células que esporularam são aproveitados por outras células da colonia que, no caminho para esporular, entram no estado chamado de competência, que consiste em abrir poros na membrana para facilitar a entrada de DNA exógeno, que pode ser utilizado para reparo do DNA e eventualmente como fonte de informação genética que as ajudará a resistir ao momento. Há ainda um outro caminho mais radical tomado por algumas células da colônia que consiste em secretar alguns fatores antibacterianos que faz com que células irmãs fiquem incapazes de esporular, causando inclusive a lise de sua membrana, em uma espécie de canibalismo. Interessante notar que, sobre a perspectiva de teoria de jogos, a estratégia de canibalismo seria predominante sobre a estratégia de entrar em competência, o que não é observado. Isto indica que possivelmente as células em competência são imunes ao canibalismo, mas não sabemos ainda se é este o caso.
Modelos teóricos/moleculares para este tipo de tomada de decisões normalmente consistem na integração de módulos gênicos formados principalmente por circuitos regulados por diversos fatores de transcrição e micro RNAs. Estes tipos de sistemas gênicos também desempenham um papel importante no desenvolvimento embrionário e de células cancerígenas, motivo pelo qual tem se dado muita atenção a este tipo de estudo.
Referencias:
Aventuras em Biologia Sintética
Eu considero os quadrinhos como uma das formas mais interessantes de se narrar uma história. São também uma ótima forma de divulgar ciência de uma maneira didática e divertida.
Drew Endy um dos pais da biologia sintética (e do Registry of Parts), juntamente com Isadora Deese (ambos do MIT) elaboraram o quadrinho Adventures in Synthetic Biology. E tem mais: o quadrinho foi publicado no website da prestigiada revista Nature.
Confira Adventures in Synthetic Biology e aprenda de maneira didática o que são biobrick, PoPs, entre outros conceitos básicos.
Links possivelmente interessantes:
- Bastidores da produção do quadrinho
- The cartoon guide to genetics
- TED talk do Jorge Cham, criador do sensacional PhD Comics
Crowdfunding: o “faça você mesmo” do financiamento
O movimento DIY (Do It Yourself) é lindo. É quase uma coisa utópica: você quer fazer alguma coisa? Aprende, vai lá e faz, oras! Basta ter aquela ideia que não te deixa dormir, ficar obcecado por ela, ser um pouco otimista e deixar a preguiça de lado. Perfeito, mas… Onde arrumar a grana para seus planos?
Regras do Jogo
Tudo bem, as coisas não são tão fáceis assim. Vivemos num mundo capitalista e as regras do jogo te impedem de colocar uma bela ideia em prática se você não tiver dinheiro. O grande problema na maioria das vezes é justamente esse: o capital inicial. Muitas ideias legais e empreendimentos não saem do papel porque o suporte para esse tipo de coisa não é algo trivial de se conseguir e os riscos são altos dependendo do caso. Por exemplo, um dos grandes problemas que enfrentamos para arranjar financiamento para a participação do iGEM foi a falta de apoio público à iniciativas tecnológicas de pequeno porte (talvez seria mais correto chamar de “mini porte”): ou as chamadas de projeto davam dinheiro demais ou não davam dinheiro nenhum. Se fôssemos uma empresa, não nos encaixaríamos em nenhuma categoria de investimento, o que não significaria que a empresa poderia gerar menos ou mais retornos que as de categorias de investimento maiores.
Mas aí você pode perguntar: “Então porque vocês não pediram dinheiro a mais mesmo?”. É porque esse tipo de apoio deve ter todos seus gastos todos justificados, afinal é um investimento! Se seu empreendimento não demanda coisas o suficiente ele acaba morrendo de fome.
 “Toc-Toc”-“Olá senhor, gostaria de investir no meu empreendimento?”
“Toc-Toc”-“Olá senhor, gostaria de investir no meu empreendimento?”
Mas calma lá também, isso não significa que para colocar sua ideia em prática será preciso ser um missionário do empreendedorismo e bater de porta em porta pedindo financiamento… É quase isso.
A diferença é que, se quiser fazer um belo DIY, terá que bater na porta de fundos de investimento privados. Eles são menos inflexíveis que os fundos públicos (como o BNDES, FINEP e etc) em termos de linhas de financiamento, contudo são um pouco mais sisudos: esses fundos são controlados pelo mercado e o grande objetivo é o retorno do investimento, sem envolver questões sociais e culturais do projeto; a não ser que você encontre um fundo supimpa que se preocupe com isso. Se você conseguir conquistar os empresários, eles deixarão o jogo mais fácil para você colocar seu sonho em prática.
O Nanoempreendedorismo
O movimento DIY não precisa de tanto dinheiro assim para se preocupar com fundos de investimento, a não ser que você queira deixar a brincadeira do “faça você mesmo” mais séria depois. Afinal para fazer um DIY que se preze, os materiais devem ser baratos e fáceis de se encontrar. Só que isso gera um problema semelhante a um empreendimento de “mini porte”: você não vai precisar (provavelmente) e não vai conseguir sozinho 10.000 ou 20.000 dólares; o que você vai precisar para projetos de garagem é da ordem de 500 a 2000 dólares. É pouco provável – devido ao porte do empreendimento – que exista um fundo de investimento que se interesse em apoiar (tudo bem, vai que você consegue!) o seu “faça você mesmo” (apesar desses valores ainda serem uma considerável graninha!). Uma categoria de investimento para “nano-empreendedores” seria mais improvável ainda: além de ser uma grande mistura de hobby com negócios – o que poderia estragar toda a ideia divertida de DIY – os riscos de investimento seriam muito altos (apesar de estar em jogo “pouca” grana).
Crowdfunding: o poder da internet em suas mãos
Como deu para perceber, apesar do dinheiro ser importante, não diria que ele é o limitante para sua ideia (apesar de aumentar pra caramba a sua moral e seu respeito social), seria mais um entrave. O essencial mesmo é conhecer pessoas. Pessoas certas na hora certa principalmente, isso sim é que é difícil. Isso vai economizar muito tempo e sola de sapato. Mas diminuindo ainda mais o tamanho das coisas, pode ser que nem sapatos sejam necessários: apenas uma conexão com a internet já basta (a internet te dá super-poderes). É assim que funciona o Crowdfunding, o “financiamento pela multidão”. É o mesmo modelo que a Wikipedia, a Avaaz e vários programas e aplicativos gratuitos que vivem de doações usam para conseguir existir.
Como sempre, as coisas legais chegam no Brasil depois de já fazerem muito sucesso “lá fora”, mas já existem iniciativas muito legais
como a Crowdfunding Brasil. Contudo, dá para pegar a onda dos gringos através de alguns sites muito legais:
RocketHub
 Disponível para usuários de todo o mundo. Funciona como todos os outros: você cria uma página para as pessoas contribuírem online, estabelece o quanto você precisa para sua ideia e dá um prazo para levantar a grana. Ajudando com diferentes valores existem diferentes categorias de “recompensas” que o contribuidor ganha. O que o RocketHub ganha com tudo isso? 4% de tudo arrecadado, para financiamentos dentro do prazo ou não. Eles recomendam essa correção nos valores antes de realizar a campanha.
Disponível para usuários de todo o mundo. Funciona como todos os outros: você cria uma página para as pessoas contribuírem online, estabelece o quanto você precisa para sua ideia e dá um prazo para levantar a grana. Ajudando com diferentes valores existem diferentes categorias de “recompensas” que o contribuidor ganha. O que o RocketHub ganha com tudo isso? 4% de tudo arrecadado, para financiamentos dentro do prazo ou não. Eles recomendam essa correção nos valores antes de realizar a campanha.
Kickstarter
Diferentemente do RocketHub, aqui o negócio é tudo ou nada: consegiu financiar no prazo? O dinheiro é seu. Não conseguiu? É do Kickstarter. É o site de Crowdfunding mais famoso, mas por enquanto não é de acesso mundial.
IndieGoGo
 É bem parecido com o RocketHub: também é uma plataforma mundial e cobra 4% de tudo arrecadado. A única diferença é que se você não conseguir levantar a quantia no prazo, a porcentagem que fica para o site é maior: 9%.
É bem parecido com o RocketHub: também é uma plataforma mundial e cobra 4% de tudo arrecadado. A única diferença é que se você não conseguir levantar a quantia no prazo, a porcentagem que fica para o site é maior: 9%.
 O único site de crowdfunding tupiniquim é o LET’S. Ainda não é tão bombado como os outros, mas com as políticas de divulação do site e do formato de financiamento de crowdfunding, espera-se que no futuro seja uma opção tão boa quanto as estrangeiras. A LET’S sempre fica com 5% do valor arrecadado caso o pagamento seja via Paypal.
O único site de crowdfunding tupiniquim é o LET’S. Ainda não é tão bombado como os outros, mas com as políticas de divulação do site e do formato de financiamento de crowdfunding, espera-se que no futuro seja uma opção tão boa quanto as estrangeiras. A LET’S sempre fica com 5% do valor arrecadado caso o pagamento seja via Paypal.
Existem muitos projetos impressionantes e interessantíssimos nesses sites, com diferentes graus de ambição. Para nossa iniciativa de participação no iGEM 2012, além de corrermos atrás de financiamentos por patrocínios de empresas, criamos um projeto lá no RocketHub! Até o presente momento conseguimos levantar incríveis 385 dólares (uhul! Estamos ricos!). Sinta-se à vontade para olhar nossas recompensas e contribuir como quiser! Conte para todo mundo também!
Enfim: fica aí a dica. Principalmente se você curte DIY Biology e tem ideias bem legais que quer colocar em prática. Só não vai criar um problema de Biossegurança hein!
SynbioBrasil no Science Blogs!

Após passar por um processo de seleção junto à outros ótimos blogs de ciência brasileiros o SynbioBrasil foi convidado a participar da rede ScienceBlogs Brasil (SBBr), uma das mais importantes e influentes comunidades de blogs sobre ciência do Brasil!
Figuram na SBBr grandes blogueiros da ciência e apoiadores da nossa iniciativa, como Átila Iamarino, do blog Rainha Vermelha, e Carlos Hotta, do Brontossauros em Meu Jardim, isso sem falar nos vários outros ótimos blogs (como o Massa Crítica; Chapéu, Chicote e Carbono-14; e Ciência e Ideias).
Todos nós do time de colaboradores, tanto dos que escrevem no blog, como os que participam da iniciativa para o iGEM 2012, recebemos com grande honra essa oportunidade e esperamos poder continuar nosso trabalho de divulgação científica sobre Biologia Sintética, iGEM e DIY Biology, seguindo nossa filosofia para uma ciência mais aberta, acessível e democrática, que estimule a interdisciplinariedade e integração de universidades, institutos e iniciativas privadas na inovação da pesquisa brasileira através da biologia sintética.
Particularmente, é muito bonito poder fazer parte dessa nova geração de blogs científicos, ainda mais podendo participar da comunidade de blogs que leio desde a adolescência e que com certeza influenciou muitas decisões da minha vida, servindo principalmente para alimentar e incentivar minha curiosidade: um dos bens mais preciosos que alguém pode ter! Obrigado! 🙂
ScienceBlogs! Lá vamos nós!
Canal do SynbioBrasil no LiveStream!
Já está feito o nosso canal para transmissão ao vivo pela internet das reuniões do Clube de Biologia sintética pelo LiveStream, um site muito legal de streamming ao vivo pela internet que qualquer um pode criar.
Agora com esse novo recurso é possível interagir ao vivo com a apresentação, além de ser possível conferir os vídeos da apresentação depois no próprio canal.
Confira nosso canal clicando na imagem acima ou na barra aqui à direita com os links do SynbioBrasil.
Veja os horários das reuniões na página do clube de biologia sintética no cabeçalho do site para saber quando o canal estará online!
Esperamos sua participação por lá! Até!
Inside iGEM: Eventos Passados
 A competição surgiu oficialmente em 2005, resultado do período de atividades independentes (AIP) do MIT, em que a universidade, assim como muitas outras, abre as portas para cursos fora do período letivo, na época de férias. Respectivamente nos anos de 2003 e 2004, times de estudantes do próprio MIT desenvolveram osciladores biológicos com proteínas-repórter fluorescentes (como vimos aqui no blog) e sistemas genéticos para criar padrões celulares (Imagem abaixo) como os de “pontinhos” (chamados de polka dots) e de “alvo” (chamados de bull’s eye formation).
A competição surgiu oficialmente em 2005, resultado do período de atividades independentes (AIP) do MIT, em que a universidade, assim como muitas outras, abre as portas para cursos fora do período letivo, na época de férias. Respectivamente nos anos de 2003 e 2004, times de estudantes do próprio MIT desenvolveram osciladores biológicos com proteínas-repórter fluorescentes (como vimos aqui no blog) e sistemas genéticos para criar padrões celulares (Imagem abaixo) como os de “pontinhos” (chamados de polka dots) e de “alvo” (chamados de bull’s eye formation).
Ainda em 2004, junto com as atividades do AIP, foi criada a “Summer Competition”, uma competição à lá iGEM mas com apenas cinco universidades participantes, todas norte-americanas:Boston University, Caltech, o próprio MIT, Princeton University e a University of Texas at Austin; foi a primeira verdadeira competição de biologia sintética.O grande destaque dessa competição pré-iGEM foi a universidade do Texas, que segundo a página da competição criou o primeiro filme fotográfico biológico do mundo, o “Coliroid Film” (Imagem acima).
Esse evento acabou impulsionando Randy Rettberg, Tom Knight e Drew Endy a fundarem em 2005 o evento internacional que conhecemos hoje, com cerca de 13 times, com os mais variados projetos.
Destaques
Desde o período de atividades independentes de 2004 até 2010, 434 times e projetos já participaram da competição, com idéias e soluções criativas e inovadoras para problemas da humanidade, o que torna difícil apresentar todos os destaques das competições além dos finalistas e vencedores do Biobrick Trophy.
Logo no início, em 2005, enquanto a competição ainda estava engatinhando e os critérios de julgamento ainda não estavam bem consolidados, não houve um BioBrick Trophy e houveram até algumas premiações um tanto não-convencionais em comparação aos iGEM’s que se seguiram, como o “Best ‘Show Must Go On’ Moment” dado à Princeton, o “George W.Bush Geography Award” (provavelmente uma brincadeira envolvendo as gafes geográficas do ex-presidente norte-americano) dado à universidade de Zurique, e o “Best Project Name” dado à universidade de Toronto devido ao trocadilho com o nome do time e o projeto: “Cell-See-US”, que desenvolveu um tipo de termômetro com bactérias, fazendo uma analogia à unidade Celsius de medida de temperatura.
Alguns destaques interessantes da competição desse ano foram:
 Harvard: Criaram componentes que escrevem e apagam para um “caderno de desenho bacteriano”, utilizando luz e calor para respectivamente induzir a expressão e degradar proteínas repórter em uma placa.
Harvard: Criaram componentes que escrevem e apagam para um “caderno de desenho bacteriano”, utilizando luz e calor para respectivamente induzir a expressão e degradar proteínas repórter em uma placa.
 UCSF (Universidade da Califórnia, São Francisco): Desenvolveram termômetro biológico programável, apesar de início, segundo a wiki de 2006, ambicionarem apenas um detector biológico de temperatura. Ganhador do primeiro “Best Device Award” do iGEM.
UCSF (Universidade da Califórnia, São Francisco): Desenvolveram termômetro biológico programável, apesar de início, segundo a wiki de 2006, ambicionarem apenas um detector biológico de temperatura. Ganhador do primeiro “Best Device Award” do iGEM.
 Penn State: Construiram um mecanismo genético de controle quimiotáxico usando BioBricks. Ganhador do “Best Brick Award”.
Penn State: Construiram um mecanismo genético de controle quimiotáxico usando BioBricks. Ganhador do “Best Brick Award”.
| Terceiro Lugar | Segundo Lugar | Vencedor | |
| 2010 |  |
 |
 |
| 2009 |  |
 |
 |
| 2008 |  |
 |
 |
| 2007 |  |
 |
 |
| 2006 |  |
 |
 |
Durante a avaliação dos projetos são escolhidos 5 finalistas, dentre eles figuram com frequência a Imperial College of London e UC Berkeley, além das famosas como Cambridge e Harvard. Fama também o que a Universidade da Eslovênia – do modesto país europeu – tem ganhado com essa competição: três vezes campeã e uma vez entre os 5 finalistas em 2007; contrariando a expectativa de reproduzir o ranking das melhores universidades do mundo, o que é confirmado pela forte presença das universidades asiáticas como a de Peking e a USTC.
É nesse ambiente plural e rico cientificamente (e porque não culturalmente?) em que florescem os projetos em biologia sintética mais impressionantes e competitivos todos os anos, mas muita coisa mudou desde 2006, e é sobre isso que vamos falar no próximo post da série Inside iGEM: O Futuro e Hoje.
Por falar em futuro, e o país do futuro? Onde entra nessa história!? Nós aqui da usplândia não somos os primeiros a pensar no iGEM. Veremos também a participação tupiniquim representada pela Unicamp em 2009 e agora em 2011. Então, até o próximo post!
Bactéria com substância química tóxica incorporada ao DNA
 Uma equipe internacional de pesquisadores alcançou sucesso em obter uma bactéria que possui o DNA no qual a base timina foi substituída por 5-Chlorouracil, uma substância sintética tóxica para outros organismos.
Uma equipe internacional de pesquisadores alcançou sucesso em obter uma bactéria que possui o DNA no qual a base timina foi substituída por 5-Chlorouracil, uma substância sintética tóxica para outros organismos.
O trabalho experimental foi baseado numa tecnologia única desenvolvida por Marlière e Mutzel, possibilitando a evolução dirigida de organismos sob condições estritamente controladas. Populações de bactérias foram cultivadas por períodos prolongados na presença de uma substância química tóxica – no caso, a 5-Chlorouracil – a níveis que não provocam a morte, dessa forma selecionando as células tolerantes a altas concentrações dessa substância.
Em resposta ao surgimento dessas mutantes na população, a concentração do 5-Chlorouracil no meio de cultura foi aumentada para manter a pressão seletiva constante. Esse procedimento de evolução a longo prazo foi aplicado para adaptar E. coli geneticamente modificadas incapazes de sintetizar timina para crescerem num meio com concentrações crescentes de 5-Chlorouracil. Após 1000 gerações aproximadamente, descendentes da linhagem inicial foram analisados, sendo que estes utilizaram o 5-Chlorouracil como substituto da timina. Análises subseqüentes do genoma revelaram numerosas mutações no DNA das bactérias adaptadas. A contribuição dessas mutações para a adaptação das células será objeto de estudos posteriores.
Além do interesse óbvio dessa mudança radical na química de sistemas vivos para a pesquisa básica, os cientistas também consideram os resultados de seu trabalho relevantes para xenobiologia, um ramo da biologia sintética. Essa nova área das ciências da vida tem como objetivo a geração de novos organismos não encontrados naturalmente, com características metabólicas otimizadas para, por exemplo, a produção de modos alternativos de energia e síntese de produtos químicos de alto valor. Assim como os transgênicos ou organismos geneticamente modificados, essas bactérias sintéticas são vistas como potencial ameaça para ecossistemas naturais quando liberadas do laboratório, seja pela competição com organismos selvagens ou através da difusão do “DNA sintético”.
É óbvio que mesmo tentando conter, não é possível evitar absolutamente que seres vivos geneticamente modificados entrem em contato com hábitats naturais, assim como os isótopos radioativos que escapam para as redondezas de uma região de usina nuclear. Entretanto, organismos sintéticos assim como os do presente trabalho – que dependem da disponibilidade de determinadas substâncias para sua proliferação ou que incorporam compostos químicos não naturais em seu DNA – não tem condições de competir com animais selvagens nem trocar DNA com eles.
Referências
Marlière, P., Patrouix, J., Döring, V., Herdewijn, P., Tricot, S., Cruveiller, S., Bouzon, M. and Mutzel, R. (2011), Chemical Evolution of a Bacterium’s Genome. Angewandte Chemie International Edition, 50:
n/a. doi: 10.1002/anie.201100535
Thymine Replacement Directs Bacterium DNA Evolution
[twitter-follow screen_name=’MnlimasBio’]
SB 5.0 – Encontro Internacional de Biologia Sintética e um breve histórico
Em julho do ano passado, cientistas do J. Craig Venter Institute “criaram” a primeira célula bacteriana sintética e auto-replicante, controlada por um genoma quimicamente sintetizado e o anúncio foi feito em maio (veja o vídeo abaixo). A equipe sintetizou um cromossomo de 1,08 milhões de pares de bases a partir de um genoma modificado de Mycoplasma mycoides. A célula sintética recebeu o nome de Mycoplasma mycoides JCVI-syn1.0 e é a prova de que genomas podem ser desenhados (no sentido de designed) em computadores, depois sintetizados quimicamente em um laboratório e inseridos em uma célula, de forma a produzir uma nova célula auto-replicante controlada apenas por um genoma sintético.
[youtube=http://www.youtube.com/watch?v=QHIocNOHd7A&feature=player_embedded]
A notícia se espalhou pelo mundo em pouco tempo, ganhando fama, criando polêmica e dividindo opiniões a respeito dos riscos e benefícios potenciais de sua descoberta. Em resposta, o Presidente Barack Obama pediu para a “Presidential Commission for the Study of Bioethical Issues” avaliar o desenvolvimento e a ética da biologia sintética e tecnologias emergentes, de forma a maximizar os benefícios e minimizar os riscos. A Comissão contou com o engajamento de cientistas, engenheiros, profissionais ligados à ética, ciências sociais.
Agora em junho, cerca de 700 pessoas de 30 países participaram do 5ª Encontro Internacional de Biologia Sintética na Universidade de Stanford, que pelo que me parece é o mais importante nessa área. Havia cientistas superstars, estudantes, biólogos DIY, engenheiros, biólogos tradicionais, entre outros. O pessoal que segue nosso twitter ou a página do facebook ficou sabendo que a conferência foi transmitida ao vivo pela internet e viu como esse pessoal está batalhando para dar os próximos passos na synbio.
A criação da célula sintética abre portas para um futuro no qual biólogos sintéticos poderão “redesenhar” (redesign) células vivas para realizarem quaisquer tarefas desejadas. A maior parte das pesquisas atuais tem focado em bactérias que executam atividades semelhantes àquelas que elas já fazem, por meio de processos e materiais que se parecem com aqueles utilizados naturalmente. Exemplos são bactérias produtoras de combustíveis.
Os cientistas têm as ferramentas necessárias para editar uma sequência genética existente em um computador, usar máquinas sintetizadoras de DNA para fabricar os fragmentos e uni-los em laboratório (Esse é só um de vários caminhos que biólogos sintéticos estão tomando.) Mas ainda é difícil predizer o que as células farão depois de serem alteradas. Pesquisadores enfrentam desafios porque as células tem um “desejo natural” de crescerem e viverem à sua maneira, mas elas precisam aprender a produzir algo útil de uma forma eficiente.
Um dos maiores obstáculos reside na criação e montagem dos fragmentos de DNA que codificam para uma função particular e são sintetizados no laboratório. Fabricar esse DNA ainda é caro e requer tempo, e qualquer outra mudança que seja necessária demanda ainda mais tempo e dinheiro.
“Algumas sequências são sintetizadas em dois meses”, enquanto outras podem nem mesmo serem feitas, por razões ainda não entendidas, disse Reshma Shetty, co-fundadora do Ginkgo Bioworks, uma companhia que monta partes de DNA.
Pamela Silver, uma professora de biologia de sistemas da Universidade de Harvard, acredita que os biólogos do futuro poderão sentar na frente de um computador, planejar um experimento, e ter o DNA no dia seguinte. Para que a biologia sintética cumpra sua promessa, a síntese de DNA deve ser barata, rápida, previsível e acurada, além de ser disponível a todos, incluindo pesquisadores cujos laboratórios não tem equipamentos ou recursos apropriados. Felizmente, o custo da síntese de DNA, assim como o do sequenciamento de DNA, vem caindo rapidamente.
Retirado e adaptado de: “What’s the Future of Synthetic Biology?“ por Katherine Bourzac
Para Saber Mais:
The Promise of Syn Bio (version 5.0)
Immaculate creation: birth of the first synthetic cell
[twitter-follow screen_name=’MnlimasBio’]










