FAQ #8 Como ver se a transformação gênica deu certo?
Já colocamos nossos plasmídeos nos bichinhos, mas ainda não acabou. Essa é a hora de saber se tudo que fizemos até aqui deu certo!
Primeiro, colocamos eles na placa de seleção com antibiótico e só aqueles que realmente incorporaram o plasmídeo vão sobreviver porque ganharam um gene amigo de resistência ao antibiótico. Agora pegamos uma parte das células sobreviventes e fazemos um teste usando a PCR (você lembra dessa técnica, né?).
Como testar com a PCR?! Roubamos os plasmídeos dessas células e tentamos multiplicar o pedaço de DNA que foi grudado nele adicionando primers, nucleotídeos e enzimas sob aquecimento e desaquecimento. Se o nosso plasmídeo modificado tiver sido incorporado pelos micro-organismos haverá a multiplicação do pedacinho e podemos vê-los em uma eletroforese. Caso contrário, nada foi copiado e nada vai aparecer na eletroforese. Então temos o sinal de que alguma coisa não deu certo por aqui e as chances de erros deste teste são bem pequenas.
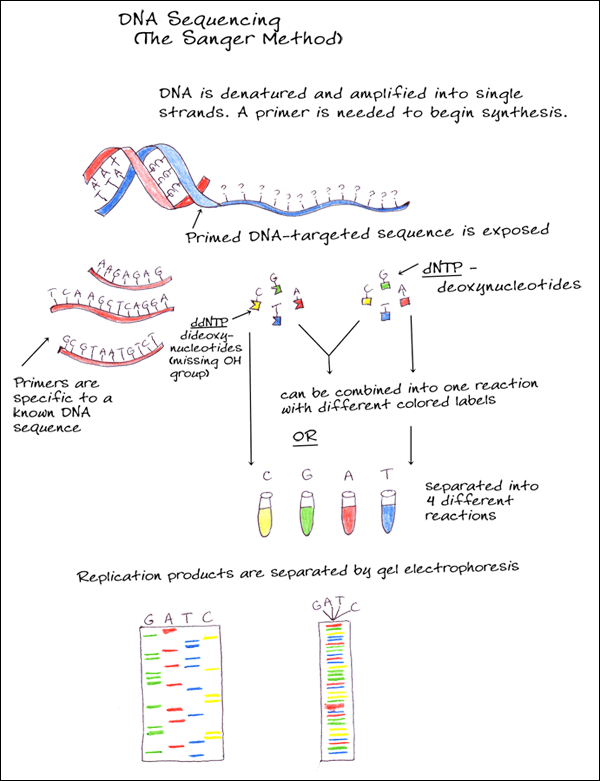
Depois vamos para um teste melhor ainda, o sequenciamento. Este teste diz exatamente qual é a sequência de pares de base da molécula do plasmídeo e acabam de vez suas dúvidas se a transformação deu certo ou não! O sequenciamento começa parecendo um processo de duplicação normal de DNA. Mas além de cadeias molde, enzimas e nucleotídeos normais, há nucleotídeos especiais sintetizados (ddNTP’s) que possuem duas boas propriedades: emitem luz e interrompem o prologamento da cadeia a partir de onde foram adicionados. Então, imagine uma molécula de DNA que possui um par de base A-T em um determinado comprimento e considere que a base A pertence à cadeia molde e a base T à cadeia complementar. Se esta base T for um nucleotídeo especial temos como identificar “quem é” e “qual sua posição” na cadeia, pois ele emitirá uma cor específica para Timina e o comprimento de sua cadeia está interrompido na posição exata (nº 2 em verde). 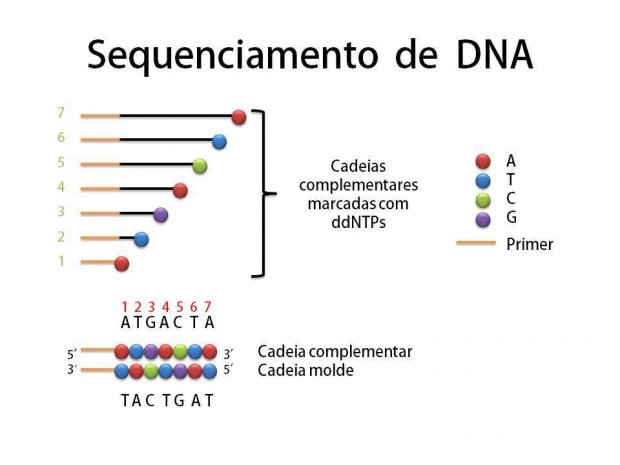 Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Para saber mais sobre sequenciamento genético, clique aqui.
Por Otto Heringer e Viviane Siratuti.
FAQs da Bioengenharia – Introdução
Preocupados com os novos amantes da biologia sintética, estamos procurando materiais de introdução no assunto e encontramos algumas coisas interessantes! 🙂
Por isso vamos colocar uma sequência de posts explicativos, partindo de uma visão geral e seguindo por perguntas mais específicas. É um pequeno tutorial-FAQ sobre alguns fundamentos que quando entendidos ajudam bastante a entender algumas de nossas discussões e outros posts deste blog. Serve para ajudar principalmente quem não é da área de biológicas ou quem ainda não se aprofundou muito neste assunto! Se continuarem com dúvidas, perguntem tá?!
Lembrem-se de ativar as legendas dos vídeos, caso necessitem! Todos possuem legendas originais em inglês e alguns em português. Você pode colocá-las traduzidas quando só houver em inglês (não muito recomendado, rs). Nos vídeos do JOVE existe a opção de colocar os textos em português. Se você não sabia que o YouTube fazia isso por você, dá uma olhada aqui (dica: barrinha inferior do vídeo).
Antes de mais nada, você sabe o que são e quais as relações entre nossas principais moléculas orgânicas (DNA, RNA e proteínas), certo? Se sua resposta é não, assista um desses vídeos!
[youtube_sc url=”http://www.youtube.com/watch?v=h3b9ArupXZg#t=0″]
[youtube_sc url=”http://www.youtube.com/watch?v=tMr9XH64rtM”]
Indo além da Estrutura
OK, agora vamos além desses conceitos básicos dos vídeos. Mais que entender a estrutura e a dinâmica dessas biomóleculas, como os cientistas a alteram? Em linhas gerais, como se faz para modificar geneticamente um organismo?
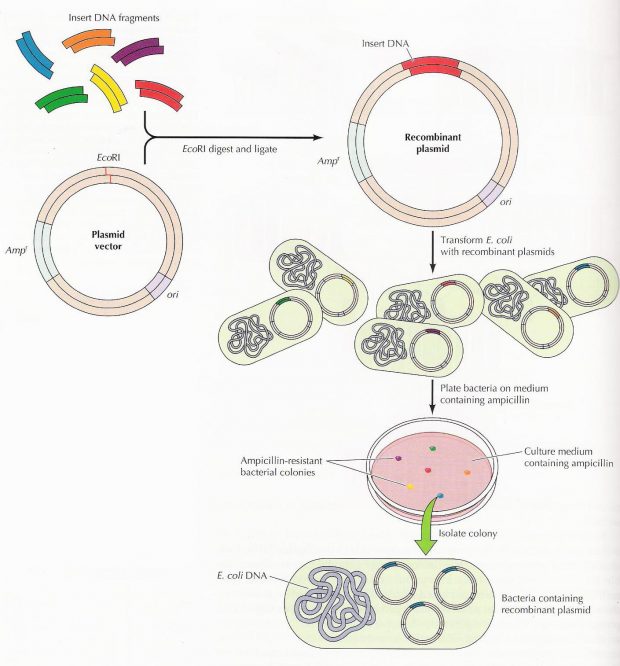
Vamos lá! Primeiro pegamos um pedacinho codificante de DNA (gene) de outro organismo que tenha certa característica que desejamos. Para isso, extraímos o DNA e depois multiplicamos só esse pedaço que nos interessa através de uma técnica chamada PCR (afinal, a eficiência das transformações é pequena e quanto mais material melhor). Precisamos também extrair e abrir os plasmídeos (DNA circular) do organismo escolhido para receber este gene e assim receber as novas características (já que o plasmídeo é nosso principal veículo de introdução dos genes nas células). Para abrir estes plasmídeos usamos enzimas de restrição que cortam as moléculas de DNA em lugares específicos e chamamos isso de digestão. Depois que tivermos vários plasmídeos e genes, podemos grudá-los com a ajuda de enzimas de ligação. E então, para saber se nosso novo plasmídeo deu certo usamos uma técnica de separação por carga e tamanho de molécula, a eletroforese em gel. Nela, as moléculas se acumulam em certas posições que nos dizem seus tamanhos (mergulhadas em um polímero) e assim podemos identificar aquelas que correspondem ao que estimamos. Além disso, fazemos testes com a técnica PCR e o sequenciamento genético. Se os testes disserem que tudo que fizemos deu certo, recuperamos o material e colocamos nossos novos plasmídeos nos organismos, geralmente utilizando choque térmico ou elétrico. Esse passo, introdução de plasmídeos, é a chamada transformação. Mas para selecionarmos somente aqueles organismos que realmente foram transformados usamos antibióticos que matam as células que não ganharam os nossos novos plasmídeos, já que não possuem os genes de resistência que foram colocados neles. E no final, cada célula que sobreviveu se multiplicará, formando colônias de organismos geneticamente modificados. Agora dá uma olhada no esquema de novo e vê se entendeu até aqui!
Por Otto Heringer e Viviane Siratuti.
Especulações e Biologia Sintética
 Minha vontade de discutir e aprender biologia, especialmente tudo aquilo que envolve a palavra DNA já me acompanha antes mesmo de entrar na faculdade. A solução que encontrei para suprir essa vontade e viagens por esse mundo, até então feitas apenas acompanhado de meu computador foi prestar uma prova de seleção para dar aulas de biologia no cursinho da UNESP.
Minha vontade de discutir e aprender biologia, especialmente tudo aquilo que envolve a palavra DNA já me acompanha antes mesmo de entrar na faculdade. A solução que encontrei para suprir essa vontade e viagens por esse mundo, até então feitas apenas acompanhado de meu computador foi prestar uma prova de seleção para dar aulas de biologia no cursinho da UNESP.
Comecei dar aulas em 2012 e o melhor, da matéria que mais gosto, citologia e genética, afinal, eu queria falar de células, DNA, Mendel e não sobre insetos que possuem três pares de patas e um de antenas.
Certo dia, véspera de feriado e com chuva, resolvi passar um filme para discussão. O filme escolhido foi GATTACA. Ao final do filme, um aluno com os olhos arregalados que aparentavam demostrar grande interesse pelo assunto, levanta a mão e pergunta: “Esse tipo de previsão é possível?”
[youtube_sc url=”http://www.youtube.com/watch?v=L8yYjKa36No”]
GATTACA trata-se de um filme hollywodiano de ficção científica, infelizmente não poderemos prever a expectativa de uma pessoa ao nascer, mesmo uma década após a conclusão do Projeto Genoma. Hoje sabemos que os organismos dependem de dois tipos de informação, uma delas é o código genético e a outra são estímulos recebidos do meio ambiente em que vivem, sobre o qual não podemos fazer previsões.
O Projeto Genoma Humano, considerado por muitos a bola de cristal do futuro, foi cercado de promessas e esperanças da mídia e de Wall Street para o lançamento de medicamentos personalizados e previsões sobre riscos de doenças como Alzheimer e cânceres. Tal projeto prometia revolucionar a medicina, ao estilo do filme de ficção científica.
Pouco mais de dez anos já se passaram desde sua conclusão, uma batalha inciada em 1990 travada entre a iniciativa pública e a privada, que chegou ao fim depois de três bilhões de pares de bases sequenciadas e três bilhões de dólares gastos, além de uma briga entre o famoso James Watson e Craig Venter, marinheiro nas horas vagas. Para Nicholas Wade do NYT, o projeto falhou e não trouxe os benefícios tão esperados.
Embora as promessas aguardadas por muitos não chegaram, ele trouxe avanços significativos para a área da biologia. O Projeto Genoma Humano foi um projeto interdisciplinar, contando com a participação de cientistas de diversas áreas como computação, matemática, biologia entre outras, além da colaboração de laboratórios ao redor do mundo, inclusive do Brasil. Ele contribuiu significativamente para o avanço na área de biocomputação e nas técnicas de sequenciamento de DNA, além de permitir a democratização da informação por meio do acesso gratuito ao sequenciamento humano e de diversos outros seres vivos. Genes agora não precisam mais ser estudados de forma individual, hoje dispomos de uma extensa lista de partes biológicas que tem permitido o avanço na área de biologia de sistemas, que promete revolucionar o modo de tratar doenças e o sistema de saúde conhecido atualmente.
Tal fato nos faz refletir sobre as promessas em torno da área de biologia sintética, a qual acredita-se ser capaz de solucionar muitos de nossos problemas atuais como o elevado preço do petróleo, desastres ambientais e uma população cada vez maior para se alimentar. Ao invés de manipular alguns genes de cada vez, como se tem feito a mais de três décadas, graças a descoberta das enzimas de restrição, o campo da biologia sintética vai além e tem como objetivo recodificar e redesenhar circuitos genéticos. Parece não haver limites do que podemos fazer, no entanto tais circuitos não se comportam como circuitos eletrônicos. Sistemas assim costumam ter muita interferência e comportamento imprevisivel, além de estarem sujeitos a mutações. Segundo o cientista Christopher Voigt, “A célula é uma espécie de burrito, tudo dentro dela está misturado” e sujeito a uma variedade de reações bioquímicas, tal complexidade tem limitado o tamanho dos circuitos criados. Drew Endy, um dos pioneiros do ramo, acredita que a biologia sintética só atingirá seu potencial quando os cientistas forem capaz de prever com exatidão como um circuito genético se comportará dentro de uma célula. Para isso tem-se aplicado estratégias de engenharia, como a padronização, dissociação e a abstração.
Outro problema enfrentado é a caracterização das partes biológicas. Embora o Registry of Standard Biological Parts possua mais de cinco mil partes disponíveis, menos da metade tem seu funcionamento confirmado. Caracterizar algumas partes biológicas pode levar anos e não gerar dados para uma publicação. Na cultura do “publique ou pereça”, o que temos é uma grande quantidade de trabalhos, porém com poucos dados reproduzíveis e úteis.
Embora o custo de sintese e sequenciamento tenham caido de forma significativa facilitando o avanço da área, não podemos negar que ainda há muitas pedras no caminho. Nosso conhecimento ainda é limitado mesmo para compreender por completo o comportamento de organismos mais simples como bactérias e fungos unicelulares. Assim como o projeto genoma a biologia sintética também está rodeada por especulação e não podemos dizer onde estaremos em dez anos. Mas uma coisa é certa, aprendemos a ler agora estamos escrevendo código genético.
Referências
- The Promise and Perils of Synthetic Biology – Jonathan B. Tucker and Raymond A. Zilinskas
- Synthetic biology: from hype to impact – Timothy S. Gardner
- Five hard truths for synthetic biology – Roberta Kwok
- Initial impact of the sequencing of the human genome – Eric S. Lander
- http://web.mit.edu/newsoffice/2012/complex-biological-circuit-1007.html
Pesquisadores de Caltech constróem a primeira rede neural artificial de DNA
 Pesquisadores da Universidade de
Pesquisadores da Universidade de
Caltech inventaram um método para construir sistemas de moléculas de DNA cujas interações simulam o comportamento de um modelo matemático simples de redes neurais artificiais.
A inteligência artificial tem sido inspiração para incontáveis livros e filmes, assim como aspiração de inúmeros cientistas e engenheiros. Pesquisadores do Instituto de Tecnologia da Califórnia (Caltech) deram um importante passo na criação da inteligência artificial – não em um robô ou chip de silicone, mas num tubo. Os pesquisadores são os primeiros a fazerem uma rede neural artificial de DNA, criando um circuito de moléculas interagindo e que apresentam lembranças baseadas em padrões incompletos, assim como um cérebro.
“O cérebro é incrível”, disse Lulu Qian, um bioengenheiro de Caltech. “Permite o reconhecimento de padrões de eventos, formação de memórias, tomada de decisões e ações. Nós nos perguntamos, ao invés de uma rede de células neurais conectadas fisicamente, será que uma sopa de moléculas interagindo pode apresentar um comportamento típico do cérebro?” A resposta, como eles mostraram é sim.
Constituída de quatro neurônios artificiais feitos de 112 fitas distintas de DNA, a rede neural foi “treinada” para “reconhecer” quatro cientistas, cujas identidades são representadas por um conjunto específico de respostas para quatro perguntas sim-ou-não.
Depois de pensar em um cientista, um jogador humano fornece um subsistema de respostas que identificam parcialmente o cientista. O jogador conduz as pistas para a rede ao gotejar fitas de DNA correspondentes àquelas respostas dentro do tubo de ensaio. A rede identifica qual era o cientista que o jogador tinha pensado através de comunicação por sinais fluorescentes. Ou, a rede pode “dizer” que havia informação insuficiente para escolher apenas um dos cientistas em sua memória ou que as pistas contradizem aquilo que foi lembrado. Os pesquisadores jogaram esse jogo com a rede utilizando 27 maneiras de responder às perguntas (de um total de 81 combinações possíveis) e ela respondeu corretamente a cada vez.
Essa rede neural de DNA demonstra a habilidade de a partir de um padrão incompleto, descobrir o que ele representa – uma das características de um cérebro. “Nós somos bons em reconhecer coisas. Podemos reconhecê-las olhando para um conjunto de pistas ou características”, disse o co-autor Jehoshua “Shuki” Bruck. A rede neural de DNA faz isso, mas de uma forma mais rudimentar.
Sistemas bioquímicos com inteligência artificial – ou pelo menos uma capacidade básica de tomar decisões – poderia ter poderosas aplicações na medicina, química e biologia, os pesquisadores disseram. No futuro, esses sistemas poderiam operar dentro das células, ajudando a responder perguntas fundamentais da biologia ou diagnosticar uma doença. Processos bioquímicos que podem responder inteligentemente à presença de outras moléculas poderiam permitir que engenheiros produzissem compostos químicos cada vez mais complexos ou construir novos tipos de estruturas, molécula a molécula.
“Embora comportamentos como o do cérebro dentro de sistemas bioquímicos artificiais tenham sido hipotéticos por décadas”, segundo Qian, “eles são muito difíceis de entender e decifrar.”
Os pesquisadores basearam sua rede neural bioquímica em um modelo neuronal simples, chamado linear threshold function. O modelo neuronal recebe sinais de entrada, multiplica-os por um fator positivo ou negativo, e se a soma ultrapassar um certo limiar, o neurônio dispara, produzindo um sinal de saída.
“Este modelo é uma simplificação dos neurônios reais”, disse o co-autor Erik Winfree. “Mas é uma boa simplificação. Tem sido um modelo extremamente produtivo para explorar como o comportamento coletivo de vários elementos computacionais simples pode levar a comportamentos como o de um cérebro, como completar o padrão e memória associativa.”
Para construir a rede neural de DNA, os pesquisadores utilizaram um processo chamado strand-displacement cascade. Anteriormente, a equipe desenvolveu esta técnica para criar o maior e mais complexo circuito de DNA até então, que computava raízes quadradas.
Dentro do tubo de ensaio, o DNA dentro da solução contém moléculas de DNA simples fita e parcialmente dupla-fita. Uma simples-fita pode mudar sua conformação para uma parcialmente dupla-fita, e se suas bases forem complementares, a simples-fita se junta a dupla-fita, deslocando a outra fita da dupla-hélice. A simples fita então atua como
sinal de entrada enquanto a fita deslocada atua como sinal de saída, que pode então interagir com outras moléculas.
Já que eles podem sintetizar fitas de DNA com a sequência que eles queiram, os pesquisadores podem programar essas interações para se comportarem como uma rede de neurônios. Ao ajustar as concentrações de cada fita de DNA na rede, eles podem ensiná-la a se lembrar dos padrões únicos de respostas sim-ou-não que pertencem a cada um dos quatro cientistas. Diferentemente de algumas redes neurais artificiais que podem aprender diretamente a partir de exemplos, os pesquisadores usaram simulações computacionais para determinar os níveis de concentração molecular necessários para implantar memória na rede neural de DNA.
Enquanto este experimento mostra a promessa de se criar redes de DNA que podem, em essência, pensar, esta rede neural é limitada. O cérebro humano consiste de 100 bilhões de neurônios, mas criar uma rede com apenas 40 desses neurônios baseados em DNA – dez vezes maior que a rede demonstrada – seria um desafio, de acordo com os pesquisadores. Além disso, o sistema é lento; a rede no tubo de ensaio demorou 8 horas para identificar cada cientista misterioso. As moléculas também são incapazes de descolar e parear com uma fita diferente de DNA, então o jogo pode ser jogado apenas uma vez. Talvez no
futuro, uma rede neural bioquímica poderia aprender a melhorar sua performance depois de muitos jogos repetidos, ou aprender novas memórias ao encontrar novas situações. Desenvolver redes que operam dentro do corpo – ou até mesmo dentro de uma célula ou placa de Petri – também é um longo caminho, já que fazer esta tecnologia operar in vivo traz desafios completamente diferentes.
Além de desafios tecnológicos, engenheirar estes sistemas poderia também fornecer uma percepção da evolução da inteligência. “Antes de o cérebro ter evoluído, organismos unicelulares também eram capazes de processar informação, tomar decisões e agir em resposta ao meio
ambiente,” Qian explicou. A fonte de comportamentos complexos deve ter sido uma rede de moléculas fluindo pela célula. “Talvez o cérebro altamente desenvolvido e a inteligência limitada vista nas células unicelulares compartilhem um modelo computacional similar que é programado em diferentes substratos.
“Nosso artigo pode ser interpretado como uma demonstração simples de princípios neuro-computacionais nos níveis moleculares e intracelulares”, de acordo com Bruck. “Uma interpretação possível é que esses princípios sejam universais no processamento de informações biológicas.
Vejam os vídeos explicativos, em inglês, feitos pelos próprios pesquisadores. Ficou bem dinâmico!
Referências
Neural network computation with DNA strand displacement cascades.
http://www.nature.com/nature/journal/v475/n7356/full/nature10262.html
http://www.eurekalert.org/pub_releases/2011-07/ciot-crc072011.php
[twitter-follow screen_name=’MnlimasBio’]
Dispositivos sintéticos: osciladores
![]()
Um oscilador eletrônico é um circuito elétrico que produz um sinal eletrônico repetitivo, frequentemente uma onda senoidal ou uma onda quadrada. Eles são amplamente utilizados em aparelhos eletrônicos, incluindo aparelhos de transmissão de rádio e televisão.
Stricker e colaboradores (2008) descrevem a construção de um oscilador sintético biológico. Para isso, uma construção muito interessante foi utilizada, baseada em um promotor híbrido Plac/ara. Este promotor possui dois operadores que respondem a dois sinais: ele é ativado por AraC na presença de arabinose e é reprimido por LacI na ausência de IPTG. Seguido a este promotor foi clonado os genes araC, lacI e o gene codificador da proteína luminescente GFP. Um pulso de IPTG foi utilizado para sincronizar as células, dessa maneira, AraC é traduzido resultando na ativação do sinal luminescente de GFP. Por outro lado, AraC ativa também a expressão de lacI resultando na repressão de araC e gfp, cessando o sinal luminescente. Como Lac I causa a repressão do próprio lacI, este acaba se reprimindo e um novo ciclo se reinicia (ver figura abaixo).
 O período de oscilação pode ser ajustado modificando a concentração do indutor, por ex, podendo variar o período de 13 a 58 minutos. Na Figura acima o gráfico apresenta um período de 40 minutos. O mais interessante, para mim, é que o tempo de geração das bactérias do experimento varia de cerca 22 a 27 min, assim, a informação e o sincronismo contido no oscilador passa de geração em geração.
O período de oscilação pode ser ajustado modificando a concentração do indutor, por ex, podendo variar o período de 13 a 58 minutos. Na Figura acima o gráfico apresenta um período de 40 minutos. O mais interessante, para mim, é que o tempo de geração das bactérias do experimento varia de cerca 22 a 27 min, assim, a informação e o sincronismo contido no oscilador passa de geração em geração.
Foi verificado que a indução da oscilação foi muito rápida (cerca de 5 min) e inicialmente bem sincronizada, analisando cada célula individualmente. A amplitude da oscilação foi caindo com o progresso do experimento, devido a dessincronização gradual de cada célula da colônia, como já era de ser esperar. Desse fenômeno, emerge um próximo tópico da biologia sintética, a comunicação célula-célula através do desenvolvimento de mecanismos de quorum sensing sintéticos. Assim, a próprias células podem se comunicar para coordenar e sincronizar o seu comportamento. Mas isso é assunto para posts futuros.
Stricker J, Cookson S, Bennett MR, Mather WH, Tsimring LS, & Hasty J (2008). A fast, robust and tunable synthetic gene oscillator. Nature, 456 (7221), 516-9 PMID: 18971928