CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
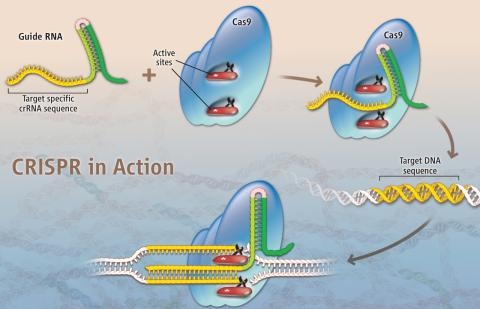 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência:
Bactérias na guerra contra o câncer
 Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Atualmente, desvendar o sistema de comunicação utilizado por células cancerígenas tem sido foco de inúmeras pesquisas. Como em qualquer guerra moderna, destruir o sistema de comunicação inimigo pode causar grandes danos. E na guerra contra o câncer provavelmente não será diferente. Impedir as células de se comunicarem pode evitar que elas adotem estratégias inteligentes tais como ficarem dormentes durante quimioterapia ou até mesmo matar células irmãs para obtenção de alimento. Uma vez entendido a linguagem utilizada por estas células, podemos utilizar isto ao nosso favor, interferindo nas mensagens e fazendo com que células dormentes sejam acordadas durante a quimioterapia ou até mesmo induzir as células cancerígenas a matarem umas as outras.
Finalmente, uma possível estratégia futura seria recrutar bactérias para derrotar o câncer. Elas poderiam ser utilizadas para “ensinar” as células do sistema imunológico a reconhecer e matar as células cancerígenas. É importante ressaltar que ainda compreendemos muito pouco sobre os mecanismos envolvidos no câncer e novas abordagens são necessárias para superá-lo. Entretanto, quem sabe num futuro próximo, estaremos em uma era de guerra cibernética biológica onde bactérias serão inteligentemente projetadas para derrotar o câncer.
Referências:
“Bacterial survival strategies suggest rethinking cancer cooperativity” Eshel Ben-Jacob, Donald S. Coffey, Herbert Levine. Trends in Microbiology. 2012.
“Bacterial linguistic communication and social intelligence” Eshel Ben-Jacob, Israela Becker, Yoash Shapira, Herbert Levine. Trends in Microbiology. 2004
Transposons: pedaços de DNA que mudam de endereço no genoma.
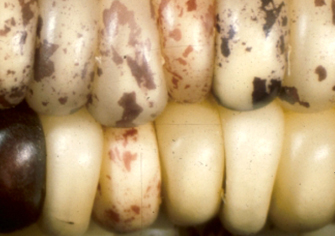
Mudança no padrão das cores do milho devido a interação entre os genes responsáveis pelo pigmento e elementos transponíveis.
Você sabia que alguns trechos de DNA têm a capacidade de mover-se no genoma, saindo de um cromossomo e se inserindo em outro? Pois é, e esta descoberta foi feita há muito tempo atrás por uma brilhante cientista, antes mesmo da descoberta da estrutura do DNA por Watson e Crick. Barbara McClintock ao observar a relação entre os padrões das cores do milho e algumas quebras cromossomais, percebeu que algumas destas quebras ocorriam com uma freqüência muito mais alta do que o esperado e, surpreendente, sempre no mesmo cromossomo. Após uma série de experimentos, a cientista percebeu que esta quebra ocorria devido a inserção de pedaços de DNA vindos de outros cromossomos que alteravam a expressão dos genes de pigmento. Confiante nos seus experimentos, Barbara então propôs que havia elementos transponíveis no genoma, ou seja, os genes não tinham um endereço fixo, mas tinham um mecanismo para movimentar-se dentro do genoma.
Se nos dias de hoje esta ideia parece bastante surpreendente, imagine como foi a recepção desta ousada ideia no começo da década de 50, quando a cientista começou a publicar seus resultados. Eles foram ignorados e ridicularizados pelos seus contemporâneos, levando a cientista a parar de publicar suas descobertas sobre o tema. Somente no início da década de 70 alguns elementos transponíveis foram identificados em bactéria validando assim a teoria de Barbara. Entretanto, o reconhecimento completo de uma das mais importantes descobertas da biologia somente aconteceu no início da década de 80, quando a cientista foi homenageada com o prêmio Nobel.
Atualmente vários estudos já relacionaram os elementos transponíveis, chamados de transposons, a uma série de doenças e como grande fonte de variabilidade genética. Hoje sabe-se que boa parte do genoma é composto destes elementos e dois mecanismos principais já foram identificados. O primeiro deles é um mecanismo de recortar-e-colar, onde alguns pedaços do DNA simplesmente saem de um lugar e movem-se para outra parte do genoma. O outro mecanismo é do tipo cortar-e-colar, gerando mais de uma cópia no genoma. Este último mecanismo é chamado de retrotransposons e consiste em quase metade do genoma humano e será tema de um próximo post.
Estratégias para tempos adversos.
Em tempos adversos é comum haver muitas mudanças e isto não acontece somente com os humanos que saem as ruas, fazem guerra ou revolução. Na natureza observamos inúmeras estratégias dos seres vivos para superar tempos difíceis, algumas bastante chocantes do ponto de vista humano, tais como infanticídio ou canibalismo. E no mundo microscópio não é diferente.
Se você pensa que as bactérias morrem pacíficas quando as condições não são favoráveis você está bastante enganado. Até mesmo para estes minúsculos seres unicelulares a vida pode ser complexa e cheias de decisões difíceis e estressantes a serem tomadas. Existem diversas estratégias para lidar com tempos difíceis, tais como escassez de alimentos e danos ao DNA. Uma das estratégias mais radicais e mais utilizadas quando a coisa está realmente feia é formar esporos, uma resposta celular bastante complexa que envolve a ativação de mais de 500 genes ao longo de aproximadamente 10 horas. Este processo termina com a morte da célula mãe e a formação de uma célula filha dormente com a capacidade de resistir a situações extremas como calor, radiação e presença de substancias químicas. Por outro lado, durante este tempo o esporo não pode tirar vantagem imediata de situações favoráveis para se reproduzir.
Esporulação é uma tomada de decisão bastante complexa e que não se inicia simplesmente com a escassez de alimentos, mas é resultado de uma série de passos que podem ser descritas como decisões celulares sobre como lidar com o stress presente. Envolve, por exemplo, uma comunicação entre as bactérias da mesma colonia por um mecanismo chamado de quorum sensing. Além disto, antes da esporulação, comumente as bactérias tentam outras táticas como ativar um flagelo para buscar alimentos, secretar antibióticos e outras substancias na tentativa de destruir outros micróbios competidores. As células também checam uma série de condições internas antes de decidir esporular tais como a integridade do DNA cromossomal.
Mesmo quando a maior parte da colonia decide por esporular, o material gerado pela lise da membranas das células que esporularam são aproveitados por outras células da colonia que, no caminho para esporular, entram no estado chamado de competência, que consiste em abrir poros na membrana para facilitar a entrada de DNA exógeno, que pode ser utilizado para reparo do DNA e eventualmente como fonte de informação genética que as ajudará a resistir ao momento. Há ainda um outro caminho mais radical tomado por algumas células da colônia que consiste em secretar alguns fatores antibacterianos que faz com que células irmãs fiquem incapazes de esporular, causando inclusive a lise de sua membrana, em uma espécie de canibalismo. Interessante notar que, sobre a perspectiva de teoria de jogos, a estratégia de canibalismo seria predominante sobre a estratégia de entrar em competência, o que não é observado. Isto indica que possivelmente as células em competência são imunes ao canibalismo, mas não sabemos ainda se é este o caso.
Modelos teóricos/moleculares para este tipo de tomada de decisões normalmente consistem na integração de módulos gênicos formados principalmente por circuitos regulados por diversos fatores de transcrição e micro RNAs. Estes tipos de sistemas gênicos também desempenham um papel importante no desenvolvimento embrionário e de células cancerígenas, motivo pelo qual tem se dado muita atenção a este tipo de estudo.
Referencias:
Minas Gerais no iGEM!
Em 2012, dos 16 times da América Latina no iGEM, 6 eram do México. Achamos fascinante o fato deles terem feito um jamboree mexicano e sabiamos que o Brasil também tem estrutura para fazer algo semelhante. Finalmente, este ano temos pela primeira vez mais de um time na competição. Somos 3 times brasileiros, dos quais já falamos dos projetos do time da USP e da UFAM. Resta agora conhecer um pouco do terceiro time que representará o nosso país na competição competição que é da UFMG.
Escrito por Marianna Kunrath Lima e Clara Guerra Duarte
Como tudo começou
Este será o primeiro ano em que uma equipe da UFMG irá participar do iGEM. A ideia de formar uma equipe veio do aluno de medicina Lucas Ribeiro. Ao ler e se interessar pelo evento, ele começou a procurar professores da UFMG que possuíam o perfil/currículo condizente com biologia sintética, que é a base do iGEM. Encontrou a Dr Liza Felicori, professora do Departamento de Bioquímica e Imunologia do ICB-UFMG, que possui experiência na área. E assim começaram os esforços para montar uma equipe da UFMG.
Em janeiro de 2013, foi feita uma palestra para os alunos de graduação e pós graduação interessados na competição. Foram selecionados 2 alunos da graduação de Ciências Biológicas, 1 aluno da graduação de Biomedicina, 1 aluno da graduação de Medicina, 1 aluno da graduação de Ciências da Computação, 1 aluna do Mestrado em Bioquímica e Imunologia, 1 aluno do Mestrado em Bioinformática, 1 aluno do Mestrado em Ciência da Computação, 1 pós-doutoranda em Bioinformática e 1 pós-doutoranda em Bioquímica e Imunologia. Também fazem parte da equipe as professoras Liza Felicori e Santuza Ribeiro, da Bioquímica e Imunologia, além do professor Omar Paranaíba, da Ciência da Computação.

Logo que a equipe foi escolhida, iniciaram-se as reuniões, todas as terças-feiras, às 14 horas. Como o grupo é bem eclético, foram necessárias reuniões introdutórias, sobre assuntos básicos de cada área, para familiarizar os integrantes sobre temas que não dominavam. Além disso, várias apresentações sobre projetos e equipes que participaram de iGEMs anteriores foram feitas, para entendermos bem a competição e termos ideias para o nosso projeto. Como somos pioneiros na universidade, tivemos alguma dificuldade. Após dois meses de reuniões, brainstorms e depois de muito quebrar a cabeça, conseguimos fechar uma ideia preliminar.
Projeto
Decidimos desenvolver um projeto que levasse à detecção de marcadores biológicos prognósticos de síndrome coronariana aguda. Problemas coronarianos têm se tornado cada vez mais frequentes na população, devido aos hábitos sedentários e à má alimentação. Doenças cardiovasculares levam a perdas econômicas, além do evidente prejuízo à saúde que culmina com a perda de vidas. Existem vários testes diagnósticos para estas enfermidades que utilizam biomarcadores, mas estes testes apenas confirmam a doença após a ocorrência de um evento drástico, como infarto ou angina, não sendo capazes de prevê-los. Assim, dada a importância do desenvolvimento de ferramentas capazes de realizar o prognóstico de doenças cardíacas, nosso projeto baseia-se na ideia de detectar biomarcadores existentes no sangue de pacientes, sendo que estes marcadores permitem a constatação prévia da doença, anteriormente a qualquer manifestação da mesma. Para realizar esta detecção, serão utilizadas bactérias Escherichia coli transformadas com plasmídeos que possuam biobricks capazes de “sentir” as entradas (biomarcadores) e biobricks capazes de gerar saídas (fluorescência) para estas entradas. Será utilizada a integração dos sinais gerados por mais de um biomarcador, a fim de aumentar a acurácia do teste. O logo e o nome do projeto (Cardbio) já foram escolhidos.
 Para testarmos a aceitação dessa ideia, bem como para recebermos críticas e sugestões, apresentamos um seminário na Pós Graduação em Bioquímica e Imunologia. Esse seminário foi de grande valia para a equipe, ele serviu não apenas para os propósitos citados acima, como também para unir os integrantes, que tiveram que se esforçar para criar uma boa apresentação e para embasar o projeto.
Para testarmos a aceitação dessa ideia, bem como para recebermos críticas e sugestões, apresentamos um seminário na Pós Graduação em Bioquímica e Imunologia. Esse seminário foi de grande valia para a equipe, ele serviu não apenas para os propósitos citados acima, como também para unir os integrantes, que tiveram que se esforçar para criar uma boa apresentação e para embasar o projeto.
Desde então, vários progressos foram feitos. As modelagens computacionais já estão em andamento, os biobricks estão sendo desenhados, ganhamos uma nova integrante da graduação em Design Gráfico, estamos desenvolvendo meios de conseguir patrocínio e estamos correndo atrás do tempo, uma vez que nossa equipe começou um pouquinho atrasada em relação às demais equipes do iGEM. Temos uma página no Facebook e um blog em construção. Gostaríamos de convidar todos os leitores da SynbioBrasil e todos os iGEMers a contribuírem para o crescimento do nosso projeto, além de agradecermos à equipe da USP pela oportunidade de divulgarmos nossas ideias.
Manaus no iGEM
A história do Brasil do iGEM começou em 2009 com a Unicamp, que também nos representou em 2011. Ano passado foi a vez do nosso time, da USP em parceria com a Unesp. Neste ano, ficamos muito felizes de saber que teremos mais times brasileiros na competição. Até o momento estão inscritos 3 times que representarão nosso país na competição!! E um deles é o time Manaus_Amazonas-Brazil.
Neste post vamos contar um pouco sobre o time do Amazonas e seu projeto. Diferentemente do time da USP, cuja iniciativa começou por parte dos estudantes, o time de Manaus começou por iniciativa do professor Carlos Nunes. Ano passado nosso time teve o prazer de conhecer o professor Carlos que acompanhou a competição para este ano montar um time na sua universidade.
Segue abaixo um pouco do time e de seu projeto, contado pelos próprios alunos integrantes:
Como tudo começou
No final do ano de 2012, graças ao incentivo do professor Carlos Nunes, começamos a nos interessar pelo iGEM e a nos reunir. No início, muitos não tinham ideia de como fazer funcionar a dinâmica do grupo, muito menos como formar um, além de qual projeto iríamos propor.
Após pesquisar e estudar os projetos das equipes anteriores e ter uma melhor noção sobre a competição, começamos a colocar nossa criatividade para funcionar e elaborar ideias/projetos bem distintos. Não foram poucas as inspirações e chegamos a algumas propostas: a bactéria armadilha para o HIV (“Colitrap”), a detectora de Incêndio (“Firebacter”), a jogadora de Pacman (“Pac-Coli”), a indicadora de temperatura (“TermoColi”), a espartana (“MagnetoColi”) e a escolhida, bactéria produtora de energia elétrica (“Electrobacter”).

O projeto
 O foco principal da Electrobacter é degradar gorduras de óleos de fritura residuais amplamente utilizados e descartados por redes de restaurantes para a geração de energia elétrica, pois os mesmos, quando são descartados inadequadamente, apresentam-se como um agente poluidor ao ambiente e o tratamento para eliminar esses poluentes requer o uso de produtos químicos tóxicos, além de o processo ser oneroso e pouco efetivo.
O foco principal da Electrobacter é degradar gorduras de óleos de fritura residuais amplamente utilizados e descartados por redes de restaurantes para a geração de energia elétrica, pois os mesmos, quando são descartados inadequadamente, apresentam-se como um agente poluidor ao ambiente e o tratamento para eliminar esses poluentes requer o uso de produtos químicos tóxicos, além de o processo ser oneroso e pouco efetivo.
Apesar de já existirem alternativas para o aproveitamento dos óleos de fritura como a produção de sabão, o direcionamento de gorduras para geração de energia trará uma nova opção de bioenergia por meio da biorremediação.
A bactéria Shewanella amazonensis que pretendemos utilizar é capaz de fazer o transporte de elétrons dispostos no meio, gerando eletricidade. A proposta é melhorá-la geneticamente para degradar estes óleos através da β-oxidação (reação bioquímica de quebra de gorduras), potencializando a produção de elétrons. O gênero Shewanella é um grupo de bactérias que foram descobertas recentemente e os estudos sobre ela ainda são escassos, principalmente na área da biologia molecular, porém seu potencial não só pela possibilidade de geração de energia, quanto pela sua habilidade de captar metais dispostos no meio. Entre as possibilidades de como modifica-la, optamos por dois caminhos: modificar o repressor ou o promotor dessa via metabólica. Tanto de uma forma quanto da outra, a β-oxidação será possível mesmo com a presença mínima de glicose no meio, porém os efeitos dessa intervenção genética deverão ser estudados de perto para que não tenhamos que sempre deixá-la em condições de estresse.
Com esse o projeto de caráter ambiental e ecológico, estamos empolgados, determinados a participar do iGEM e ser o primeiro time a representar o estado do Amazonas.
As dificuldades
Infelizmente, não só de determinação e força de vontade vive um time do iGEM. Uma das principais dificuldades é pagar a inscrição do time na competição que custa em torno de 3 mil dólares, que paga não somente a inscrição como o envio das partes biologicas necessárias ao projeto e custos do evento. A universidade, bem como agências de fomento de pesquisa, não tem programas de apoio para uma competição como o iGEM. Ano passado, contamos com a colaboração de mais de 40 pessoas do mundo todo que nos apoiaram através do chamado crowdfunding.
Se você ficou triste pois perdeu a oportunidade de apoiar um time brasileiro a fazer ciência de uma forma inovadora e divertida, o time de Manaus está precisando de apoio e você pode colaborar por aqui.
Para saber mais sobre o grupo:
Aventuras em Biologia Sintética
Eu considero os quadrinhos como uma das formas mais interessantes de se narrar uma história. São também uma ótima forma de divulgar ciência de uma maneira didática e divertida.
Drew Endy um dos pais da biologia sintética (e do Registry of Parts), juntamente com Isadora Deese (ambos do MIT) elaboraram o quadrinho Adventures in Synthetic Biology. E tem mais: o quadrinho foi publicado no website da prestigiada revista Nature.
Confira Adventures in Synthetic Biology e aprenda de maneira didática o que são biobrick, PoPs, entre outros conceitos básicos.
Links possivelmente interessantes:
- Bastidores da produção do quadrinho
- The cartoon guide to genetics
- TED talk do Jorge Cham, criador do sensacional PhD Comics
Espetáculo da ciência
“We’ve arranged a global civilization in which most crucial elements profoundly depend on science and technology. We have also arranged things so that almost no one understands science and technology. This is a prescription for disaster. We might get away with it for a while, but sooner or later this combustible mixture of ignorance and power is going to blow up in our faces.”
(Carl Sagan, The Demon-Haunted World: Science as a Candle in the Dark )
[youtube_sc url=”http://youtu.be/cP2VhadII84″ title=”A%20ciência%20como%20uma%20enigmatica%20caixa%20preta%20que%20ao%20ser%20manipulada%20por%20um%20misterioso%20personagem,%20o%20cientista,%20produz%20inovações%20tecnológicas.”]
Caixa preta é um conceito que designa um sistema cujos detalhes de funcionamento são desconhecidos ou mesmo ignorados. Computadores, micro-ondas, aparelho celulares são alguns dos vários exemplos de objetos que são amplamente usados pela sociedade mas que, em geral, não há uma preocupação maior por parte dos usuários com relação ao seu funcionamento.
Grande parte da população brasileira não tem acesso à educação científica, fazendo com que os avanços tecnológicos que a ciência promove apareçam aos olhos do cidadão sob uma forma não muito diferente de um espetáculo mágico. Tem-se uma sociedade industrializada derivada dos avanços na ciência e tecnologia (sem mais discussões apontarei estas duas como indissociáveis) sentada na platéia de olho apenas para o output de uma caixa preta cuja única razão de ser é a geração de mais aplicações tecnológicas. Já o enigmático e estereotipado personagem que manipula a caixa, o cientista, faz parte de uma pequena porcentagem que detém os saberes do que está por trás do funcionamento da mesma.
Apesar de atualmente haver uma maior mobilização relacionada à divulgação científica no Brasil (como o scienceblogs), maior cobertura em revistas e jornais televisivos e principalmente na internet, o quadro ainda é frágil. A mídia, não raramente, apresenta a ciência como um empreendimento espetacular, realizado por pessoas super-dotadas, gênios que nunca cometem equívocos e que fornecem informações cem por cento confiáveis. Consequência dessa mídia exagerada é uma visão distorcida de como funciona a ciência, exemplo recente (e trágico em todos sentidos) foi o ocorrido com cientistas italianos que foram condenados por não preverem um terremoto. É dado uma grande ênfase as aplicações da ciência mas o processo de sua produção, seu contexto, suas limitações e incertezas são ignorados.
Essa falta de compreensão, reflexão e educação cientifica no entanto não impede que tenhamos uma sociedade aberta à ciência (sim!!), e como vimos bem confiante no saber tecnocientífico que superficialmente lhe é apresentada. Não é incomum, por exemplo, comerciais de televisão usarem dessa confiança no conhecimento cientifico afim de ganhar os clientes, do tipo “está comprovada cientificamente a eficácia do produto…”. Ficamos diante de uma sociedade impaciente, exigente e que aguarda cada vez mais da ciência mas que muitas vezes não consegue filtrar as informações sensacionalistas e contraditórias que lhe são apresentadas (Parece milagre! Novo remédio faz emagrecer 7 a 12 quilos em cinco meses. E sem grandes efeitos colaterais!!).
A sociedade comanda direta e indiretamente o que é produzido e proposto cientificamente. A ciência, que antes tinha somente o papel investigativo e sociologicamente marginal, tornou-se hoje o centro da sociedade humana controlada pelos poderes econômicos, estatais e subjulgada a exigências dessa sociedade. Portanto, ao mesmo tempo que a ciência possui autoridade e é dominadora, ela também é dominada por essa demanda da sociedade meramente usuária. Isso é perigoso, vamos olhar como exemplo para um cenário particular de interesse aqui do blog; a biologia sintética é hoje uma área onde a sociedade está depositando muito de suas expectativas (e dinheiro). Esta pressão social por inovações pode ser bastante trágica nesta área, uma vez que pequenos bugs num produto biológico pode apresentar consequências drásticas.
O ponto principal a ser pensado aqui é a importância e necessidade que TODOS tenhamos consciência do que estamos criando e para que isto está sendo feito frente ao avanço cientifico e tecnológico. Para os envolvidos diretamente com ciência resta pensar no poder do que produzem, um poder pelo qual muitas vezes pode-se perder o poder. Instala-se a necessidade de uma verdadeira reflexão sobre os caminhos que os cientistas e o conhecimento científico podem seguir para que se tornem mais acessíveis. O avanço na divulgação científica tem que continuar crescendo, no entanto, prezando pela boa qualidade. Para os não envolvidos ansiosos pelas novidades do ano, pelos produtos recém-saídos do forno, embalados e “pronto para consumo” resta tomar uma postura crítica maior com relação a mudanças que afetam diretamente as suas vidas. Abrir caixas pretas é uma tarefa árdua mas o primeiro passo é se dar conta de que existem. Vale a tentativa(!!!) uma vez que o progresso da ciência e por sua vez de toda sociedade depende disso, depende das ideias inovadoras, das discussões, das críticas. Dependemos do conhecimento.
Autoria: Carolina Menezes Silvério e Marcelo Boareto.
Modelagem em biologia (sintética), um guia para ateus em modelagem.
 Para uma pessoa que lida diariamente com biologia, pode ser bastante difícil imaginar uma maneira de abordar matematicamente um problema em sua área ou como isto poderia ter alguma utilidade. A biologia é uma ciência considerada bastante complexa, pois são muitas as variáveis que podem afetar o sistema. Até mesmo um sistema relativamente simples como a expressão de uma proteína em E. coli, pode se tornar um problema bastante complexo de se modelar se todas as variáveis que afetam a expressão desta proteína forem levadas em consideração. Na realidade, não há nem mesmo quem seja capaz de listar todas estas variáveis. Assim sendo, como é possível fazer um modelo disto, se não consigo nem mesmo listar as variáveis que alteram meu sistema? Neste caso, melhor mesmo fazer como o Calvin e ser um ateu em modelagem, não é mesmo?
Para uma pessoa que lida diariamente com biologia, pode ser bastante difícil imaginar uma maneira de abordar matematicamente um problema em sua área ou como isto poderia ter alguma utilidade. A biologia é uma ciência considerada bastante complexa, pois são muitas as variáveis que podem afetar o sistema. Até mesmo um sistema relativamente simples como a expressão de uma proteína em E. coli, pode se tornar um problema bastante complexo de se modelar se todas as variáveis que afetam a expressão desta proteína forem levadas em consideração. Na realidade, não há nem mesmo quem seja capaz de listar todas estas variáveis. Assim sendo, como é possível fazer um modelo disto, se não consigo nem mesmo listar as variáveis que alteram meu sistema? Neste caso, melhor mesmo fazer como o Calvin e ser um ateu em modelagem, não é mesmo?
Na verdade não, fazer um modelo não é tão complicado assim. Pensar que é necessário colocar tudo no modelo é o erro conceitual mais comum de quem tem formação em áreas complexas como a biologia. Já os físicos por exemplo, tem uma visão dita reducionista, de tentar entender um problema dividindo-o em pequenas partes fundamentais e começar pelo modelo mais simples possível para depois aumentar a complexidade, se necessário (ou possível). Reza a lenda que um dono galinheiro certa vez chamou um fisico para solucionar o porquê as galinhas não estavam botando. Uma semana depois o fisico apareceu com a solução. Entretanto, a solução só era válida para galinhas esfericamente simétricas e no vácuo. True story!
Pode parecer contraditório, mas um modelo que leva mais variáveis em consideração não necessariamente é melhor ou mais “realistico”. Na verdade se um modelo leva mais variáveis em consideração do que outro e ambos tem a mesma efeciência, o segundo modelo é considerado melhor, conforme veremos.
Portanto, o melhor caminho para fazer um bom modelo é considerar as poucas e relevantes variáveis do problema, ou seja, ao fazer um modelo, a principal regra é:
Keep it simple, stupid!!
Este é o famoso princípio KISS, uma boa regra para começar um modelo. A maioria dos modelos funcionam melhor e são melhor entendidos se mantidos simples. Complexidade desnecessária deve ser evitada, mas obviamente, simplicidade demasiada não deve resultar em um modelo útil (como no caso das galinhas). Assim, uma boa maneira de se começar um modelo é pensar quais são as variáveis que devem ser realmente importantes para o problema. Tente formular seu modelo com o mínimo de variáveis e veja se seus resultados condizem com o esperado, ou com os experimentos. Se isto não ocorrer, é um sinal de que seu modelo ou é demasiadamente simples e você esqueceu alguma variável muito importante ou você pode ter feito hipóteses que não sejam válidas. Fazer hipóteses condizentes não é uma tarefa na simples e exige um conhecimento profundo do problema em questão.
O principio KISS é um conceito bastante semelhante à famosa navalha de Occam. Este princípio, introduzido por William Occam diz:
“Se em tudo o mais forem idênticas as várias explicações de um fenômeno, a mais simples é a melhor.”
Exemplo
Para exemplificar o que foi dito, vamos brincar de modelar com um simples exemplo que discutimos certa vez em nosso grupo.
O problema consiste em estimar a concentração de uma proteína (nosso caso era a Cre recombinase) dentro das bactérias E.coli, ou seja, quantas Cre-recombinases existem, em média, por bactéria? Esta inferência é base para estimar o PoPS (veja post anterior)
Este parece ser um problema simples mas pode se tornar bastante complicado de resolver caso o princípio KISS não seja utilizado. Quem é importante neste problema? Devo considerar a temperatura? Devo considerar a quantidade de alimento no meio?
Você pode pensar, e com razão, que a temperatura e a quantidade de alimento são importantes pois afetam a taxa de produção das proteínas. Entretanto estes são exemplos de variáveis que não precisam
ser levados em consideração pois, nos experimentos não trabalharemos com situações extremas de escassez de alimento nem de mudanças de temperatura e pequenas variações destas variáveis (fora de um regime extremo) não devem afetar significantemente a produção da proteína. Muitas são as variáveis que não afetam significamente o sistema e ter intuição disto é fundamental e repito, exige um bom entendimento do problema.
OK, mas por onde começar?
Bom, sabemos que para se produzir uma proteína primeiramente precisamos da produção do RNA mensageiro. A quantidade de mRNA certamente é uma variável relevante!!!
Portanto, vamos tentar criar uma equação sobre como o mRNA deve variar no tempo. A variação temporal de uma variável é representada matematicamente pela derivada da variável no tempo ![]()
Sabemos que nosso mRNA deve ser produzido pela leitura do DNA, feita pela DNA polimerase. OK, mas com que velocidade ela lê isto? Uma boa referencia, é o Bionumbers (tipo um google para dados biológicos)
Lá encontramos que nossa taxa de transcrição (Ktrans) é de, em média, 40 pares de base por segundo. Mas então precisamos saber qual o tamanho do RNA que gera nossa proteína. No caso da Cre é de 1032 pares de base (Nbp). Portanto, quantidade de proteína produzida por tempo e por volume (V) é de:
![]()
Dividimos pelo volume pois queremos saber a variação de concentração, ou seja, quantidade de proteínas por volume (unidade em Molar). Este será o primeiro termo de nossa equação, que se refere a produção do mRNA. Existem outras maneiras dele ser produzido? Se sim, novos termos devem ser adicionados. Neste caso, aparentemente esta é a unica forma dele ser produzido. Mas ele pode ser degradado, não é mesmo? Então precisamos de mais um termo, o de degradação. Novamente se formos até o bionumbers teremos a taxa de degradação (KdRNA) do mRNA. Este novo termo fará com que a taxa do mRNA diminua no tempo, e por este motivo ele é negativo. Portanto nossa equação fica:
![]()
Onde o termo positivo se refere a produção e o negativo se refere a degradação.
Agora vamos escrever uma equação para a tradução do mRNA em proteína. Neste caso encontramos uma taxa de tradução de 15 aminoacidos por segundo. Como nossa proteina tem 1032 pares de base ela deve ter 1032/3=344 aminoacidos. Como, além de produzida, nossa proteína também pode ser degradada então temos uma equação bastante semelhate à anterior:
![]()
Podemos supor que inicialmente a concentração desta proteína é zero, isto não fará diferença nos cálculos mas suponhamos que não haja proteina inicialmente. Ao longo do tempo, a concentração
desta proteína irá crescer até alcançar o equilibrio entre produção e degradação. Neste equilibrio, a concentração das proteínas não mudam mais no tempo e portanto nossas equações são iguais a zero. Para entender o equilibrio, pense na equação logistica que descreve a curva de crescimento de uma população de bactérias. Inicialmente temos um crescimento exponencial, mas depois de um tempo a população satura, ou seja, estabiliza em uma determinada população. Este ponto de saturação é que chamamos de ponto de equilibrio, onde a quantidade de bactérias não muda mais no tempo. Neste ponto, a quantidade de bactérias que morrem é proporcional às que “nascem”. Matematicamente o ponto de equilibrio é um ponto onde a derivada no tempo é igual a zero, portanto:
![]()
![]()
ou seja:
![]()
e
![]()
Agora podemos isolar a concentração do mRNA na primeira equação e substituir na segunda. Com isto, chegamos a:
![]()
Qual o sentido deste valor? Bem você pode utilizar o volume da bactéria e calcular qual a concentração de uma única proteína dentro de uma bactéria e você chegará que isto é aproximadamente 1 nM. Portanto, nosso resultado nos diz que há aproximadamente 2.000 proteínas, em média, dentro da bactéria.
OK, isto quer dizer que se eu fizer um experimento eu vou encontrar exatamente 2000 proteínas dentro da bactéria?
Obviamente não, devemos ter em mente a limitação de nosso modelo. Aproximações foram feitas e há muitas variáveis que podem fazer com que este valor mude. Entretanto, podemos dizer com certa segurança que teriamos algo de 1.000 à 10.000 proteínas na bactéria. Pode parecer muito inexato e que nosso modelo não foi tão útil por não ser preciso. Mas devemos lembrar que inicialmente não tínhamos nenhuma ideia de quantas proteínas haviam. Se alguém chutasse que há somente 10 ou 100 proteínas em média poderiamos pensar que era uma estimativa boa. Com o modelo sabemos que esta estimativa não é boa, que devem haver bem mais proteínas que isto!
Além da quantidade de proteína, com este simples modelo poderiamos estimar o tempo que demora para que a quantidade de proteína sature, ou seja, atinja o ponto de equilibrio. Estas são estimativas que podem ser muito úteis na hora de definir um protocolo experimental e pode economizar uma razoável quantidade de tempo e reagentes durante os experimentos. Portanto, não é necessário de ser ateu em modelagem, mas, tampouco, é recomendado acreditar religiosamente no modelo!!









