CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
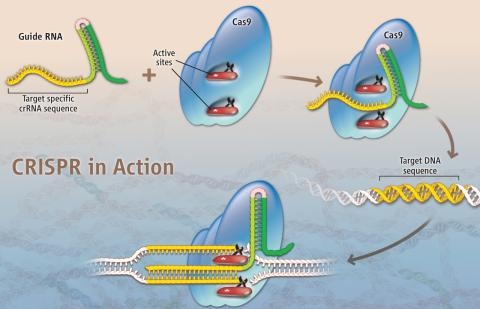 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência:
Modelagem em biologia (sintética), um guia para ateus em modelagem.
 Para uma pessoa que lida diariamente com biologia, pode ser bastante difícil imaginar uma maneira de abordar matematicamente um problema em sua área ou como isto poderia ter alguma utilidade. A biologia é uma ciência considerada bastante complexa, pois são muitas as variáveis que podem afetar o sistema. Até mesmo um sistema relativamente simples como a expressão de uma proteína em E. coli, pode se tornar um problema bastante complexo de se modelar se todas as variáveis que afetam a expressão desta proteína forem levadas em consideração. Na realidade, não há nem mesmo quem seja capaz de listar todas estas variáveis. Assim sendo, como é possível fazer um modelo disto, se não consigo nem mesmo listar as variáveis que alteram meu sistema? Neste caso, melhor mesmo fazer como o Calvin e ser um ateu em modelagem, não é mesmo?
Para uma pessoa que lida diariamente com biologia, pode ser bastante difícil imaginar uma maneira de abordar matematicamente um problema em sua área ou como isto poderia ter alguma utilidade. A biologia é uma ciência considerada bastante complexa, pois são muitas as variáveis que podem afetar o sistema. Até mesmo um sistema relativamente simples como a expressão de uma proteína em E. coli, pode se tornar um problema bastante complexo de se modelar se todas as variáveis que afetam a expressão desta proteína forem levadas em consideração. Na realidade, não há nem mesmo quem seja capaz de listar todas estas variáveis. Assim sendo, como é possível fazer um modelo disto, se não consigo nem mesmo listar as variáveis que alteram meu sistema? Neste caso, melhor mesmo fazer como o Calvin e ser um ateu em modelagem, não é mesmo?
Na verdade não, fazer um modelo não é tão complicado assim. Pensar que é necessário colocar tudo no modelo é o erro conceitual mais comum de quem tem formação em áreas complexas como a biologia. Já os físicos por exemplo, tem uma visão dita reducionista, de tentar entender um problema dividindo-o em pequenas partes fundamentais e começar pelo modelo mais simples possível para depois aumentar a complexidade, se necessário (ou possível). Reza a lenda que um dono galinheiro certa vez chamou um fisico para solucionar o porquê as galinhas não estavam botando. Uma semana depois o fisico apareceu com a solução. Entretanto, a solução só era válida para galinhas esfericamente simétricas e no vácuo. True story!
Pode parecer contraditório, mas um modelo que leva mais variáveis em consideração não necessariamente é melhor ou mais “realistico”. Na verdade se um modelo leva mais variáveis em consideração do que outro e ambos tem a mesma efeciência, o segundo modelo é considerado melhor, conforme veremos.
Portanto, o melhor caminho para fazer um bom modelo é considerar as poucas e relevantes variáveis do problema, ou seja, ao fazer um modelo, a principal regra é:
Keep it simple, stupid!!
Este é o famoso princípio KISS, uma boa regra para começar um modelo. A maioria dos modelos funcionam melhor e são melhor entendidos se mantidos simples. Complexidade desnecessária deve ser evitada, mas obviamente, simplicidade demasiada não deve resultar em um modelo útil (como no caso das galinhas). Assim, uma boa maneira de se começar um modelo é pensar quais são as variáveis que devem ser realmente importantes para o problema. Tente formular seu modelo com o mínimo de variáveis e veja se seus resultados condizem com o esperado, ou com os experimentos. Se isto não ocorrer, é um sinal de que seu modelo ou é demasiadamente simples e você esqueceu alguma variável muito importante ou você pode ter feito hipóteses que não sejam válidas. Fazer hipóteses condizentes não é uma tarefa na simples e exige um conhecimento profundo do problema em questão.
O principio KISS é um conceito bastante semelhante à famosa navalha de Occam. Este princípio, introduzido por William Occam diz:
“Se em tudo o mais forem idênticas as várias explicações de um fenômeno, a mais simples é a melhor.”
Exemplo
Para exemplificar o que foi dito, vamos brincar de modelar com um simples exemplo que discutimos certa vez em nosso grupo.
O problema consiste em estimar a concentração de uma proteína (nosso caso era a Cre recombinase) dentro das bactérias E.coli, ou seja, quantas Cre-recombinases existem, em média, por bactéria? Esta inferência é base para estimar o PoPS (veja post anterior)
Este parece ser um problema simples mas pode se tornar bastante complicado de resolver caso o princípio KISS não seja utilizado. Quem é importante neste problema? Devo considerar a temperatura? Devo considerar a quantidade de alimento no meio?
Você pode pensar, e com razão, que a temperatura e a quantidade de alimento são importantes pois afetam a taxa de produção das proteínas. Entretanto estes são exemplos de variáveis que não precisam
ser levados em consideração pois, nos experimentos não trabalharemos com situações extremas de escassez de alimento nem de mudanças de temperatura e pequenas variações destas variáveis (fora de um regime extremo) não devem afetar significantemente a produção da proteína. Muitas são as variáveis que não afetam significamente o sistema e ter intuição disto é fundamental e repito, exige um bom entendimento do problema.
OK, mas por onde começar?
Bom, sabemos que para se produzir uma proteína primeiramente precisamos da produção do RNA mensageiro. A quantidade de mRNA certamente é uma variável relevante!!!
Portanto, vamos tentar criar uma equação sobre como o mRNA deve variar no tempo. A variação temporal de uma variável é representada matematicamente pela derivada da variável no tempo ![]()
Sabemos que nosso mRNA deve ser produzido pela leitura do DNA, feita pela DNA polimerase. OK, mas com que velocidade ela lê isto? Uma boa referencia, é o Bionumbers (tipo um google para dados biológicos)
Lá encontramos que nossa taxa de transcrição (Ktrans) é de, em média, 40 pares de base por segundo. Mas então precisamos saber qual o tamanho do RNA que gera nossa proteína. No caso da Cre é de 1032 pares de base (Nbp). Portanto, quantidade de proteína produzida por tempo e por volume (V) é de:
![]()
Dividimos pelo volume pois queremos saber a variação de concentração, ou seja, quantidade de proteínas por volume (unidade em Molar). Este será o primeiro termo de nossa equação, que se refere a produção do mRNA. Existem outras maneiras dele ser produzido? Se sim, novos termos devem ser adicionados. Neste caso, aparentemente esta é a unica forma dele ser produzido. Mas ele pode ser degradado, não é mesmo? Então precisamos de mais um termo, o de degradação. Novamente se formos até o bionumbers teremos a taxa de degradação (KdRNA) do mRNA. Este novo termo fará com que a taxa do mRNA diminua no tempo, e por este motivo ele é negativo. Portanto nossa equação fica:
![]()
Onde o termo positivo se refere a produção e o negativo se refere a degradação.
Agora vamos escrever uma equação para a tradução do mRNA em proteína. Neste caso encontramos uma taxa de tradução de 15 aminoacidos por segundo. Como nossa proteina tem 1032 pares de base ela deve ter 1032/3=344 aminoacidos. Como, além de produzida, nossa proteína também pode ser degradada então temos uma equação bastante semelhate à anterior:
![]()
Podemos supor que inicialmente a concentração desta proteína é zero, isto não fará diferença nos cálculos mas suponhamos que não haja proteina inicialmente. Ao longo do tempo, a concentração
desta proteína irá crescer até alcançar o equilibrio entre produção e degradação. Neste equilibrio, a concentração das proteínas não mudam mais no tempo e portanto nossas equações são iguais a zero. Para entender o equilibrio, pense na equação logistica que descreve a curva de crescimento de uma população de bactérias. Inicialmente temos um crescimento exponencial, mas depois de um tempo a população satura, ou seja, estabiliza em uma determinada população. Este ponto de saturação é que chamamos de ponto de equilibrio, onde a quantidade de bactérias não muda mais no tempo. Neste ponto, a quantidade de bactérias que morrem é proporcional às que “nascem”. Matematicamente o ponto de equilibrio é um ponto onde a derivada no tempo é igual a zero, portanto:
![]()
![]()
ou seja:
![]()
e
![]()
Agora podemos isolar a concentração do mRNA na primeira equação e substituir na segunda. Com isto, chegamos a:
![]()
Qual o sentido deste valor? Bem você pode utilizar o volume da bactéria e calcular qual a concentração de uma única proteína dentro de uma bactéria e você chegará que isto é aproximadamente 1 nM. Portanto, nosso resultado nos diz que há aproximadamente 2.000 proteínas, em média, dentro da bactéria.
OK, isto quer dizer que se eu fizer um experimento eu vou encontrar exatamente 2000 proteínas dentro da bactéria?
Obviamente não, devemos ter em mente a limitação de nosso modelo. Aproximações foram feitas e há muitas variáveis que podem fazer com que este valor mude. Entretanto, podemos dizer com certa segurança que teriamos algo de 1.000 à 10.000 proteínas na bactéria. Pode parecer muito inexato e que nosso modelo não foi tão útil por não ser preciso. Mas devemos lembrar que inicialmente não tínhamos nenhuma ideia de quantas proteínas haviam. Se alguém chutasse que há somente 10 ou 100 proteínas em média poderiamos pensar que era uma estimativa boa. Com o modelo sabemos que esta estimativa não é boa, que devem haver bem mais proteínas que isto!
Além da quantidade de proteína, com este simples modelo poderiamos estimar o tempo que demora para que a quantidade de proteína sature, ou seja, atinja o ponto de equilibrio. Estas são estimativas que podem ser muito úteis na hora de definir um protocolo experimental e pode economizar uma razoável quantidade de tempo e reagentes durante os experimentos. Portanto, não é necessário de ser ateu em modelagem, mas, tampouco, é recomendado acreditar religiosamente no modelo!!
Clube de Biologia Sintética 2012
Começaremos novamente nesta quarta feira (15 de agosto) as reuniões do Clube de Biologia Sintética!
Não importa a área: se você se interessar por biotecnologia, pesquisa interdisciplinar, gosta de aprender coisas novas e de resolver desafios, está mais que convidado!
Você de Exatas
No Clube você pode descobrir um novo mundo para “programar”, modelar e resolver problemas utilizando conceitos de engenharia para se fazer o design de sistemas biológicos.
NOTA: Nesse ano colocamos um Físico em um laboratório de Biologia Molecular; foi mais ou menos assim:
[youtube_sc url=”http://www.youtube.com/watch?v=3drspKkfIyE”]
Você de Biológicas
Nas reuniões você pode descobrir que a Biologia pode ser muito mais exata do que você conhece, e a diferença é só de abordagem.
E qual o Objetivo de Tudo Isso!?
Usar criatividade, interdisciplinariedade e empreendedorismo para esboçar um projeto para a competição internacional de máquinas geneticamente modificadas de 2013! E se divertir no processo, é claro!
A ideia é gerar propostas para serem apresentadas nas reuniões à partir de brainstormmings e referências indicadas por qualquer participante do grupo. É uma oportunidade de se aprender a como se iniciar um projeto do zero, planejar experimentos, verificar viabilidades, procurar materiais, custos, buscar financiamento, organizar pessoas, tempo e recursos; ou seja: tudo o que você precisa para se dar bem em qualquer empreendimento.
Já temos um pequeno cronograma ainda com detalhes a definir, mas com temas já escolhidos. Depois disso quem fará as reuniões serão os próprios participantes, convidados a apresentar suas ideias, assuntos interessantes e relevantes de synbio e qualquer outra coisa que se encaixe na reunião.
Nosso projeto para o iGEM 2012 ainda não acabou, mas vamos trazer toda a experiência que estamos acumulando para melhorar ainda mais ano que vem!
Horário: Das 18:30 às 20:00 hrs
Local: Sala “Fava Netto”, do ICB II – USP.
Vamos transmitir a reunião por Livestream. Colocaremos o link no facebook, twitter e aqui no blog quando iniciarmos a transmissão. Fiquem ligados!
Scinamate Synthetic Biology
Vídeo legal sobre SynBio
[youtube=http://www.youtube.com/watch?v=xOx3B2Z_qqE]
[twitter-follow screen_name=’MnlimasBio’]
ACS Synthetic Biology
A American Chemical Society anunciou o lançamento de uma nova
revista de artigos científicos (revisada por pares), a ACS Synthetic Biology. O editor-chefe será o Christopher A. Voigt, do MIT, e a revista publicará artigos de pesquisas de alta qualidade, cartas, notas técnicas, tutoriais e revisões que nos farão entender melhor a organização e a função das células, tecidos e organismos em seus sistemas. A revista é particularmente interessada em estudos do design e síntese de novos circuitos genéticos e produtos gênicos; métodos computacionais para o design de sistemas; e abordagens integrativas e aplicadas para o entendimento de doenças e metabolismo. A revista começará a aceitar submissões em julho deste ano.
Alguns dos tópicos incluem * :
- Design e otimização de sistemas gênicos
- Design de circuitos gênicos e seus princípios para sua organização em programas
- Métodos computacionais para ajudar no design de sistemas gênicos
- Métodos experimentais para quantificar partes genéticas, circuitos e fluxos metabólicos
- Bibliotecas de partes genéticas: sua criação, análise, e representação ontológica
- Engenharia de proteínas incluindo o design computacional
- Engenharia metabólica e produção celular, incluindo conversão de biomassa
- Engenharia e produção de produtos naturais
- Aplicações inovadoras e criativas de programação celular
- Aplicações médicas, engenharia de tecidos, e a programação de células terapêuticas
- Design e construção da “célula mínima”
- Engenharia viral
- Metodologias de síntese de DNA
- Biologia de Sistemas e métodos para integrar múltiplos dados
*(veja a lista completa no site, que não se limita aos tópicos mencionados)
[twitter-follow screen_name=’MnlimasBio’]
Biologia sintética e a computação
Ontem tivemos nossa primeira reunião do Clube Científico de Biologia Sintética para discutir o artigo “Synthetic biology: new engineering rules for an emerging disciplines.” A imagem abaixo resume bastante a abordagem dos autores do Departamento de Engenharia Elétrica Princeton para conduzir a revisão sobre o assunto.
Os autores traçam um paralelo entre a biologia e os computadores, no qual, não apenas se procura explicar a biologia celular utilizando a computação como analogia, mas também, mostra que já foram desenvolvidos componentes biológicos que funcionam como componentes de computadores. São dados exemplos de várias construções biológicas sintéticas que funcionam como componentes elétricos, como inversores (inverters devices), flip-flops (toggle-swicthes), osciladores (oscilators), amplificadores de sinais (transcriptonal cascades modules) e desviadores de sinais (diverter scaffolds). Restando assim, poucos módulos para se construir um microcomputador celular sintético.
Os autores comentam como estes módulos sintéticos e a condição endógena celular influenciam o comportamento um do outro, sendo que qualquer flutuação nos processos da célula hospedeira podem influenciar o módulo e sua reposta (output). Dessa maneira, torna-se necessário combinar técnicas de estimação de parâmetros e técnicas de análises de fluxos metabólicos para entender o contexto celular e os impactos desses módulos na célula. Para explicar isto de uma maneira resumida, a conectividade dos módulos entre si e com a célula não é suficiente para definir a dinâmica de uma rede, é preciso também incluir parâmetros cinéticos e regulatórios (velocidade das reações, feedbacks, efeito de reguladores…) que podem variar sua atividade de acordo com as mudanças realizadas no sistema original. Estes cálculos, porém, são muitos complicados e demandam uma matématica muito avançada. O que demonstra, mais uma vez, a necessidade de equipes multidisciplinares para a formação de grupos de pesquisa em synbio.
O artigo mostra também que células sintéticas estão se tornando cada vez mais fáceis de construir. Não só pela nossa capacidade de manipular os componentes celular, mas pelo aumento da nossa capacidade de sintetizar DNA. Existem porém, desafios e limitações nos tipos de atividades complexas que uma célula independente consegue realizar de uma forma confiável. Assim, uma nova fronteira para a synbio é distribuir redes e módulos sintéticos entre múltiplas células, formando sistemas de comunicações célula-célula, visando aumentar a possibilidade de desenhos e superar a confiança limitada de células sintéticas individuais. Para isso, já estão se desenvolvendo módulos de quorum sensing (mecanismos de comunicação celular) sintéticos que possibilitam a coordenação do comportamento de comunidades microbianas. Verifica-se, portanto, que muitos avanços têm sido realizados para aumentar a complexidade da arquitetura das redes sintéticas.
Este artigo é particularmente interessante porque mostra a visão de engenheiros elétricos do que é a biologia sintética. É importante destacar que existem diferentes visões e abordagens de pesquisa a respeito do que é a biologia sintética e como ela pode ser aplicada, dependendo da especialidade e background do grupo de pesquisa.
Nas próximas reuniões pretendemos abordar tópicos mais específicos da biologia sintética, como a construção de um oscilador sintético, e mostrar diferentes visões da biologia sintética.
Até lá!
Andrianantoandro, E., Basu, S., Karig, D., & Weiss, R. (2006). Synthetic biology: new engineering rules for an emerging discipline Molecular Systems Biology, 2 DOI: 10.1038/msb4100073
BioBricks: fabricação de pequenos fragmentos de DNA
 Este protocolo é utilizado para a fabricação de BioBricks pequenos, como promotores ou sítios de ligação do ribossomo (RBS). Para isso, utiliza o anelamento e extensão dos primers para criar um pequeno fragmento de DNA (~ 100 bp) utilizando Taq polimerase de alta fidelidade. O fragmento de DNA pode ser imediatamente utilizado em uma reação de clonagem utilizando TOPO-TA. Caso se deseje realizar uma etapa de digestão do fragmento de DNA, uma etapa de purificação de produto de PCR é necessária.
Este protocolo é utilizado para a fabricação de BioBricks pequenos, como promotores ou sítios de ligação do ribossomo (RBS). Para isso, utiliza o anelamento e extensão dos primers para criar um pequeno fragmento de DNA (~ 100 bp) utilizando Taq polimerase de alta fidelidade. O fragmento de DNA pode ser imediatamente utilizado em uma reação de clonagem utilizando TOPO-TA. Caso se deseje realizar uma etapa de digestão do fragmento de DNA, uma etapa de purificação de produto de PCR é necessária.
Materiais:
– Dois primers que se sobrepõe por ~20 bp.
Primer 1: 5′ ———————————– 3′
Primer 2: 3′ ———————————– 5′
– Mix para PCR Taq alta fidelidade
Método
1. Diluir os dois oligos a uma concentração de 25 μM utilizando H2O. Para primers maiores que 50-60 bp podem ocorrer problemas como erros e deleções, por isso, pode valer a pena incluir uma etapa de purificação extra PAGE (Invitrogen).
2. Misturar os reagentes em um tubo estéril de 0.6 mL:
- 9 μL PCR supermix
- 0.5 μL primer 1
- 0.5 μL primer 2
3. A reação de anelamento e extensão dos primers ocorrem no termociclador segundo o seguinte protocolo:
- 94°C por 5 mins
- 94°C por 30 seconds
- 55°C por 30 seconds (ou qualquer outra temperatura de anelamento)
- 72°C por 30 seconds
- Repita os passos 2-4 por 2-3 ciclos
- 72°C por 5 mins
4. Utilize 1μL de produto de PCR fresco (feito no mesmo dia) numa reação de clonagem com TOPO TA cloning.
Para desenhar um BioBrick por esse método não se esqueça de colocar o prefixo e sufixo dos BioBricks nos primers. Pronto, já descrevemos os protocolos para desenhar e montar um circuito sintético com promotores, RBS e genes.
Boa sorte e qualquer dúvida, por favor, pergunte!
Referências
Stemmer WP, Crameri A, Ha KD, Brennan TM, and Heyneker HL. Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides. Gene 1995 Oct 16; 164(1) 49-53. pmid:7590320. PubMed HubMed PubGet [Stemmer-Gene-1995].
http://openwetware.org/wiki/Knight:Annealing_and_primer_extension_with_Taq_polymerase







