CRISPR: nova e revolucionária técnica para edição de genoma
 Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Ter o genoma sequenciado por apenas 1000 dólares em breve será uma realidade. E não é somente ler o genoma que está se tornando cada vez mais acessível, descobertas recentes resultaram em uma nova ferramenta de edição de genoma que promete revolucionar a pesquisa médica e o tratamento de algumas doenças. Este mecanismo, chamado de CRISPR, é baseado num sistema de defesa contra vírus, uma espécie de sistema imunológico, encontrado em bactérias. E mais uma aprendemos algo interessante com estes seres unicelulares (discutimos num post anterior como bactérias podem ajudar a combater o cancer).
Editando o genoma para curar doenças
Tornar o tratamento de desordens genéticas é sem dúvida uma das mais excitantes possibilidades desta nova técnica, principalmente disordens causadas por uma ou poucas mutações, tais como doença de Huntington. Para comprovar que isto pode ser feito, cientistas do MIT, em experimentos em camundongos, conseguiram curar em uma doença rara que ataca o fígado e é causado pela mutação de apenas um par de base de DNA. Esta doença, que também ocorre em humanos, afeta 1 em cada 100.000 pessoas e consiste na falha da quebra do aminoácido tirosina que acumula e afeta o funcionamento do fígado. Utilizando a técnica de CRISPR os cientistas conseguiram corrigir o gene para 1 em cada 250 células do figado (hepatócitos) dos camundongos. Depois de 30 dias estas células proliferaram e substituiram parte das células com o gene defeituoso chegando a um terço da população total de células, o que foi suficiente para curar a doença. Veja o artigo publicado na Nature.
Mecanismo básico das ferramentas de edição de genoma
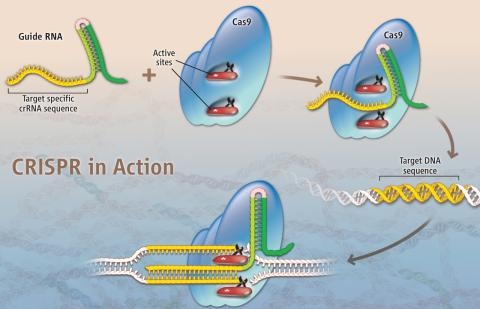 Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
Basicamente, o mecanismo de edição de genoma consiste em um sistema para reconhecer o sítio onde haverá a mudança combinado a um mecanismo de corte do DNA (nucleases). Uma vez reconhecido o local de corte as nucleases agem fazendo um corte nas duas fitas do DNA. Uma vez cortado, mecanismos de reparação do genoma tendem a juntar as fitas novamente e neste processo um pedaço de DNA pode ser removido ou até mesmo trocado por outro pedaço de DNA.
As primeiras técnicas desenvolvidas, tanto Zinc finger nucleases quanto TALEN, utilizam proteínas para reconhecer o sítio de corte no genoma. Proteínas são pesadas e díficeis de projetar, diferentemente de RNA que pode ser facilmente sintetizado. E é aí que está a grande inovação da técnica de CRISPR, em utilizar pequenos pedaços de RNA para identificar o sítio de corte, o que torna a técnica simples e de baixo custo.
Recomendo os seguintes vídeos/animações para uma ilustração do mecanismo de edição de genomas. O primeiro video (em inglês) fala um pouco sobre os mecanismos gerais destas técnicas. O segundo vídeo (também em inglês) ilustra o mecanismo baseado na CRISPR.
Referência:
O que é Biologia Sintética?
 Este post deveria ser um dos primeiros artigos para um site que se diz especializado em biologia sintética. Mas confesso para vocês que definir o que é biologia sintética, para mim, não foi (é) uma tarefa fácil. Como podem ser vistos nos posts no blog, existem vários aspectos da synbio que permitem diferentes definições de acordo com o ponto de vista de quem está fazendo biologia sintética. Por exemplo, um engenheiro interessado em criar dispositivos computacionais sintéticos em uma bactéria, ou um biotecnólogo interessado em produzir biocombustíveis ou um biólogo querendo montar uma bactéria a partir de simples elementos químicos. Claramente, todas estas áreas estão conectadas, mas criar uma definição que consiga embarcar todas as possibilidades não é trivial. Por isso, digo que este post é orgânico, que deve mudar à medida que o meu conhecimento sobre o assunto se aprofunda.
Este post deveria ser um dos primeiros artigos para um site que se diz especializado em biologia sintética. Mas confesso para vocês que definir o que é biologia sintética, para mim, não foi (é) uma tarefa fácil. Como podem ser vistos nos posts no blog, existem vários aspectos da synbio que permitem diferentes definições de acordo com o ponto de vista de quem está fazendo biologia sintética. Por exemplo, um engenheiro interessado em criar dispositivos computacionais sintéticos em uma bactéria, ou um biotecnólogo interessado em produzir biocombustíveis ou um biólogo querendo montar uma bactéria a partir de simples elementos químicos. Claramente, todas estas áreas estão conectadas, mas criar uma definição que consiga embarcar todas as possibilidades não é trivial. Por isso, digo que este post é orgânico, que deve mudar à medida que o meu conhecimento sobre o assunto se aprofunda.
Uma característica que une todos os biológos sintéticos é a vontade de tornar o processo de engenharia de sistemas biológicos mais fácil e confiável. Dentro desse contexto, existem quatro diferentes níveis de atuação da biologia sintética:
(i) partes biológicas: sendo o DNA a linguagem de programação, as partes biológicas são uma sequência de dados (AGCTA…) que possuem funções determinadas. Por exemplo, uma sequência de DNA que faz a célula mudar a cor de verde para amarelo. Estas partes são descritas, catalogadas e respeitam determinado padrão físico de montagem (leia mais sobre os biobricks). Espera-se que com o tempo se possam descrever as características de inúmeras partes biológicas para serem utilizadas para a construção de dispositivos, sistemas,…. Essa é uma das funções da Secretária de Partes Biológicas Padrão do MIT.
(ii) dispositivos sintéticos: são compostos por partes biológicas capazes de processar sinais. Processam inputs em outputs. Para a construção de dispositivos robustos e eficientes são necessárias partes que funcionem de uma maneira previsível. Veja mais sobre dispositivos sintéticos.
(iii) sistemas sintéticos: são um conjunto dispositivos capazes de captar sinais, processar informações e realizar funções determinadas, como por exemplo, uma célula capaz de captar algum sinal do ambiente e decidir se irá realizar uma determinada função como combater uma célula tumoral, produzir determinado metabólito etc.
(iv) por útimo, existe a arquitetura sintética de populações em que, por ex, cada microrganismo possui um dispositivo diferente, sendo necessário que estes dispositivos trabalhem em conjunto para realizar determinada função. Trabalhar em conjunto, como uma população que precisa trabalhar com sincronismo, é o caso dos osciladores. Para isso, é necessário dominar mecanismos robustos de comunicação célula-célula.
A biologia sintética pode ser aplicada em praticamente todas áreas da biologia molecular, biotecnologia e engenharia genética. Porém existem algumas áreas que se destacam como sendo próprias da biologia sintética:
1. Construção de uma célula mínima: identificação das partes básicas para construção de uma célula.
2. Reconstrução de células: tendo como objetivo central a construção de formas de vida artificiais a partir de elementos químicos.
3. Construção de novos códigos genéticos.
5. Construção de células capazes de realizar funções diferentes daquelas encontradas na natureza, como a obtenção de novas rotas bioquímicas de produção de novos compostos. .
Essa última, talvez seja a que mais tenha impacto nas nossas vidas cotidianas a curto-prazo, através do desenvolvimento de remédios mais baratos e com a produção de combustíveis e químicos utilizando recursos renováveis.
Dispositivos sintéticos: uma bactéria que conta!
Para quem não leu, antes de ler este post, vale a pena ler o post anterior do interruptor para entender alguns conceitos básicos. Um contador é um componente central para circuitos digitais e de computação que guarda (e às vezes mostra) o número de eventos ou processos que ocorreram.
Neste trabalho é descrita a construção de contadores sintéticos para bactérias. Após a construção de um dispositivo aparentemente simples, Friedland et al. (2009) sofisticam bastante a construção do regulador para garantir uma resposta (contagem) robusta.
O primeiro contador descrito pelo trabalho é o “contador riborregulado por uma cascada transcricional” (riboregulated transcriptional cascade counter). Ele conta pulsos de arabinose no meio de cultura utilizando como sinal uma proteína luminescente (GFP). São riborregulados porque o taRNA (controlado por um promotor sensível a arabinose) se liga a cr e impede a formação de um grampo (pareamento) entre RBS e cr. Dessa maneira, permite o reconhecimento do RBS (sítio de ligação do ribossomo) pelo ribossomo e a tradução do gene (ver Figura abaixo). O interessante é que a transcrição do taRNA é regulada por um promotor sensível a arabinose. Assim, no primeiro pulso de arabinose, ocorre a transcrição de taRNA que permite a tradução da RNA polimerase T7. Após o primeiro pulso a arabinose e taRNA são degradados. No segundo pulso ocorre, da mesma maneira, a transcrição da RNA polimerase T3 através do promotor PT7 que é especificamente reconhecido pela RNA polimerase T7. No terceiro pulso, ocorre também através da mediação de taRNA e da polimerase T3, a transcrição de GFP. Dessa maneira, a bactéria responde aos 3 pulsos de arabinose com um sinal luminescente.
 Porém foi verificado um vazamento de expressão de GFP nos pulsos intermediários, além disso, demonstrou-se que se o pulso de arabinose não é dado a uma intensidade e frequência corretas, o contador não funciona direito. Isto provavelmente ocorre devido aos limites cinéticos intrínsecos envolvendo os tempos de transcrição e degradação de mRNA.
Porém foi verificado um vazamento de expressão de GFP nos pulsos intermediários, além disso, demonstrou-se que se o pulso de arabinose não é dado a uma intensidade e frequência corretas, o contador não funciona direito. Isto provavelmente ocorre devido aos limites cinéticos intrínsecos envolvendo os tempos de transcrição e degradação de mRNA.
Por esse motivo, um segundo (e muito mais bacana) contador foi construído, chamado contador de cascada de DNA invertase (DNA invertase cascade counter). Que basicamente acoplou ao modelo anterior um controle de invertases na região promotora.
Uma invertase é uma enzima muito interessante, as enzimas Cre, por ex., reconhecem determinadas sequências de DNA que flanqueiam uma determinada região de DNA e invertem a orientação dessa região. Essa enzima é utilizada para ativar os promotores através da inversão de sua orientação (ver Figura abaixo). Com essa construção foi possível obter um controle muito maior da expressão de GFP, ocorrendo, após o terceiro pulso, uma expressão muito maior de GFP. Este contador agora é muito mais robusto em relação à variação de tempo entre os pulsos de arabinose, podendo contar pulsos realizados em intervalos de tempo de 2 a 12 horas. Este novo dispositivo, além de mostrar o sinal luminescente, grava a informação no DNA através das recombinações.
Os autores foram ainda mais longe, trocaram o promotor de sensibilidade a arabinose por promotores capazes de responder a diferentes sinais. Dessa maneira, o dispositivo pode ser programado para gravar e responder diferentes sinais na ordem desejada. Incrível!
Esse dispositivo genético pode ser programado para diferentes funções a fim de sincronizar diferentes sinais para uma determinada resposta. Dependendo dos sinais acoplados ao dispositivo, estes mecanimos podem ser utilizados para biosensores, biorremediação ou na medicina.
Friedland, A., Lu, T., Wang, X., Shi, D., Church, G., & Collins, J. (2009). Synthetic Gene Networks That Count Science, 324 (5931), 1199-1202 DOI: 10.1126/science.1172005
Revistas científicas de Biologia Sintética
Há alguns posts atrás eu reuni alguns laboratórios de biologia sintética espalhados pelo mundo. É muito bacana para saber quem é quem nesse mundo científico. Porém, para saber o que estas pessoas e outras pessoas andam pensando é preciso ler o que eles publicam. Por isso, desta vez eu reuni as principais revistas de Biologia Sintética.
Fica aí a dica: dar uma olhada nessas revistas todo mês para ver o que está acontecendo no mundo synbio!
Nature – Molecular Systems Biology
Springer – Systems and Synthetic Biology
Journal of Biological Engineering
Journal of the Royal Society – Focus on Systems Biology





