Bactérias na guerra contra o câncer
 Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Vimos num post anterior, que bactérias idênticas em seu DNA podem tomar diferentes decisões quando estão sobre stress tais como escassez de alimento. Diversas estratégias tais como ficar dormentes (esporulação), entrar em competência ou até mesmo canibalismo são utilizadas para aumentar as chances de sobrevivência da colônia. Nos últimos anos, tem aumentado o número de evidências de que células cancerígenas agem de maneira bastante semelhante. Utilizando um avançado sistema de cooperação e comunicação celular, estas células são capazes de se espalhar pelo corpo colonizando novos órgãos (metástase) ou resistir a ações clínicas tais como quimioterapia. Uma das maneiras de resistir a quimioterapia, por exemplo, é pela estratégia de tornar-se “dormente” adotada por algumas das células do tumor, processo análogo a esporulação das bactérias.
Atualmente, desvendar o sistema de comunicação utilizado por células cancerígenas tem sido foco de inúmeras pesquisas. Como em qualquer guerra moderna, destruir o sistema de comunicação inimigo pode causar grandes danos. E na guerra contra o câncer provavelmente não será diferente. Impedir as células de se comunicarem pode evitar que elas adotem estratégias inteligentes tais como ficarem dormentes durante quimioterapia ou até mesmo matar células irmãs para obtenção de alimento. Uma vez entendido a linguagem utilizada por estas células, podemos utilizar isto ao nosso favor, interferindo nas mensagens e fazendo com que células dormentes sejam acordadas durante a quimioterapia ou até mesmo induzir as células cancerígenas a matarem umas as outras.
Finalmente, uma possível estratégia futura seria recrutar bactérias para derrotar o câncer. Elas poderiam ser utilizadas para “ensinar” as células do sistema imunológico a reconhecer e matar as células cancerígenas. É importante ressaltar que ainda compreendemos muito pouco sobre os mecanismos envolvidos no câncer e novas abordagens são necessárias para superá-lo. Entretanto, quem sabe num futuro próximo, estaremos em uma era de guerra cibernética biológica onde bactérias serão inteligentemente projetadas para derrotar o câncer.
Referências:
“Bacterial survival strategies suggest rethinking cancer cooperativity” Eshel Ben-Jacob, Donald S. Coffey, Herbert Levine. Trends in Microbiology. 2012.
“Bacterial linguistic communication and social intelligence” Eshel Ben-Jacob, Israela Becker, Yoash Shapira, Herbert Levine. Trends in Microbiology. 2004
Transposons: pedaços de DNA que mudam de endereço no genoma.
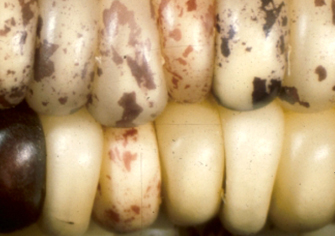
Mudança no padrão das cores do milho devido a interação entre os genes responsáveis pelo pigmento e elementos transponíveis.
Você sabia que alguns trechos de DNA têm a capacidade de mover-se no genoma, saindo de um cromossomo e se inserindo em outro? Pois é, e esta descoberta foi feita há muito tempo atrás por uma brilhante cientista, antes mesmo da descoberta da estrutura do DNA por Watson e Crick. Barbara McClintock ao observar a relação entre os padrões das cores do milho e algumas quebras cromossomais, percebeu que algumas destas quebras ocorriam com uma freqüência muito mais alta do que o esperado e, surpreendente, sempre no mesmo cromossomo. Após uma série de experimentos, a cientista percebeu que esta quebra ocorria devido a inserção de pedaços de DNA vindos de outros cromossomos que alteravam a expressão dos genes de pigmento. Confiante nos seus experimentos, Barbara então propôs que havia elementos transponíveis no genoma, ou seja, os genes não tinham um endereço fixo, mas tinham um mecanismo para movimentar-se dentro do genoma.
Se nos dias de hoje esta ideia parece bastante surpreendente, imagine como foi a recepção desta ousada ideia no começo da década de 50, quando a cientista começou a publicar seus resultados. Eles foram ignorados e ridicularizados pelos seus contemporâneos, levando a cientista a parar de publicar suas descobertas sobre o tema. Somente no início da década de 70 alguns elementos transponíveis foram identificados em bactéria validando assim a teoria de Barbara. Entretanto, o reconhecimento completo de uma das mais importantes descobertas da biologia somente aconteceu no início da década de 80, quando a cientista foi homenageada com o prêmio Nobel.
Atualmente vários estudos já relacionaram os elementos transponíveis, chamados de transposons, a uma série de doenças e como grande fonte de variabilidade genética. Hoje sabe-se que boa parte do genoma é composto destes elementos e dois mecanismos principais já foram identificados. O primeiro deles é um mecanismo de recortar-e-colar, onde alguns pedaços do DNA simplesmente saem de um lugar e movem-se para outra parte do genoma. O outro mecanismo é do tipo cortar-e-colar, gerando mais de uma cópia no genoma. Este último mecanismo é chamado de retrotransposons e consiste em quase metade do genoma humano e será tema de um próximo post.
FAQ #8 Como ver se a transformação gênica deu certo?
Já colocamos nossos plasmídeos nos bichinhos, mas ainda não acabou. Essa é a hora de saber se tudo que fizemos até aqui deu certo!
Primeiro, colocamos eles na placa de seleção com antibiótico e só aqueles que realmente incorporaram o plasmídeo vão sobreviver porque ganharam um gene amigo de resistência ao antibiótico. Agora pegamos uma parte das células sobreviventes e fazemos um teste usando a PCR (você lembra dessa técnica, né?).
Como testar com a PCR?! Roubamos os plasmídeos dessas células e tentamos multiplicar o pedaço de DNA que foi grudado nele adicionando primers, nucleotídeos e enzimas sob aquecimento e desaquecimento. Se o nosso plasmídeo modificado tiver sido incorporado pelos micro-organismos haverá a multiplicação do pedacinho e podemos vê-los em uma eletroforese. Caso contrário, nada foi copiado e nada vai aparecer na eletroforese. Então temos o sinal de que alguma coisa não deu certo por aqui e as chances de erros deste teste são bem pequenas.
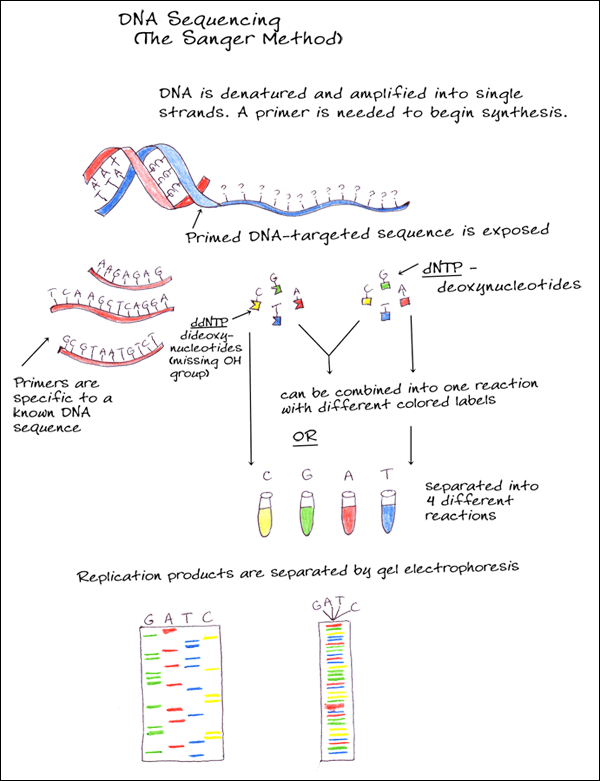
Depois vamos para um teste melhor ainda, o sequenciamento. Este teste diz exatamente qual é a sequência de pares de base da molécula do plasmídeo e acabam de vez suas dúvidas se a transformação deu certo ou não! O sequenciamento começa parecendo um processo de duplicação normal de DNA. Mas além de cadeias molde, enzimas e nucleotídeos normais, há nucleotídeos especiais sintetizados (ddNTP’s) que possuem duas boas propriedades: emitem luz e interrompem o prologamento da cadeia a partir de onde foram adicionados. Então, imagine uma molécula de DNA que possui um par de base A-T em um determinado comprimento e considere que a base A pertence à cadeia molde e a base T à cadeia complementar. Se esta base T for um nucleotídeo especial temos como identificar “quem é” e “qual sua posição” na cadeia, pois ele emitirá uma cor específica para Timina e o comprimento de sua cadeia está interrompido na posição exata (nº 2 em verde). 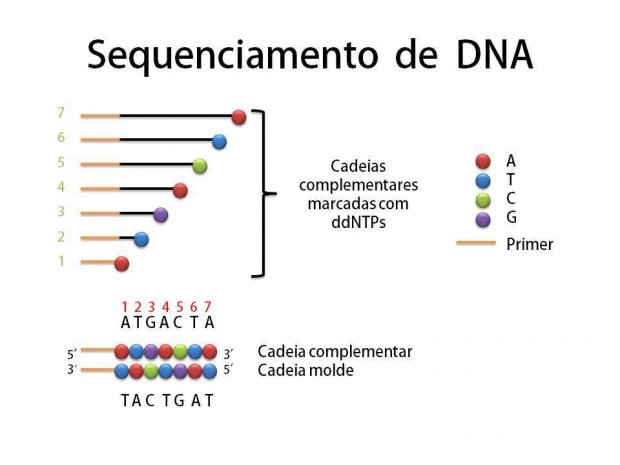 Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Podemos detectar a cor emitida através de um espectrógrafo e a posição pelo tamanho revelado na eletroforese em gel (o sequenciador é basicamente a união dos dois). Ou detectar o tipo de nucleotídeo presente não pela cor, mas colocando os filamentos “lavados” por cada tipo de nucleotídeo em poços diferentes do gel e interpretando a sequência a olho nu/manualmente. Depois de identificado o nucleotídeo da cadeia complementar é possível saber quem ocupa a mesma posição na cadeia molde, no caso citado é a base Adenina. Expanda esse raciocínio para todas as outras bases da molécula, com uma amostra grande teremos cadeias de todos os comprimentos possíveis, interrompidas por um dos 4 tipos de nucleotídeos especiais (A, T, C, G), e assim é possível sequenciar toda a molécula de DNA!
Para saber mais sobre sequenciamento genético, clique aqui.
Por Otto Heringer e Viviane Siratuti.
FAQ #7 Como enfiar o plasmídeo nas células?
Dar e receber plasmídeos não é nenhuma novidade para algumas células!
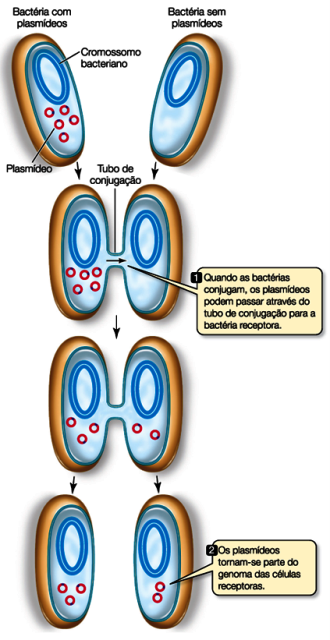
As bactérias costumam trocar informações genéticas assim e esse processo é chamado de conjugação, portanto elas já possuem toda maquinaria necessária pra isso! Desse jeito elas aumentam muito a variabilidade e as chances de sobrevivência da população.
Podemos usar esse mecanismo natural para enfiar os plasmídeos, mas na maioria das vezes damos uma ajudinha usando choques térmicos ou elétricos.
Para entender o choque térmico, assista o vídeo do JOVE aqui. E do choque elétrico, aqui.
Por Otto Heringer e Viviane Siratuti.
FAQ #6 Como ver se nossos plasmídeos incorporaram os pedacinhos de DNA?
Sabendo o tamanho do plasmídeo e dos pedacinhos estimamos um tamanho para nosso novo plasmídeo e agora é só conferir se estão como esperado!
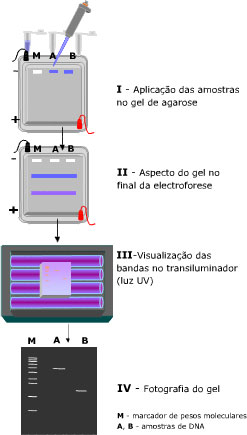
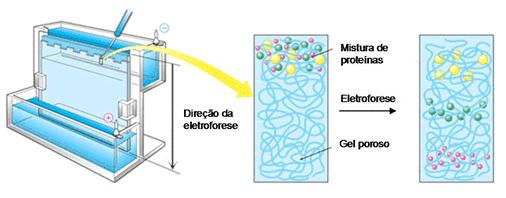
Para isso, existe uma técnica de separação chamada eletroforese em gel, onde as moléculas são “peneiradas” por algum polímero (gel de poliacrilamida ou de agarose) quando se movimentam atraídas ou repulsadas pelos eletrodos de cargas opostas que são colocados nas extremidades do gel. Ou seja, uma molécula com carga negativa caminha para o eletrodo de carga positiva e vice-versa. E as moléculas de mesma carga correm de maneiras diferentes pelo gel de acordo com seus pesos moleculares e tamanhos, as menores e mais “leves” passam com mais facilidade pelo gel enquanto as maiores e mais “pesadas” ficam mais retidas. No final, temos as moléculas separadas ao longo do gel e podemos comparar com a ladder (uma “régua” feita de moléculas com pesos e tamanhos já conhecidos) para saber se nossos plasmídeos estão no tamanho esperado.
Depois de confirmar pela eletroforese que nossos plasmídeos foram modificados, precisamos “salvá-los” do gel para colocar nas células. Basicamente, cortamos o pedaço de gel que está com nossos plasmídeos e colocamos em tubinhos com uma pequena coluna de sílica dentro (imagem aí embaixo). Então as moléculas de DNA se ligam à coluna, fazemos uma lavagem para tirar o gel e depois diluímos o DNA para retirá-lo da coluna!
Para saber mais sobre eletroforese, assista o vídeo do JOVE Science aqui e purificação aqui. 🙂
Por Otto Heringer e Viviane Siratuti.
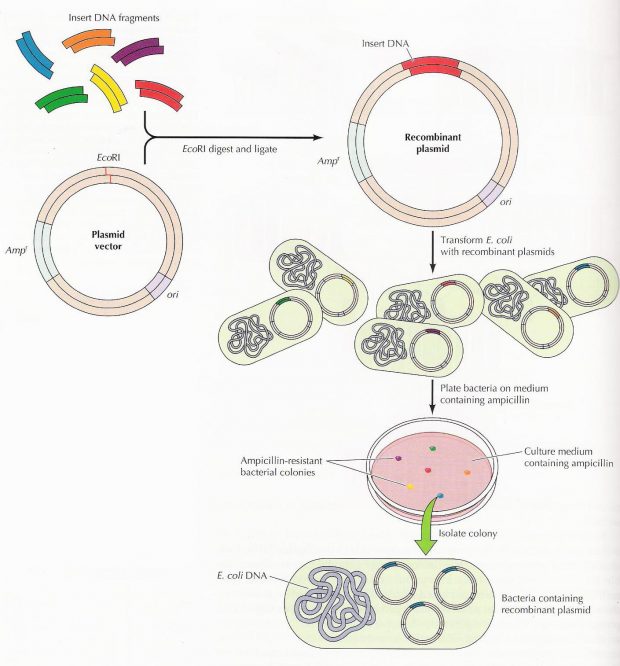
FAQ #4 Onde colocar esses pedaços de DNA?
“No genoma, né, dêr!”. Certo, mas como? E será que o genoma é o único lugar que podemos colocar esse novo pedacinho de DNA no micro-organismo que queremos modificar? Não! Existe outro lugar também e ele se chama plasmídeo (quem já jogou BioShock vai soltar umas sinapses a mais agora).
O plasmídeo é um DNA circular presente em várias espécies de seres vivos e é responsável por conter informações valiosas envolvendo a sobrevivência do organismo a fatores externos (como por exemplo sua resistência a um antibiótico), além de ser o principal ator da transferência horizontal de informação genética, ou seja, a passagem de um DNA funcional de um ser vivo para outro sem haver hereditariedade (é aí que o jogo BioShock extrapola isso para seres humanos). Adivinha de onde veio a ideia de usar o plasmídeo como transmissor – “vetor” – de informação genética para modificar as células? Veio exatamente desse mecanismo natural de realizar transferência horizontal de genes que vários micro-organismos possuem, então aproveitamos para fazer a transferência das informações genéticas que nós queremos!
E se vamos usar plasmídeos é preciso extraí-los também! Os plasmídeos são moléculas de DNA assim como os pedacinhos obtidos a partir de um genoma, e multiplicados pela PCR, que vamos introduzir no microorganismo, mas aqui há uma etapa importante durante a extração de DNA onde é feita a separação do conteúdo plasmidial do genômico.
O vídeozinho abaixo tem uma animação no ínicio e depois mostra o procedimento do isolamento do plasmídeo em lab!
[youtube_sc url=”http://www.youtube.com/watch?v=8xEDEJ0DHFA”]
Por Otto Heringer e Viviane Siratuti.
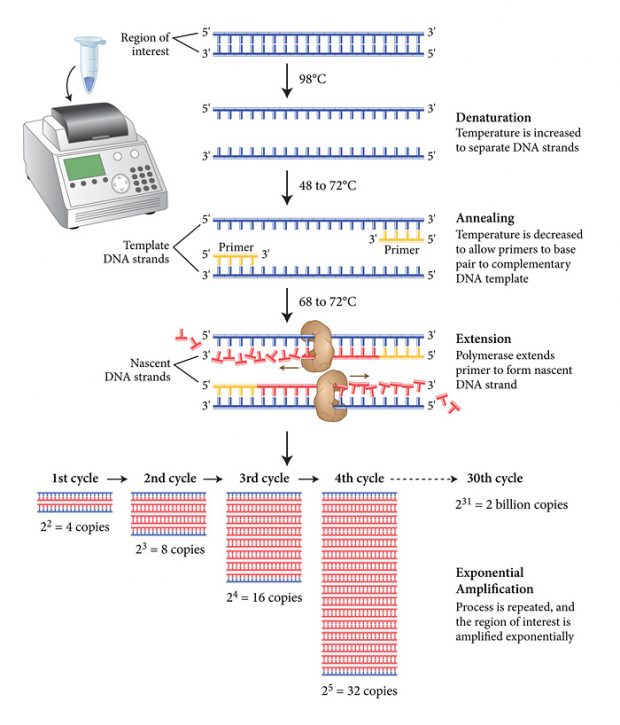
FAQ #3 Como copiar o código em laboratório?
Ahá! Essa é a pergunta que mudou a biotecnologia. Até conseguirmos fazer isso, nós (i.e. humanidade) passamos por um caminho bem interessante envolvendo prêmios nobel e momentos epifânicos. O videozinho explica muito melhor do que esse texto corrido como funciona a metodologia pra se fazer isso, a Reação em Cadeia da Polimerase (Polimerase Chain Reaction), o tão amado e odiado (quando simplesmente não funciona) PCR!
[youtube_sc url=”http://www.youtube.com/watch?v=vmlLj1aLZ7s”]
Encurtando a história: usando uma enzima que trabalha “sentando” no molde de uma fita única de DNA, aquecimento e desaquecimento (para “ligar” e “desligar” essa enzima) e pedacinhos de nucleotídeo, conseguimos fazer milhões de cópias de apenas uma única molécula de DNA – é por isso que o pessoal do CSI (o seriado) consegue uma grande informação genética usando apenas resíduos quase desprezíveis de material biológico.
Por Otto Heringer e Viviane Siratuti.
FAQ #2 Como extrair um pedaço de DNA desejado?
Queremos pegar um pedacinho codificante de DNA (responsável por alguma característica no organismo) de um outro pedaço maior de DNA. OK, mas onde fica esse outro pedaço? Bem, existem pedaços disso praticamente a todo o seu redor. Onde há vida, há informação genética que pode ser “pega”. Extraímos ele basicamente fazemos lavagens usando solventes de diferentes “afinidades de dissolução” (a grosso modo) com as biomoléculas da célula até restar o DNA.
Se você quer saber, existem até kits comerciais para se fazer isso (como por exemplo a imagem abaixo), são os famigerados “kits de miniprep”. “Mini” porque em geral são bastante usados kits que trabalham com pequenos volumes (microlitros), mas também existem os kits de “midiprep” e “maxiprep”, para volumes maiores.
Assista o vídeo caso você queira saber mais do processo (aviso: a realidade às vezes não é algo tão excitante como você imaginava).
[youtube_sc url=”http://www.youtube.com/watch?v=P12DPa-g8Ro”]
Aliás, se você ficou com vontade de extrair DNA em casa, é bem simples, clique aqui.
Por Otto Heringer e Viviane Siratuti.
FAQs da Bioengenharia – Introdução
Preocupados com os novos amantes da biologia sintética, estamos procurando materiais de introdução no assunto e encontramos algumas coisas interessantes! 🙂
Por isso vamos colocar uma sequência de posts explicativos, partindo de uma visão geral e seguindo por perguntas mais específicas. É um pequeno tutorial-FAQ sobre alguns fundamentos que quando entendidos ajudam bastante a entender algumas de nossas discussões e outros posts deste blog. Serve para ajudar principalmente quem não é da área de biológicas ou quem ainda não se aprofundou muito neste assunto! Se continuarem com dúvidas, perguntem tá?!
Lembrem-se de ativar as legendas dos vídeos, caso necessitem! Todos possuem legendas originais em inglês e alguns em português. Você pode colocá-las traduzidas quando só houver em inglês (não muito recomendado, rs). Nos vídeos do JOVE existe a opção de colocar os textos em português. Se você não sabia que o YouTube fazia isso por você, dá uma olhada aqui (dica: barrinha inferior do vídeo).
Antes de mais nada, você sabe o que são e quais as relações entre nossas principais moléculas orgânicas (DNA, RNA e proteínas), certo? Se sua resposta é não, assista um desses vídeos!
[youtube_sc url=”http://www.youtube.com/watch?v=h3b9ArupXZg#t=0″]
[youtube_sc url=”http://www.youtube.com/watch?v=tMr9XH64rtM”]
Indo além da Estrutura
OK, agora vamos além desses conceitos básicos dos vídeos. Mais que entender a estrutura e a dinâmica dessas biomóleculas, como os cientistas a alteram? Em linhas gerais, como se faz para modificar geneticamente um organismo?
Vamos lá! Primeiro pegamos um pedacinho codificante de DNA (gene) de outro organismo que tenha certa característica que desejamos. Para isso, extraímos o DNA e depois multiplicamos só esse pedaço que nos interessa através de uma técnica chamada PCR (afinal, a eficiência das transformações é pequena e quanto mais material melhor). Precisamos também extrair e abrir os plasmídeos (DNA circular) do organismo escolhido para receber este gene e assim receber as novas características (já que o plasmídeo é nosso principal veículo de introdução dos genes nas células). Para abrir estes plasmídeos usamos enzimas de restrição que cortam as moléculas de DNA em lugares específicos e chamamos isso de digestão. Depois que tivermos vários plasmídeos e genes, podemos grudá-los com a ajuda de enzimas de ligação. E então, para saber se nosso novo plasmídeo deu certo usamos uma técnica de separação por carga e tamanho de molécula, a eletroforese em gel. Nela, as moléculas se acumulam em certas posições que nos dizem seus tamanhos (mergulhadas em um polímero) e assim podemos identificar aquelas que correspondem ao que estimamos. Além disso, fazemos testes com a técnica PCR e o sequenciamento genético. Se os testes disserem que tudo que fizemos deu certo, recuperamos o material e colocamos nossos novos plasmídeos nos organismos, geralmente utilizando choque térmico ou elétrico. Esse passo, introdução de plasmídeos, é a chamada transformação. Mas para selecionarmos somente aqueles organismos que realmente foram transformados usamos antibióticos que matam as células que não ganharam os nossos novos plasmídeos, já que não possuem os genes de resistência que foram colocados neles. E no final, cada célula que sobreviveu se multiplicará, formando colônias de organismos geneticamente modificados. Agora dá uma olhada no esquema de novo e vê se entendeu até aqui!
Por Otto Heringer e Viviane Siratuti.
iGEM World Jamboree – Parte 1: Overview do Evento
*este post sofreu com o fato do autor ter perdido o cartão de memória com fotos do evento. Algumas foram recuperadas, embora com qualidade muito pior u.u…. faremos a melhor limonada possível dos limões recebidos e esperamos que gostem!
Entre os dias 1 e 4 de novembro de 2013 ocorreu a etapa mundial do iGEM, o iGEM World Jamboree, onde equipes selecionadas nas fases regionais do mundo todo reúnem-se no MIT (Massachusetts Institute of Technology), em Boston, para apresentarem seus projetos finalizados nesta temporada. Apesar de nosso projeto não ter sido selecionado, mandamos nossos enviados especiais, Pedro e Danilo, para acompanhar o evento e trazer essa experiência!
O evento apresenta o mesmo formato que as fases regionais, com a diferença de ser muito maior: são três salas de apresentações simultâneas, nas categorias “undergraduate” e “overgraduate”, e uma sala com a categoria Entrepreneurship e Software. Ao todo são em média 80 apresentações diferentes durante o evento todo, das quais acompanhamos cerca de 30.
Este é o primeiro de uma série de posts originados deste evento, nos quais contaremos as impressões gerais da experiência. Nos próximos posts iremos abordar os nossos projetos favoritos, as possibilidades brasileiras e latino americanas no evento e como o iGEM se tornou uma máquina azeitada de inovação (mesmo contando com apenas 7 funcionários).
A cidade, o MIT e tudo mais
O evento não ocorre propriamente em Boston, mas sim em Cambridge, cidade onde estão localizados o MIT e Harvard. Cambridge é uma cidade bonitinha com a estética da Nova Inglaterra: prédios de tijolinhos, esquilos, carvalhos e com aquela beleza clássica de outono do clima temperado totalmente alheia a nós, mas que nos acostumamos a ver nos desenhos da Disney. Fora isso, é a moradia de milhares de estudantes de alto nível do mundo todo que desenvolvem suas atividades nas universidades locais.
Os campi de Harvard e MIT são bem integrados à cidade. Harvard, apesar de ser cercada de muros, possui os portões abertos, amplos gramados e um visual clássico, o movimento de estudantes é bastante intenso o dia inteiro. O MIT, por sua vez, não possui muros e conta com edificações clássicas e também modernas. Percebe-se que a relação com o espaço é bem diferente do que a que temos no campi da USP, que, apesar de ser de uma universidade pública, é bem menos integrado à cidade do que as universidades citadas acima.
O MIT, em específico, não deve em nada à USP quanto à sinalização dos prédios e ruas: ambos são bastante confusos para um visitante. Fora essa dificuldade, a estrutura é realmente boa: auditórios espaçosos, salas adequadas para conversas, espaços para estudo independente e boa rede wifi formam um ambiente propício para o desenvolvimento de idéias. Com certeza é um lugar onde qualquer desenvolvedor de projetos gostaria de estar.
O evento
O primeiro dia do evento é apenas para inscrições e um breve treino. Apesar do clima de excitação em meio à pizza (UHUL!), o ambiente não é de muita confraternização. Os times revezam-se nas salas, onde têm apenas meia hora para treinar suas apresentações (acredite, em meia hora com certeza alguém estará batendo na sua porta para que você saia), e podem aproveitar o tempo para escrever o que quiser nas lousas do hall.
No segundo e terceiro dias temos o que realmente interessa. As apresentações começam pontualmente e são intercaladas com perguntas dos juízes e da platéia. Os times possuem 20 minutos para a apresentação e mais 10 para perguntas e não ocorreram problemas nesse sentido: tudo parecia muito bem alinhado e ensaiado (com slides previamente preparados para responder as perguntas que pudessem surgir, inclusive), com algumas excessões específicas. Devo dizer, porém, que nem sempre os juízes faziam perguntas claras e objetivas, ainda mais levando-se em conta que para muitos times o inglês não era a língua nativa.
Ao contrário do evento regional da América Latina, os pôsteres são exibidos em todos os momentos de intervalo e, no nosso caso em específico, alguns foram importantes para decidir o que veríamos. Os pôsteres do iGEM são ligeiramente diferentes do que estamos acostumados, com mais texto e maior preocupação com o design de como as informações são apresentadas. Nesse sentido, ainda não me convenci que o houvesse algum pôster melhor que o nosso, haha.
Ao fim, dos seis finalistas das duas categorias, Overgraduate e Undergraduate, uma surpresa: Nenhum time americano na final. 5 times europeus, sendo 3 alemães, 1 francês e um inglês, mais um time chinês. (confira!). Os resultados finais, bem como awards estão nesta página. Com destaque para, até onde eu sei, o primeiro award ganho por um time Latino americano: Melhor modelagem para o time de Buenos Aires!
Enfim, nos próximos posts teremos uma seleção dos melhores projetos segundo nós mesmos, bem como relatos da categoria de empreendedorismo e afins!
Até lá!
*Hard Level Bonus Stage: Ache-nos na foto oficial!

Dica: Dois solitários vermelhos.